| Faktor A | Kovariate $c$ | Messwert $y$ |
|---|---|---|
| A.1 | 0.8 | 7 |
| A.1 | 1.6 | 8 |
| A.1 | 2.4 | 9 |
| A.2 | 3.2 | 2 |
| A.2 | 4.0 | 3 |
| A.2 | 4.8 | 4 |
| A.3 | 5.6 | 11 |
| A.3 | 6.4 | 12 |
| A.3 | 7.2 | 13 |
33 Die ANCOVA
Letzte Änderung am 04. March 2025 um 17:47:26
“It’s better to solve the right problem approximately than to solve the wrong problem exactly.” — John Tukey
Eigentlich hat sich die Analysis of Covariance (ANCOVA) etwas überlebt. Wir können mit dem statistischen Modellieren eigentlich alles was die ANCOVA kann plus wir erhalten auch noch Effektschätzer für die Kovariaten und die Faktoren. Dennoch hat die ANCOVA ihren Platz in der Auswertung von Daten. Wenn du ein oder zwei Faktoren hast plus eine numerische Variable, wie das Startgewicht, für die du die Analyse adjustieren möchtest, dann ist die ANCOVA für dich gemacht. Es gibt auch noch andere Möglichkeiten, die Idee ist eben, dass du für jeden deiner Beobachtungen noch eine kontinuierliche Variable zusätzlich zu deinen Faktoren erhebst, die eben nicht dein Messwert ist. Im Bereich der Agrarwissenschaften ist die ANCOVA eher selten zu finden.
33.1 Allgemeiner Hintergrund
Also kurz gesprochen adjustiert die Analysis of Covariance (ANCOVA) die Faktoren einer ANOVA um eine kontinuierliche Covariate. Eine Kovariate ist eine Variable, die mit berücksichtigt wird, um mögliche verzerrende Einflüsse auf die Analyseergebnisse (ungebräuchlich Konfundierung) abzuschätzen oder zu verringern. Adjustiert bedeutet in dem Fall, dass die Effekte des unterschiedlichen Startgewichts von Pflanzen durch das Einbringen der Kovariate mit in der statistischen Analyse berücksichtigt werden. Wir werden hier auch nur über die Nutzung in R sprechen und auf die theoretische Herleitung verzichten.
Wie immer gibt es auch passende Literatur um die ANCOVA herum. Karpen (2017) beschreibt in Misuses of Regression and ANCOVA in Educational Research nochmal Beispiele für die falsche oder widersprüchliche Nutzung der Regression und ANCOVA. Ich nutze die Quelle immer mal wieder in meinen vertiefenden Vorlesungen. Da wir hier ja nicht nur Agrawissenschaftler haben, hilft die Arbeit von Kim (2018) mit Statistical notes for clinical researchers: analysis of covariance (ANCOVA) auch nochmal weiter. Die Besonderheit bei der klinischen Forschung ist ja die “Beweglichkeit” der Beobachtungen. Menschen lassen sich eben nicht vollständig kontrollieren und daher ist die Thematik eine andere als bei Pflanzen und Tieren.
Das Modell
Wenn wir neben einem bis mehreren Faktoren \(f\) noch eine numerische Kovariate \(c\) mit modellieren wollen, dann nutzen wir die ANCOVA (eng. Analysis of Covariance). Hier kommt dann immer als erstes die Frage, was heißt den Kovariate \(c\)? Hier kannst du dir eine numerische Variable vorstellen, die ebenfalls im Experiment gemessen wird. Es kann das Startgewicht oder aber die kummulierte Wassergabe sein. Wir haben eben hier keinen Faktor als Kategorie vorliegen, sondern eben etwas numerisch gemessen. Daher ist unsere Modellierung etwas anders.
\[ y \sim f_A + f_B + ... + f_P + f_A \times f_B + c_1 + ... + c_p \]
mit
- \(y\) gleich dem Messwert oder Outcome
- \(f_A + f_B + ... + f_P\) gleich experimenteller Faktoren
- \(f_A \times f_B\) gleich einem beispielhaften Interaktionsterm erster Ordnung
- \(c_1 + ... + c_p\) gleich einer oder mehrer numerischer Kovariaten
Hier muss ich gleich die Einschränkung machen, dass wir normalerweise maximal ein zweifaktorielles Modell mit einem Faktor \(A\) und einem Faktor \(B\) sowie einer Kovariate \(c\) betrachten. Sehr selten haben wir mehr Faktoren oder gar Kovariaten in dem Modell. Wenn das der Fall sein sollte, dann könnte eine andere Modellierung wie eine multiple Regression eine bessere Lösung sein.
Hypothesen
Wenn wir von der ANCOVA sprechen, dann kommen wir natürlich nicht an den Hypothesen vorbei. Die ANCOVA ist ja auch ein klassischer statistischer Test. Hier müssen wir unterscheiden, ob wir eine Behandlung mit zwei Gruppen, also einem Faktor \(A\) mit \(2\) Leveln vorliegen haben. Oder aber eine Behandlung mit drei oder mehr Gruppen vorliegen haben, also einem Faktor \(A\) mit \(\geq 3\) Leveln in den Daten haben. Da wir schnell in einer ANOVA mehrere Faktoren haben, haben wir auch schnell viele Hypothesen zu beachten. Jeweils ein Hypothesenpaar pro Faktor muss dann betrachtet werden. Das ist immer ganz wichtig, die Hypothesenpaare sind unter den Faktoren mehr oder minder unabhängig. Bei der ANCOVA kommt dann noch die Kovariate mit hinzu, die wir dann ja auch noch testen müssen.
Bei einem Faktor \(A\) mit nur zwei Leveln \(A.1\) und \(A.2\) haben wir eine Nullhypothese, die du schon aus den Gruppenvergleichen wie dem t-Test kennst. Wir wollen zwei Mittelwerte vergleichen und in unserer Nullhypothese steht somit die Gleichheit. Da wir nur zwei Gruppen haben, sieht die Nullhypothese einfach aus.
\[ H_0: \; \bar{y}_{A.1} = \bar{y}_{A.2} \]
In der Alternativehypothese haben wir dann den Unterschied zwischen den beiden Mittelwerten. Wenn wir die Nullhypothese ablehnen können, dann wissen wir auch welche Mittelwertsunterschied signifikant ist. Wir haben ja auch nur einen Unterschied getestet.
\[ H_A: \; \bar{y}_{A.1} \neq \bar{y}_{A.2} \] Das Ganze wird dann etwas komplexer im Bezug auf die Alternativehypothese wenn wir mehr als zwei Gruppen haben. Hier kommt dann natürlich auch die Stärke der ANOVA zu tragen. Eben mehr als zwei Mittelwerte vergleichen zu können.
Die klassische Nullhypothese der ANOVA hat natürlich mehr als zwei Level. Hier einmal beispielhaft die Nullhypothese für den Vergleich von drei Gruppen des Faktors \(A\). Wir wollen als Nullhypothese testen, ob alle Mittelwerte der drei Gruppen gleich sind.
\[ H_0: \; \bar{y}_{A.1} = \bar{y}_{A.2} = \bar{y}_{A.3} \]
Wenn wir die Nullhypothese betrachten dann sehen wir auch gleich das Problem der Alternativehypothese. Wir haben eine Menge an paarweisen Vergleichen. Wenn wir jetzt die Nullhypothese ablehnen, dann wissen wir nicht welcher der drei paarweisen Mittelwertsvergleiche denn nun unterschiedlich ist. Praktisch können es auch alle drei oder eben zwei Vergleiche sein.
\[ \begin{aligned} H_A: &\; \bar{y}_{A.1} \ne \bar{y}_{A.2}\\ \phantom{H_A:} &\; \bar{y}_{A.1} \ne \bar{y}_{A.3}\\ \phantom{H_A:} &\; \bar{y}_{A.2} \ne \bar{y}_{A.3}\\ \phantom{H_A:} &\; \mbox{für mindestens einen Vergleich} \end{aligned} \]
Wenn wir die Nullhypothese abgelhent haben, dann müssen wir noch einen sogenannten Post-hoc Test anschließen um die paarweisen Unterschiede zu finden, die dann signifikant sind. Das ganze machen wir dann aber in einem eigenen Kapitel zum Post-hoc Test.
Bei der Kovariate testen wir, ob es eine Änderung über die Faktoren gibt. Hierbei ist natürlich keien Änderung ein paraleller Verlauf über die Faktorenlevel. Oder anders ausgedrückt, die Steigung \(\beta_c\) von der Kovariate \(c\) ist Null. Wir haben also als Nullhypothese keine Änderung in der Steigung. Damit haben wir auch eine Ähnlichkeit mit der Nullhypothese in der linearen Regression. Dort testen wir ja auch die Steigung.
\[ H_0: \beta_c = 0 \]
Die Alternativhypothese ist dann natürlich die Verneinung der Nullhypothese und damit eine Steigung oder Veränderung der Kovariate über die Faktoren. Wir können also eine Veränderung in den Mittelwerten der Faktoren beobachten, wenn sich die Kovariate ändert. Wir haben dann eben einen globalen Mittelwert mit einer Steigung in unserem Modell.
\[ H_A: \beta_c \neq 0 \]
Jetzt ist natürlich die Frage, wollen wir eine signifikante Kovariate oder nicht? Manchmal haben wir eine signifkante Kovariate im Modell und wollen unsere Faktoren für den Effekt der Kovariate adjustieren. Das machen wir dann indem wir die Kovariate mit ins Modell nehmen. In den Agrarwissenschaften ist es aber eher selten, dass wir eine signifikante Kovariate als erstrebenswert erachten.
Welche Pakete gibt es eigentlich?
Die Frage lässt sich relativ einfach beantworten. Es gibt neben der klassischen Implementierung in R mit der Funktion aov() nch die Möglichkeit die R Pakete {car} und die Funktion Anova() zu nutzen. Wenn es etwas komplexer sein soll und wir eventuell dann noch Messwiederholungen mit in den Daten haben, dann könnne wir auch das R Paket {afex} nutzen. Bei einer klassichen ANCOVA würde ich dann aber bei dem Standard in {base} oder {car} bleiben. Es gibt hier eigentlich keinen Grund noch komplexer zu werden.
33.2 Theoretischer Hintergrund
Betrachten wir jetzt einmal den theoretischen Hintergrund der ANCOVA. Ich habe dazu im Folgenden einmal einen sehr theoretischen Datensatz zusammengebaut. Es geht hier auch eher darum das Prinzip zu verstehen als die Formeln der ANCOVA zu zerlegen. Wir haben in dem Datensatz einen Faktor A mit drei Leveln vorliegen sowie eine Kovariate \(c\). Erhoben haben wir den Messwert \(y\) in unserem Experiment. Dabei ist jetzt wichtig, dass die Kovariate \(c\) in den Leveln des Faktors A aufsteigend sortiert ist. Darüber hinaus sind die Abstände zwischen den Messwerten immer gleich. Das habe ich sogebaut, dass wir damit keinen Restfehler mehr in den Daten vorliegen haben.
Jetzt können wir einmal die Mittelwerte der Faktorlevel sowie den Mitelwert der Kovariate für jedes Faktorlevel berechnen. Damit haben wir dann folgende Tabelle vorliegen. Die Idee ist hier, dass wir neben dem Effekt des Faktors noch einen Effekt der Kovariate vorliegen haben. In unserem Fall steigt die Kovariate stetig mit dem Messwert an. Daher kann ein Teil der Variation in den Messwerten \(y\) durch die Kovariate erklärt werden.
| Faktor A | Mittelwert Messwert | Mittelwert Kovariate |
|---|---|---|
| A.1 | 8 | 1.6 |
| A.2 | 3 | 4.0 |
| A.3 | 12 | 6.4 |
Berechnen wir also einmal das Modell mit lm() und lassen uns die Koeffizienten wiedergeben. Dann können wir eventuell das Modell und die ANCOVA dann besser verstehen. Nochmal, wir haben keinen Restfehler in den Daten, deshals sind die Werte so schon glatt, wenn ich runde.
R Code [zeigen / verbergen]
lm(rsp ~ fa + cov, data = f1_ancova_theo_tbl) |>
coef() |> round(2)(Intercept) faA.2 faA.3 cov
6.00 -8.00 -2.00 1.25 Was haben wir nun als Koeffizienten aus dem Modell vorliegen? Der y-Achsenabschnitt liegt bei \(6\) und fällt damit mit dem Mittelwert der ersten Levels \(A.1\) unter der Berücksichtigung des Effekts der Kovariate \(cov\) zusammen. Daher ist der Mittelwert des Levels \(A.1\) gleich \(6 + 1.6 \times 1.25\). Die \(1.6\) ist der Mittelwert der Kovariate für das Level \(A.1\) des Faktors. Faktisch kippst du den globalen Mittelwert \(\beta_0\) der ANOVA um die Steigung oder den Effekt der Kovariate \(c\). Du adjustierst also den globalen Mittelwert zu dem verglichen werden soll. In der folgenden Abbildung 33.1 siehst du nochmal den Zusammenhang der Daten zu den Koeffizienten aus dem Modell dargestellt. Die Koeffizienten des Level \(A.2\) und \(A.3\) sind jetzt nicht mehr die Änderungen zu einem fixen Wert sondern zu dem Wert auf der Geraden, die durch die Steigung der Kovariate \(c\) gegeben ist. Eigentlich eine ganz geschickte Idee.
Wie immer gibt es auch für die Frage nach dem Tutorium für die ANCOVA verschiedene Quellen. Ich kann noch folgende Informationen und Hilfen empfehlen.
- How to perform ANCOVA in R liefert nochmal mehr Code und weitere Ideen zu der Analyse in R.
- ANCOVA in R beschreibt auch nochmal etwas anders die ANCOVA und deren Anwendung in R
- Kovarianzanalyse ist eine deutsche Quelle, die nochmal vertiefend auf die Kovarianzanalyse eingeht, was eigentlich dann auch nichts anderes ist als eine ANCOVA zu rechen.
33.3 Genutzte R Pakete
Wir wollen folgende R Pakete in diesem Kapitel nutzen.
R Code [zeigen / verbergen]
pacman::p_load(tidyverse, magrittr, broom, quantreg, car, afex,
see, performance, emmeans, multcomp, janitor, scales,
parameters, conflicted)
conflicts_prefer(dplyr::select)
conflicts_prefer(dplyr::filter)
cbbPalette <- c("#000000", "#E69F00", "#56B4E9", "#009E73",
"#F0E442", "#0072B2", "#D55E00", "#CC79A7")An der Seite des Kapitels findest du den Link Quellcode anzeigen, über den du Zugang zum gesamten R-Code dieses Kapitels erhältst.
33.4 Daten
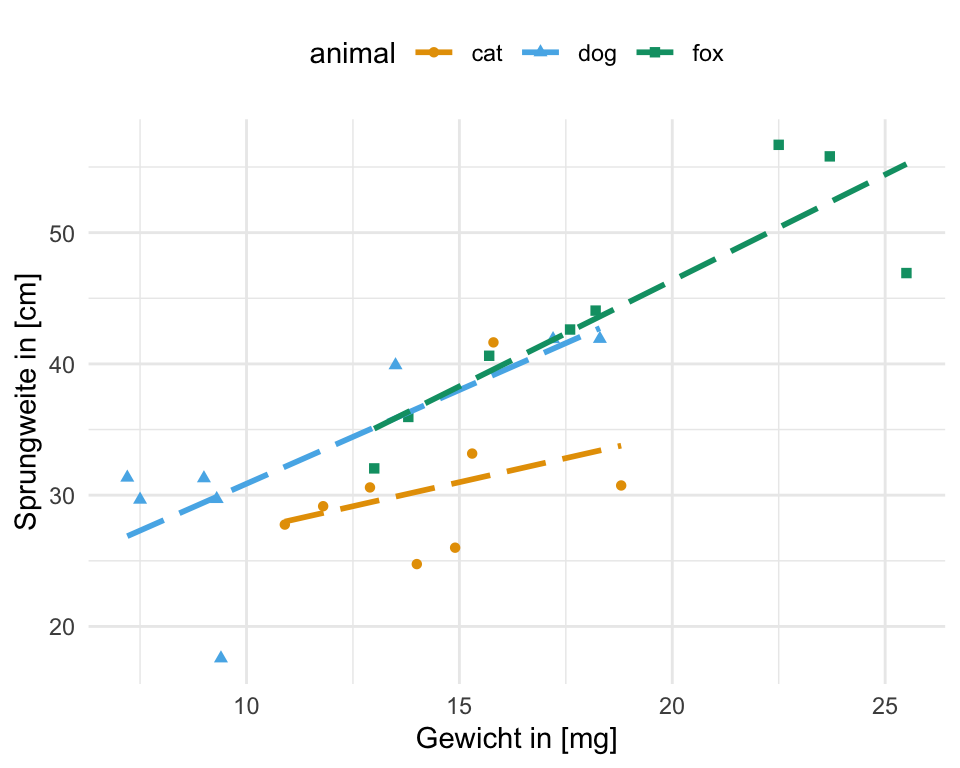
Dann fangen wir wie immer mit den Daten als Beispiel an. Ich habe hier dann einmal einen Datensatz mit einem Faktor A und einer Kovariate c zusammengebaut. Der Faktor ist in diesem Fall die Flohart mit animal und die Kovariate weight mit dem jeweiligen Gewicht des entsprechenden Flohs. Wir wollen nun wissen, ob sich die Sprungweite der Floharten auch in Teilen durch das Gewicht der Flöhe erklären lässt. Immerhin könnten ja schwerere Flöhe eventuell nicht so weit springen oder aber gerade weiter springen, da diese Flöhe mehr Muskeln haben. Was hier dann der Grund ist, werden wir zwar nicht im Detail erfahren, wohl aber, ob das Gewicht einen Einfluß hat.
R Code [zeigen / verbergen]
fac1_cov_tbl <- read_excel("data/fleas_complex_data.xlsx", sheet = "covariate-fac1") |>
select(animal, weight, jump_length) |>
mutate(animal = as_factor(animal),
weight = round(weight, 2),
jump_length = round(jump_length, 2)) |>
rownames_to_column(".id")In der folgenden Tabelle siehst du einmal die Sprungweiten in [cm] der einzelnen Flöhe sowie das Gewicht in [mg]. Jeder Floh statt damit von einem Tier ab und hat ein Gewicht zugeordnet. Dabei ist das Gewicht kontinuierlich gemessen und die Flohart ist dann der kategorielle Faktor.
| animal | weight | jump_length |
|---|---|---|
| cat | 14 | 24.75 |
| cat | 12.9 | 30.59 |
| cat | 15.8 | 41.64 |
| ... | ... | ... |
| fox | 23.7 | 55.81 |
| fox | 13 | 32.04 |
| fox | 25.5 | 46.92 |
Dann wollen wir uns auch einmal die Daten als eine Abbildung anschauen. Wir sehen, dass wir einen Effekt des Gewichts auf die Sprungweite haben. Flöhe die ein höheres Gewicht haben, springen im generellen weiter als leichtere Flöhe. Diesen Zusammenhang sehen wir dann auch in jeder der Floharten. Zwar nicht überall gleich stark ausgeprägt, aber dennoch sichtbar.
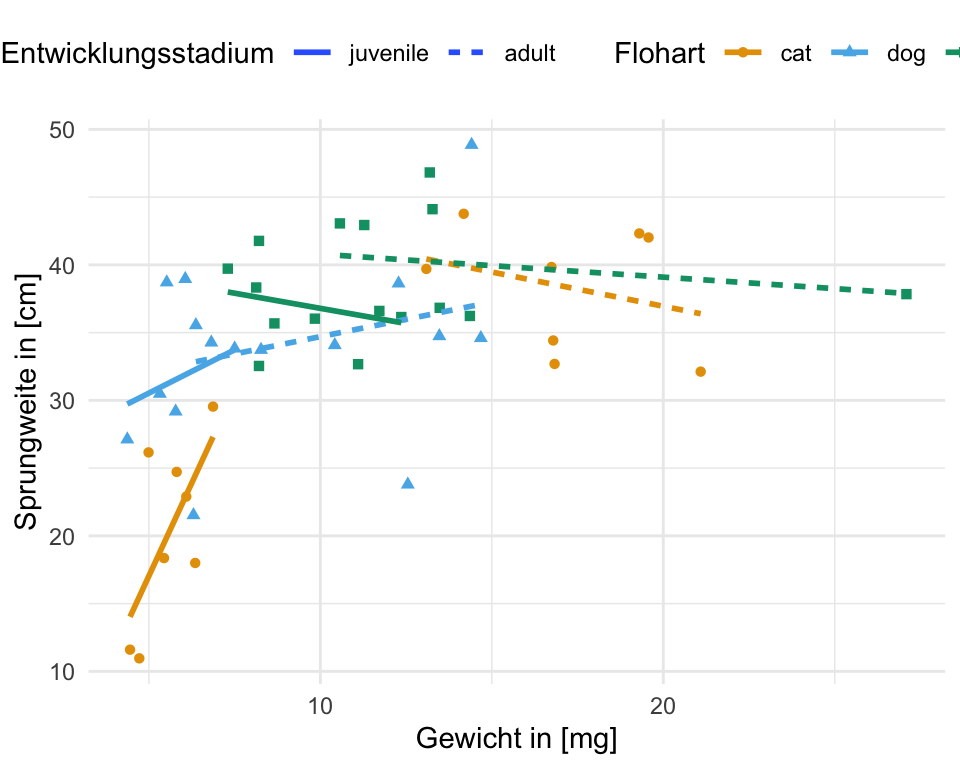
Als einen zweiten Datensatz schauen wir uns dann ein ähnliches Experiment an. Hier haben wir dann aber zwei Faktoren vorliegen. Zum einen betrachten wir wieder die Flohart, schauen uns aber noch zusätzlich den Entwicklungstand der Flöhe an. Dazu haben wir dann als Kovariate noch das Gewicht der einzelnen Flöhe gemessen. Wir fragen uns also, ob es einen Zusammenhang zwischen der Sprungweite und den Floharten sowie dem Entwicklungstand der Flöhe gibt. Darüber hinaus sin eben die Flöhe auch unterschiedlich schwer. Das hat zum einen mit dem Entwicklungsstand zu tun, auf der anderen Seite könnte es aber auch einen Effekt auf die Sprungweite haben.
R Code [zeigen / verbergen]
fac2_cov_tbl <- read_excel("data/fleas_complex_data.xlsx", sheet = "covariate-fac2") |>
select(animal, stage, weight, jump_length) |>
mutate(animal = as_factor(animal),
stage = factor(stage, level = c("juvenile", "adult")),
weight = round(weight, 2),
jump_length = round(jump_length, 2)) |>
rownames_to_column(".id")In der folgenden Tabelle siehst du dann einmal die Sprungweite in [cm] und das Gewicht in [mg] für die Hunde-, Katzen- und Fuchsflöhen dargestellt. Dabei ist das Gewicht kontinuierlich gemessen und die Flohart sowie der Entwicklungsstand sind dann die kategoriellen Faktor.
| animal | stage | weight | jump_length |
|---|---|---|---|
| cat | adult | 16.74 | 39.84 |
| cat | adult | 16.79 | 34.42 |
| cat | adult | 19.3 | 42.32 |
| … | … | … | … |
| fox | juvenile | 7.3 | 39.72 |
| fox | juvenile | 8.66 | 35.68 |
| fox | juvenile | 8.13 | 38.33 |
Auch hier schauen wir uns in der folgenden Abbildung einmal den Zusammenhang zwischen der Sprungweite und dem Gewicht in einem Scatterplot an. Wir sehen, dass mit ansteigendem Gewicht auch die Flöhe weiter springen. Was wir auch sehen ist, dass sich dieser Effekt dann doch in den einzelnen Faktorkombinationen aus der Flohart und dem Entwicklungsstadium unterscheidet. Die Steigungen der Geraden sind nicht überall gleich. Wir haben vermutlich eine Interaktion vorliegen, die wir uns dann in der ANCOVA nochmal näher anchauen müssen.
33.5 Einfaktoriell
Häufig wenn es um die einfaktorielle ANCOVA geht, herrscht etwas Verwirrung, da wir ja dann doch zwei Variablen auf der rechten Seite der Tilde ~ haben. Hier haben wir dann aber einen Faktor und eine numerische Kovariate vorliegen. Deshalb ist die einfaktorielle ANCOVA auch nur einfaktoriell, da wir nur einen Faktor betrachten. Ich zeige hier wieder die Anwendung in verschiedenen R Paketen. Die Ergebnisse sind alle sehr ähnlich und eigentlich sticht hier nur das R Paket {car} hervor, da wir auch hier für Varianzheterogenität adjustieren können. Mehr dazu dann auch in dem entsprechenden Kapitel zu der ANOVA.
Beginnen wir mit der Standardfunktion aov() für die einfaktorielle ANCOVA. Wir nehmen einfach ein Modell und spezifizieren noch die Datenquelle. Dann nutze ich noch die Funktion tidy() aus dem R Paket {broom} für eine schönere Ausgabe. Dann habe ich alles zusammen.
R Code [zeigen / verbergen]
aov(jump_length ~ animal*weight, data = fac1_cov_tbl) |>
tidy()# A tibble: 4 × 6
term df sumsq meansq statistic p.value
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 animal 2 877. 438. 15.7 0.000115
2 weight 1 665. 665. 23.8 0.000122
3 animal:weight 2 26.4 13.2 0.471 0.632
4 Residuals 18 504. 28.0 NA NA Wir können die Ausgabe der Funktion aov() direkt weiter bei {emmeans} verwenden. Die etwas allgemeinere Form eine einfaktorielle ANOVA zu rechnen, ist erst das lineare Modell zu schätzen. Hier nutze ich einmal eine lineare Regression mit der Funktion lm(). Dann nutzen wir aber die Funktion anova() um die ANOVA zu rechnen.
R Code [zeigen / verbergen]
lm(jump_length ~ animal*weight, data = fac1_cov_tbl) |>
anova() |>
tidy()# A tibble: 4 × 6
term df sumsq meansq statistic p.value
<chr> <int> <dbl> <dbl> <dbl> <dbl>
1 animal 2 877. 438. 15.7 0.000115
2 weight 1 665. 665. 23.8 0.000122
3 animal:weight 2 26.4 13.2 0.471 0.632
4 Residuals 18 504. 28.0 NA NA Wir sind am Ende aber nur an den p-Werten interessiert und auch hier ist der p-Wert signifikant, der unter dem Signifikanzniveau \(\alpha\) gleich 5% liegt. Wir haben also einen signifikanten Einfluss der Flohart wie auch von dem Gewicht des Flohs auf die Sprungweite. Wie du siehst sind die Ergebnisse numerisch gleich. Warum dann überhaupt zwei Funktionen? Mit lm() und anova() bist du etwas flexibler was das genutzte Modell angeht.
Jetzt könnte man meinen, warum noch eine weitere Implementierung der ANOVA, wenn wir schon die Funktion aov() haben? Die Funktion aov() rechnet grundsätzlich Type I ANCOVAs. Das macht jetzt bei einer einfaktoriellen ANOVA nichts aus, aber wir können eben in {car} auch Type II und Type III ANOVAs rechnen. Darüber hinaus erlaubt das Paket auch für Varianzheterogenität in der ANOVA zu adjustieren.
Varianzhomogenität
In dem klassischen Aufruf der Funktion Anova() nutzen wir wieder ein Modell und dann gebe ich dir wieder die aufgeräumte Ausgabe mit tidy() wieder. Wenn du nichts extra angibst, dann rechnest du natürlich eine ANOVA unter der Annahme der Varianzhomogenität.
R Code [zeigen / verbergen]
lm(jump_length ~ animal*weight, data = fac1_cov_tbl) |>
Anova() |>
tidy()# A tibble: 4 × 5
term sumsq df statistic p.value
<chr> <dbl> <dbl> <dbl> <dbl>
1 animal 261. 2 4.67 0.0233
2 weight 665. 1 23.8 0.000122
3 animal:weight 26.4 2 0.471 0.632
4 Residuals 504. 18 NA NA Wenn du die Zahlen mit der Funktion aov() vergleichst, siehst du das da fast die gleichen Werte rauskommen. Das sollte natürlich auch in einer einfaktoriellen ANOVA mit den gleichen Annahmen so sein. Wir nutzen hier aber einen leicht anderen Weg, os dass wir dann auch etwas andere p-Werte rauskriegen. Die Entscheidung ist jedoch in beiden Fällen die gleiche. Die Flohart und das Gewicht der Flöhe ist signifikant.
Varianzheterogenität
Der Vorteil des Pakets {car} ist, dass wir in der Funktion Anova() auch für Varianzheterogenität adjustieren können. Wir nutzen dazu die Option white.adjust = TRUE. Dann haben wir auch etwas andere Werte für die Abweichungsquadrate.
R Code [zeigen / verbergen]
lm(jump_length ~ animal*weight, data = fac1_cov_tbl) |>
Anova(white.adjust = TRUE) |>
tidy()Coefficient covariances computed by hccm()# A tibble: 4 × 4
term df statistic p.value
<chr> <dbl> <dbl> <dbl>
1 animal 2 4.24 0.0309
2 weight 1 23.3 0.000134
3 animal:weight 2 0.338 0.718
4 Residuals 18 NA NA Wir sind am Ende aber nur an den p-Werten interessiert und auch die sind hier signifikant, da der p-Wert unter dem Signifikanzniveau \(\alpha\) gleich 5% liegt.
Das R Paket {afex} ist eigentlich für Messwiederholungen am besten geeignet, aber ich zeige es auch hier im einfaktoriellen Fall. Das einzige was etwas anders ist, ist das wir eine ID für die Funktion brauchen. Das Paket {afex} möchte nämlich explizit wissen, welcher Wert zu welcher Beobachtung gehört.
R Code [zeigen / verbergen]
aov_car(jump_length ~ animal*weight + Error(.id), data = fac1_cov_tbl,
factorize = FALSE) Anova Table (Type 3 tests)
Response: jump_length
Effect df MSE F ges p.value
1 animal 2, 18 27.98 0.09 .010 .916
2 weight 1, 18 27.98 13.76 ** .433 .002
3 animal:weight 2, 18 27.98 0.47 .050 .632
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1Hier kriegen wir dann in der Type III ANCOVA tatsächlich was anderes raus. Auf einmal ist unsere Flohart nicht mehr signifikant, da der p-Wert über dem Signifikanzniveau \(\alpha\) gleich 5% liegt. Hier macht es dann Sinn einmal eine Type II ANCOVA zu rechnen und den nicht signifikanten Interaktionsterm einmal aus dem Modell zu nehmen.
R Code [zeigen / verbergen]
aov_car(jump_length ~ animal + weight + Error(.id), data = fac1_cov_tbl,
factorize = FALSE, type = "II") Anova Table (Type II tests)
Response: jump_length
Effect df MSE F ges p.value
1 animal 2, 20 26.50 4.93 * .330 .018
2 weight 1, 20 26.50 25.08 *** .556 <.001
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 133.6 Zweifaktoriell
Betrachten wir jetzt nochmal die zweifaktorielle ANCOVA mit einer zusätzlichen Kovariate. Hier haben wir dann natürlich die Möglichkeit eine Menge mehr an Interaktionen zu betrachten. Auch wird hier dann die Wahl des Algorithmus immer wichtiger. Je nachdem welches Paket du nutzt, kiegst du dann auch leicht andere Werte raus. Teilweise sind die Abweichungen dann auch stärker als ich es selber erwartet hätte, aber das ist eben so manchmal der Fall. Beginnen wir also unsere Analyse der Sprunglänge in Abhängigkeit der Flohart sowie dem Entwicklungsstand und dem Flohgewicht.
Beginnen wir mit der Standardfunktion aov() für die zweifaktorielle ANCOVA. Wir nehmen einfach ein Modell und spezifizieren noch die Datenquelle. Wichtig ist, dass die Reihenfolge der Faktoren in deinem Modell eine Rolle spielt. Daher nehme immer deine wichtigere Behandlung als erstes in das Modell oder nutze eine Type II ANOVA im R Paket {car}. Dann nutze ich noch die Funktion tidy() aus dem R Paket {broom} für eine schönere Ausgabe. Dann habe ich alles zusammen.
R Code [zeigen / verbergen]
aov(jump_length ~ animal*stage*weight, data = fac2_cov_tbl) |>
tidy()# A tibble: 8 × 6
term df sumsq meansq statistic p.value
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 animal 2 687. 344. 11.6 0.000127
2 stage 1 818. 818. 27.7 0.00000673
3 weight 1 132. 132. 4.47 0.0414
4 animal:stage 2 448. 224. 7.57 0.00180
5 animal:weight 2 32.0 16.0 0.542 0.586
6 stage:weight 1 20.9 20.9 0.708 0.406
7 animal:stage:weight 2 146. 73.2 2.48 0.0983
8 Residuals 36 1064. 29.6 NA NA Wir können die Ausgabe der Funktion aov() direkt weiter bei {emmeans} verwenden. Die etwas allgemeinere Form eine zweifaktorielle ANOVA zu rechnen, ist erst das lineare Modell zu schätzen. Auch hier ist die Reihenfolge von Wichtigkeit. Hier nutze ich einmal eine lineare Regression mit der Funktion lm(). Dann nutzen wir aber die Funktion anova() um die ANCOVA zu rechnen.
R Code [zeigen / verbergen]
lm(jump_length ~ animal*stage*weight, data = fac2_cov_tbl) |>
anova() |>
tidy()# A tibble: 8 × 6
term df sumsq meansq statistic p.value
<chr> <int> <dbl> <dbl> <dbl> <dbl>
1 animal 2 687. 344. 11.6 0.000127
2 stage 1 818. 818. 27.7 0.00000673
3 weight 1 132. 132. 4.47 0.0414
4 animal:stage 2 448. 224. 7.57 0.00180
5 animal:weight 2 32.0 16.0 0.542 0.586
6 stage:weight 1 20.9 20.9 0.708 0.406
7 animal:stage:weight 2 146. 73.2 2.48 0.0983
8 Residuals 36 1064. 29.6 NA NA Wir sind am Ende aber nur am p-Wert der beiden Faktoren und der Kovariate interessiert und auch der ist hier für die Floharten animal signifikant, da der p-Wert unter dem Signifikanzniveau \(\alpha\) gleich 5% liegt. Der p-Wert für den Messort stage ist ebenfalls signifkant. Das Gewicht ist nur leicht signifikant, da der p-Wert nahe an 5% liegt. Wir haben eine signifikante Interaktion animal:stage vorliegen.
Jetzt könnte man meinen, warum noch eine weitere Implementierung der ANOVA, wenn wir schon die Funktion aov() haben? Die Funktion aov() rechnet grundsätzlich Type I ANCOVAs. Das macht jetzt bei einer zweifaktoriellen ANCOVA schon einen Unterschied welchen Typ ANOVA wir rechnen. Wir können eben in {car} auch Type II und Type III ANCOVAs rechnen. Darüber hinaus erlaubt das Paket auch für Varianzheterogenität in der ANOVA zu adjustieren. Häufig haben wir dann eine Interaktion vorliegen, da wir bei drei Variablen dann schon eher eine signifikante Interaktion finden. Wenn du keine Interaktion vorliegen hast, dann nutze die Type II ANCOVA.
Varianzhomogenität
In dem klassischen Aufruf der Funktion Anova() nutzen wir wieder ein Modell und dann gebe ich dir wieder die aufgeräumte Ausgabe mit tidy() wieder. Wenn du nichts extra angibst, dann rechnest du natürlich eine ANOVA unter der Annahme der Varianzhomogenität.
R Code [zeigen / verbergen]
lm(jump_length ~ animal*stage*weight, data = fac2_cov_tbl,
contrasts = list(
animal = "contr.sum",
stage = "contr.sum"
)) |>
Anova(type = "III") |>
tidy() |>
mutate(p.value = pvalue(p.value))# A tibble: 9 × 5
term sumsq df statistic p.value
<chr> <dbl> <dbl> <dbl> <chr>
1 (Intercept) 1205. 1 40.8 <0.001
2 animal 153. 2 2.58 0.090
3 stage 163. 1 5.53 0.024
4 weight 85.9 1 2.90 0.097
5 animal:stage 196. 2 3.31 0.048
6 animal:weight 126. 2 2.14 0.133
7 stage:weight 95.3 1 3.22 0.081
8 animal:stage:weight 146. 2 2.48 0.098
9 Residuals 1064. 36 NA <NA> Hier ändert sich jetzt doch einiges unter der Berücksichtigung der Interaktion in dem Modell der ANCOVA. Wir sehen aber jetzt nur noch eine schwache Interaktion und könnten überlegen einfach nur eine Type II ANCOVA mit dem Haupteffekten zu rechnen.
R Code [zeigen / verbergen]
lm(jump_length ~ animal + stage + weight, data = fac2_cov_tbl) |>
Anova(type = "II") |>
tidy() |>
mutate(p.value = pvalue(p.value))# A tibble: 4 × 5
term sumsq df statistic p.value
<chr> <dbl> <dbl> <dbl> <chr>
1 animal 670. 2 8.42 <0.001
2 stage 89.8 1 2.26 0.140
3 weight 132. 1 3.32 0.075
4 Residuals 1711. 43 NA <NA> Das sieht dann schonmal etwas anders aus und eigentlich auch überzeugend. Wir haben jetzt einen statistischen Engel erwischt, da wir die leicht signifikante Interaktion ignorieren, aber damit können wir leben.
Varianzheterogenität
Der Vorteil des Pakets {car} ist ja, dass wir in der Funktion Anova() auch für Varianzheterogenität adjustieren können. Wir nutzen dazu die Option white.adjust = TRUE. Ja, ich weiß, das ist überhaupt nicht naheliegend. Wenn wir das machen, dann werden die Abweichungsquadrate anders berechnet und unsere Ausgabe ändert sich entsprechend. Auch hier nutze ich dann mal die Type II ANOVA und betrachte nur die Haupteffekte.
R Code [zeigen / verbergen]
lm(jump_length ~ animal + stage + weight, data = fac2_cov_tbl) |>
Anova(type = "II", white.adjust = TRUE) |>
tidy()Coefficient covariances computed by hccm()# A tibble: 4 × 4
term df statistic p.value
<chr> <dbl> <dbl> <dbl>
1 animal 2 7.52 0.00158
2 stage 1 1.58 0.216
3 weight 1 1.12 0.297
4 Residuals 43 NA NA Unter der Berücksichtigung der Varianzheterogenität ist dann unser Gewicht auf einmal dann doch nicht mehr signifikant. Spannend, was die unterschiedlichen Algorithmen dann alles rauskriegen. Ob hier dann dieses Ergebnisses wahrer ist als bei der Varianzhomogenität kann man diskutieren. Hier muss man sich die Sachlage dann nochmal in einer Regression anschauen. Dann kann ich die Effektschätzer besser interpretieren.
Der Vorteil von dem R Paket {afex} ist, dass wir hier mit der Funktion aov_car() eine Type III ANCOVA rechnen, die korrekt angepasst ist. Wir müssen also nicht noch extra ein Modell mit Kontrasten anpassen. Wir können auch die ANOVA Type II rechnen. Eine Besonderheit ist, dass wir einen Fehlerterm mit Error() der Funktion übergeben müssen, damit {afex} rechnen kann. Die Stärke von {afex} liegt eigentlich in der repeated und mixed ANOVA. Dort brauchen wir dann die Information zwingend, welche Zeile zu welcher Beobachtung gehört.
R Code [zeigen / verbergen]
aov_car(jump_length ~ animal*stage*weight + Error(.id), data = fac2_cov_tbl,
factorize = FALSE) Anova Table (Type 3 tests)
Response: jump_length
Effect df MSE F ges p.value
1 animal 2, 36 29.56 2.58 + .125 .090
2 stage 1, 36 29.56 5.53 * .133 .024
3 weight 1, 36 29.56 2.90 + .075 .097
4 animal:stage 2, 36 29.56 3.31 * .155 .048
5 animal:weight 2, 36 29.56 2.14 .106 .133
6 stage:weight 1, 36 29.56 3.22 + .082 .081
7 animal:stage:weight 2, 36 29.56 2.48 + .121 .098
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1Am Ende erhalten wir hier nur noch einen signifikanten Unterschied für den Faktor stage und damit dem Entwicklungsstand. Hier muss man dann auch nochmal spielen und schauen, was die anderen Typen der ANCOVA ergeben. Da ja auch hier nur eine marginale signifikante Interaktion zwischen den Floharten und dem Entwicklungsstand vorliegt, würde ich auch hier zu einer Type II ANCOVA greifen.