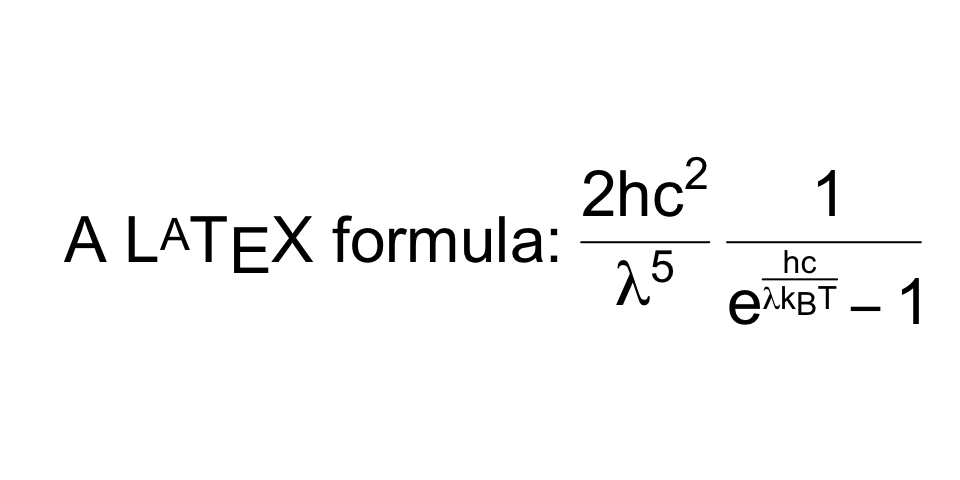# Ein `{ggplot}` Cookbook {#sec-ggplot-cookbook}
*Letzte Änderung am `r format(fs::file_info("programing-ggplot.qmd")$modification_time, '%d. %B %Y um %H:%M:%S')`*
```{r}
#| warning: false
#| echo: false
pacman::p_load(tidyverse, readxl, knitr, kableExtra, conflicted)
cb_pal <- c("#000000", "#E69F00", "#56B4E9", "#009E73",
"#F0E442", "#F5C710", "#0072B2", "#D55E00", "#CC79A7")
```
> *"Frei ist, wer missfallen kann." --- Annette Oschmann in [Mädchen stärken](https://www.annette-oschmann.de/maedchen-staerken/)*
Willkommen in einer weiteren wilden Linksammlung und R Code sowie einiges an interessanten R Paketen im Kontext der Visualisierung von Daten. Hier dreht sich dann fast alles um das R Paket `{ggplot}` und wie wir dieses R Paket sinnvoll nutzen können. Daher ist alles hier sehr stückhaft und manchmal einfach nur da. Das hat den Grund, dass ich das Kapitel hier eher als Steinbruch nutze als ein echtes Kapitel zum von oben nach unten Lesen. Sehe das Kapitel also eher als Referenzsammlung als ein Lernkapitel. Es lädt dann zum Stöbern ein. Es sind also eher kleine Rezepte in einem Kochbuch. Am Ende kommst du vermutlich aber nicht um @wickham2016r und [R for Data Science (2e)](https://r4ds.hadley.nz/) oder aber dem Buch [ggplot2: Elegant Graphics for Data Analysis (3e)](https://ggplot2-book.org/annotations.html) herum. Am Ende kann ich noch das [R Graphics Cookbook](https://r-graphics.org/) empfehlen, wenn es um konkrete Probleme geht.
::: {.callout-caution collapse="true"}
## Was soll das hier? Was für ein Kochbuch?
{#fig-math-cartoon fig-align="center" width="100%"}
Dieses Kapitel dient dazu Programmierung in `{ggplot}` zu präsentieren. Es ist eine Sammlung von Code und Rezepten, die ich immer mal wieder nutze und immer wieder vergesse. Deshalb hat dieses Kapitel auch den Titel *Cookbook*. Wenn dich also Rumfummeln in `{ggplot}` interessiert, dann kannst du dir hier noch was anschauen. Das Kapitel ist eigentlich nie fertig, da ich sicherlich immer mal wieder was ergänzen werde.
:::
## Allgemeiner Hintergrund
In diesem Kapitel geht es hauptsächlich um das R Paket `{ggplot}`. Korrekterweise müsste ich natürlich eigentlich noch die korrekte Version ergänzen, aber wir schreiben selten `{ggplot2}` da eigentlich niemand mehr die uralte Version nutzt. Warum dieses Kapitel? Weil es eben dann doch so viel zu entdecken gibt, dass ich mir nicht alles merken kann. Daher habe ich einmal hier die Sammlung an nützlichen R Paketen um `{ggplot}` erstellt. Darüber hinaus war dann auch das [Kapitel zur Visualisierung](#sec-eda-ggplot) einfach zu voll. Viele wollen dann einfach einen `{ggplot}` erstellen und nicht noch durch zig Pakete scrollen. Daher ist das hier eher für mich oder für Leute, die mal rechts und links gucken wollen was da noch am Straßenrand liegt. Vielleicht findest du was, was dich inspiriert. Daher auch die Einteilung in Abschnitte, wo ich ein Problem löse und dann nochmal Abschnitte mit teilweise den gleichen R Paketen als Vorstellung.
::: callout-tip
## Woher habe ich alle Pakete her?
Wenn es einen Link zu einer Seite gibt, den ich immer mal wieder Besuche, ist es der folgende Link: [ggplot2 extensions](https://exts.ggplot2.tidyverse.org/gallery/). Du erfährst hier eine Menge neues an Paketen und es gibt immer wieder spannendes zu entdecken. Ein Besuch lohnt sich auf jeden Fall.
:::
## Genutzte R Pakete
Wir wollen folgende R Pakete in diesem Kapitel nutzen.
```{r echo = TRUE}
#| message: false
pacman::p_load(tidyverse, readxl, latex2exp, see, duke,
wesanderson, ggbreak, patchwork, ggrepel,
tidyplots, ggh4x, ggtext, gganimate,
ggpmisc, broom, ggdist, distributional, conflicted)
conflicts_prefer(ggplot2::annotate)
```
An der Seite des Kapitels findest du den Link *Quellcode anzeigen*, über den du Zugang zum gesamten R-Code dieses Kapitels erhältst.
## Daten
Beginnen wir hier nochmal mit einem Datenbeispiel für unsere Flöhe. Wir schauen uns hier einmal an, ob es einen Unterschied in den Ernährungsformen sowie dem Entwicklungsstand gibt. Dabei messen wir dann wieder die Sprungweite. Darüber hinaus nehme ich noch das Gewicht der Flöhe mit in unseren Datensatz. Damit haben wir auch schon genug um mal etwas hier mit `{ggplot}` spielen zu können. Ob hier irgendwas interessantes in den Daten vorliegt interessiert uns eigentlich ja nicht. Hier geht es ums visualisieren.
```{r}
#| message: false
ggplot_tbl <- read_xlsx("data/fleas_model_data.xlsx") |>
mutate(feeding = as_factor(feeding),
stage = as_factor(stage)) |>
select(feeding, stage, weight, jump_length)
```
Betrachten wir als erstes einen Auszug aus der Datentabelle. Wir werden hier immer den Messwert auf die y-Achse legen und uns dann je nach Anwendung sowie Algorithmus für eine Einflussvariable entscheiden.
```{r}
#| echo: false
#| message: false
#| warning: false
#| label: tbl-model-table
#| tbl-cap: "Tabelle der vier Variablen für die Darstellung mit `{ggplot}`. Der Messwert ist hier die Sprungweite. Die anderen Variablen dienen als Einflussvariablen."
ggplot_raw_tbl <- read_xlsx("data/fleas_model_data.xlsx") |>
select(feeding, stage, weight, jump_length)
rbind(head(ggplot_raw_tbl, n = 3),
rep("...", times = ncol(ggplot_raw_tbl)),
tail(ggplot_raw_tbl, n = 3)) |>
kable(align = "c", "pipe")
```
Bitte den Datensatz hier nicht allzu überinterpretieren. Ich brauche einfach nur ein paar Daten um die Funktionen aufrufen zu können. Da ich aber nicht die generischen Daten nutzen will, mache ich hier alles mit meinen Flöhen. Das sind ja meine Tiere hier in der Bio Data Science und deshalb nehmen wir das hier mal auch als Beispiel.
## Probleme von Interesse
Im Folgenden dann noch eine Sammlung an nützlichen Optionen und Möglichkeiten, die einem das Leben einfacher machen und die Abbildungen dann noch schöner. Nicht alles musst du in `{ggplot}` machen, manchmal geht es dann in PowerPoint dann doch schneller mal eben einen Text zu ergänzen. Sehe das hier deshalb als Ergänzung und meinen privaten Raum, den ich nutze um mir den Code zu merken. Viele der Probleme sind dann auch eher so Sachen, die du mit der Zeit als erstrebenswert empfinden magst.
### Striche, Punkte und Text
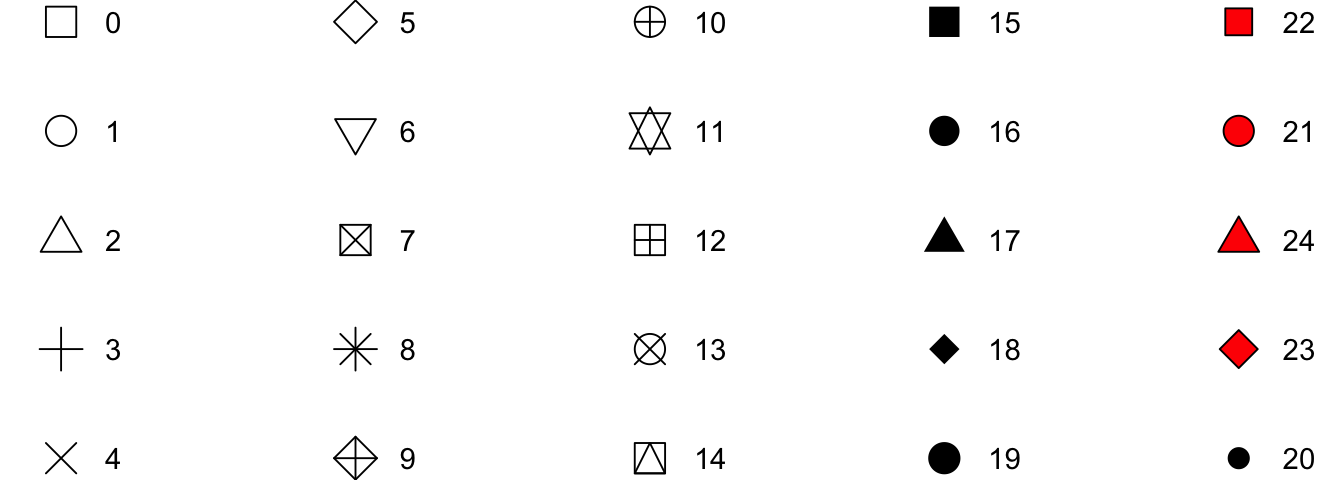
Der folgende Code und die entsprechenden Abbildungen sind einfach nur Copy& Paste von der Vignette [Aesthetic specifications in `{ggplot}`](https://ggplot2.tidyverse.org/articles/ggplot2-specs.html) und dienen nur meinem schnellen Zugriff. Dann habe ich hier alles was ich brauche einmal zusammen. Häufig sucht man dann doch nochmal die Form eines Punktes oder die Art der Linie.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-ggplot-shape
#| fig-align: center
#| fig-height: 2.5
#| fig-width: 7
#| fig-cap: "Verschiedene Formen der Punkte die durch die `shape`-Option geändert werden können. Teilweise lassen sich die Punkte auch mit der Option `fill` entsprechebd einfärben. *[Zum Vergrößern anklicken]*"
shapes <- data.frame(
shape = c(0:19, 22, 21, 24, 23, 20),
x = 0:24 %/% 5,
y = -(0:24 %% 5)
)
ggplot(shapes, aes(x, y)) +
geom_point(aes(shape = shape), size = 5, fill = "red") +
geom_text(aes(label = shape), hjust = 0, nudge_x = 0.15) +
scale_shape_identity() +
expand_limits(x = 4.1) +
theme_void()
```
Dann gibt es noch die Möglichkeit sich verschiedene Arten von Linien zeichnen zu lassen. Im Folgenden dann erstmal der Standard, der dann manchmal nicht so richtig passt. Dafür gibt es dann aber gleich nochmal eine Option die Abstände zwischen den Teillinien zu ändern.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-ggplot-linetype
#| fig-align: center
#| fig-height: 2.5
#| fig-width: 7
#| fig-cap: "Verschiedene Arten der Linien die durch die `linetype`-Option geändert werden können. *[Zum Vergrößern anklicken]*"
lty <- c("solid", "dashed", "dotted", "dotdash", "longdash", "twodash")
linetypes <- data.frame(
y = seq_along(lty),
lty = lty
)
ggplot(linetypes, aes(0, y)) +
theme_void() +
geom_segment(aes(xend = 5, yend = y, linetype = lty)) +
scale_linetype_identity() +
geom_text(aes(label = lty), hjust = 0, nudge_y = 0.2) +
scale_x_continuous(NULL, breaks = NULL) +
scale_y_reverse(NULL, breaks = NULL)
```
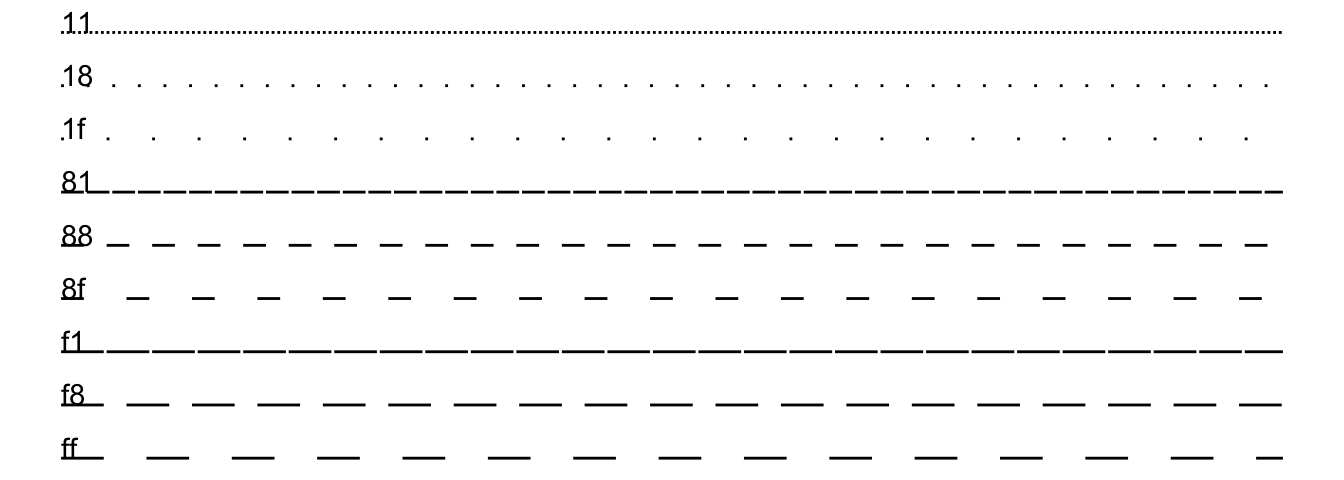
Manchmal will ich dann aber nicht so große Abstände haben. Hier können wir dann mit der Option und verschiedenen Buchstaben spielen. Mehr gibt es in der Vignette [Aesthetic specifications in `{ggplot}`](https://ggplot2.tidyverse.org/articles/ggplot2-specs.html) dazu. Hier reichen mir die folgenden Beispiele um mal schnell die passende Linie zu finden.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-ggplot-linetype-2
#| fig-align: center
#| fig-height: 2.5
#| fig-width: 7
#| fig-cap: "Verschiedene Arten der Linien die durch die `linetype`-Option geändert werden können. Hier können dann individuell die Abstände zwischen den Linien angepasst werden. *[Zum Vergrößern anklicken]*"
lty <- c("11", "18", "1f", "81", "88", "8f", "f1", "f8", "ff")
linetypes <- data.frame(
y = seq_along(lty),
lty = lty
)
ggplot(linetypes, aes(0, y)) +
theme_void() +
geom_segment(aes(xend = 5, yend = y, linetype = lty)) +
scale_linetype_identity() +
geom_text(aes(label = lty), hjust = 0, nudge_y = 0.2) +
scale_x_continuous(NULL, breaks = NULL) +
scale_y_reverse(NULL, breaks = NULL)
```
Manchmal wollen wir dann noch den Text positionieren. Das können wir dann entweder durch die Option `hjust` mit den Worten `"left"`, `"center"` oder `"right"` machen oder eben dann mit Zahlen. Das ganz geht dann neben der horizontalen Ebene auch mit `vjust` auf der vertikalen Ebene. Für mich ist es immer etwas Voodoo und ich schaue hier dann immer regelmäßig rein.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-ggplot-just
#| fig-align: center
#| fig-height: 2
#| fig-width: 7
#| fig-cap: "Positionen von Text durch die Optionen `vjust` und `hjust`. *[Zum Vergrößern anklicken]*"
just <- expand.grid(hjust = c(0, 0.5, 1), vjust = c(0, 0.5, 1))
just$label <- paste0(just$hjust, ", ", just$vjust)
ggplot(just, aes(hjust, vjust)) +
theme_void() +
geom_point(colour = "grey70", size = 3) +
geom_text(aes(label = label, hjust = hjust, vjust = vjust))
```
Natürlich geht wie immer auch mehr. Dazu dann aber mehr in der passenden Vignette oder eben auf den entsprechenden Hilfeseiten von `{ggplot}`. Aus meiner Erfahrung habe ich dann meistens nicht mehr gebraucht oder es mir dann situativ zusammengesucht.
### Überschriften, Achsen und Legenden
Die Abbildungen einfach nur so machen ist eigentlich nur in einer reinen explorativen Datenanalyse sinnvoll. Sobald du die Abbildungen dann irgendwie veröffentlichen willst, dann sind die normalen Beschriftungen der Achsen in `{ggplot}` mit den Variablennamen nicht mehr schön. Wenn du dann mehr machen willst, also die Überschriften anpassen oder aber die Achsenbeschriftung ändern, dann gibt es hier global Hilfe im [`{ggplot}` Manual](https://ggplot2.tidyverse.org/reference/index.html). In den folgenden Abbildungen siehst du einmal einen veränderten Titel und Beschriftungen sowie die Legenden. Die Möglichkeiten sind nahezu unbegrenzt und sprengen auch hier den Rahmen. Die Webseite [R Cookbook](https://ggplot2.tidyverse.org/reference/index.html) hat auch spezielle Hilfe für `{ggplot}`.
#### Überschriften und Achsenbeschriftung {.unnumbered .unlisted}
Die [Überschriften von Abbildungen](http://www.cookbook-r.com/Graphs/Titles_(ggplot2)/) können wir beliebig anpassen. Hier können wir die Funktion `ggtitle()` oder aber die Funktion `labs()` nutzen. Die [Achsenbeschriftung](http://www.cookbook-r.com/Graphs/Axes_(ggplot2)/) können wir in der Funktion `labs()` einfach ändern. Das ist dann am Ende immer eine Geschmacksfrage was dir besser gefällt. Ich nutze hier gerne die Funktion `labs()`, dann habe ich alles zusammen.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-title-labs
#| fig-align: center
#| fig-height: 4
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit Titel und geänderter Achsenbeschrittung. *[Zum Vergrößern anklicken]*"
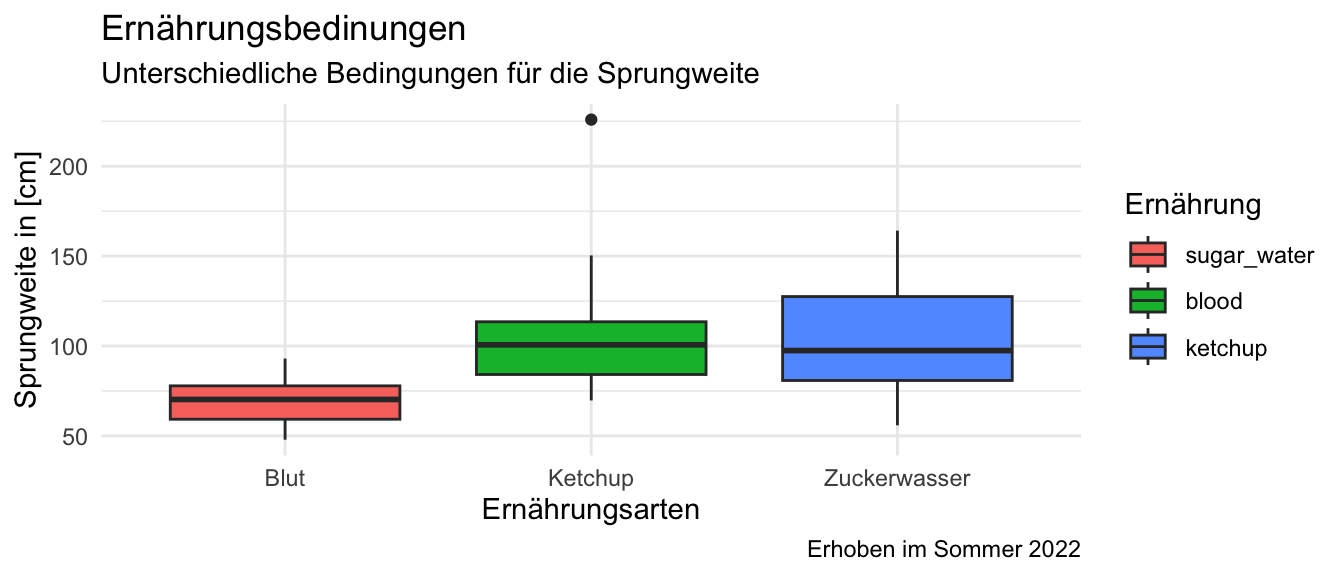
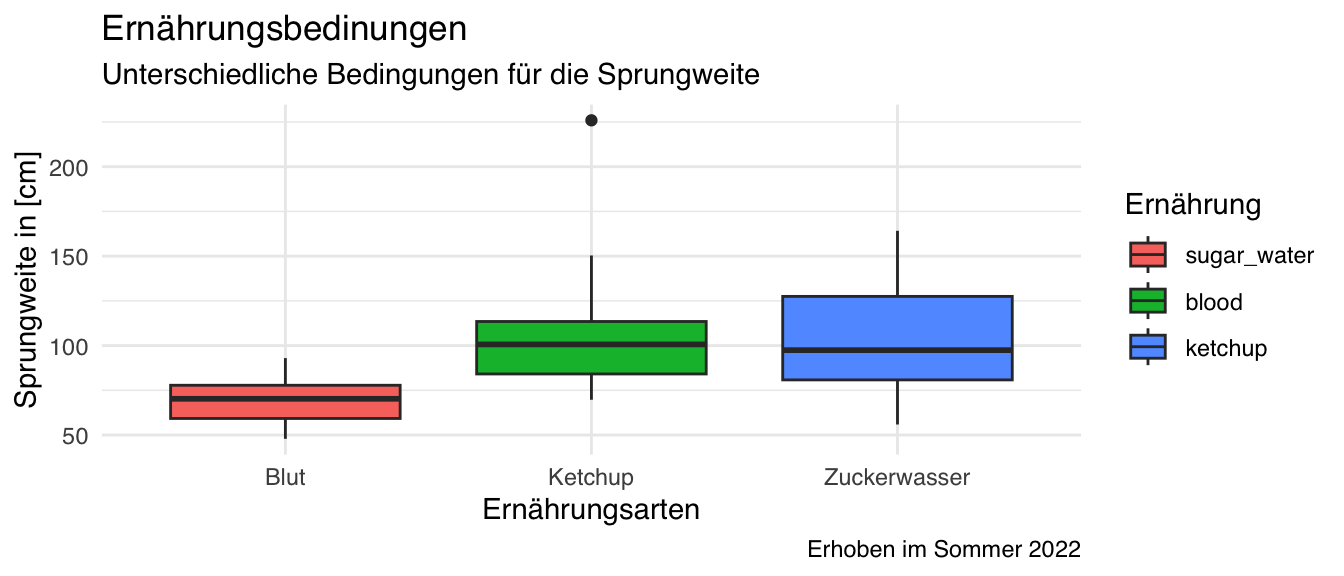
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
labs(title = "Ernährungsbedinungen",
subtitle = "Unterschiedliche Bedingungen für die Sprungweite",
caption = "Erhoben im Sommer 2022",
x = "Ernährungsarten", y = "Sprungweite in [cm]",
fill = "Ernährung")
```
#### Legende anpassen {.unnumbered .unlisted}
Die [Legende](http://www.cookbook-r.com/Graphs/Legends_(ggplot2)/) können wir ebenfalls in `labs()` und dann in den entsprechenden `scales`-Funktionen anpassen. Hier musst du im Zweifel etwas probieren. Faktoren würde ich in den Daten über die Funktion `factor()` anpassen. Du hast dann hier eben auch die Wahl wie du vorgehst. Manchmal macht es dann auch Sinn dir ein eigenes `tibble` für die Erstellung der Abbildungen zu bauen. In den beiden folgenden Tabs zeige ich dir mal beide Wege.
::: panel-tabset
## `factor()`
Wir bauen hier erstmal den Faktor mit der Funktion `factor()` in der wir dann die Sortierung und dann die Labels der Level ändern können. Ich empfehle dir erst die Sortierung zu bestimmen und dann umzubenennen. Sonst kann da schnell was durcheinanderkommen. Hier mag dann auch das [R Paket `{forcats}`](https://forcats.tidyverse.org/) helfen.
```{r}
ggplot_factor_tbl <- ggplot_tbl |>
mutate(feeding = factor(feeding,
levels = c("sugar_water", "blood", "ketchup"),
labels = c("Zuckerwasser", "Blut", "Ketchup")))
```
Dann können wir uns auch schon einmal die folgende Abbildung bauen in dem wir die Level des Faktors geändert haben.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-factor
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit Titel und geänderter Achsenbeschrittung. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_factor_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot()
```
## `scale_*_discrete()`
Es ist auch möglich direkt in `{ggplot}` die Level des Faktors umzubenennen. Das ist genau so gut, wenn wir hier nicht so viel mit der Sortierung ändern wollen. Wie immer würde ich hier empfehlen nur Kosmetik zu machen und den Rest sauber über das [R Paket `{forcats}`](https://forcats.tidyverse.org/) oder eben die Funktion `factor()`
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-discrete
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit Titel und geänderter Achsenbeschrittung. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
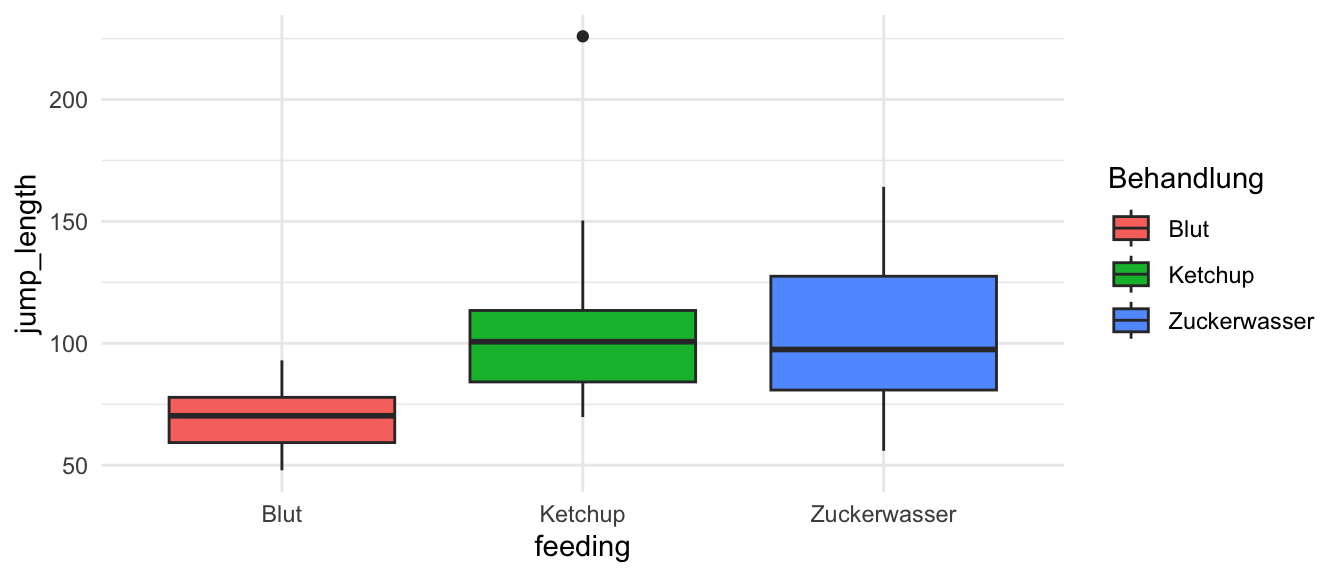
scale_x_discrete(labels = c("Blut", "Ketchup", "Zuckerwasser")) +
scale_fill_discrete(name = "Behandlung",
labels = c("Blut", "Ketchup", "Zuckerwasser"))
```
:::
### Schriftarten
Das Thema Schriftarten ist so eine Sache. Am Ende des Abschnitts gebe ich dir nochmal eine Linksammlung für die Hilfe. Es ist nämlich wichtig in welchem Betriebssystem (eng. *operating system*, abk. *OS*) du arbeitest. Je nach Betriebssystem hast du unterschiedliche Schriften installiert und die lassen sich dann nicht so einfach exportieren oder aber in anderen Betriebssystemen darstellen. Fangen wir also erstmal mit einer Abbildung an, an der wir dann mal was ändern wollen. Wir speichern die Abbildung einmal als Objekt `p1` ab und ändern nachträglich die Schriftarten.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-ggplot-schrift-base
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit Titel und geänderter Achsenbeschrittung in der die Schriftarten geändert werden sollen. *[Zum Vergrößern anklicken]*"
p1 <- ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
scale_x_discrete(labels = c("Blut", "Ketchup", "Zuckerwasser")) +
labs(title = "Ernährungsbedinungen",
subtitle = "Unterschiedliche Bedingungen für die Sprungweite",
caption = "Erhoben im Sommer 2022",
x = "Ernährungsarten", y = "Sprungweite in [cm]",
fill = "Ernährung")
p1
```
Wenn wir allgemein die Schriftart in der ganzen Abbildung ändern wollen, dann können wir einfach folgende Option in `theme()` setzen. Hier ändern wir alles an Text einmal auf `"Times New Roman"` um. Welche Möglichkeiten du hast, hängt von deinem Betriebssystem und deinen installierten Schriften ab. Dazu mehr weiter unten.
```{r}
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
p1 +
theme(text = element_text(family = "Times New Roman"))
```
Wir können auch vollkommen frei drehen und dann alles mögliche ändern. Ich habe hier mal alles verwendet was es so in meinem Schriftordner so gab. Leider ist es dann auch hier wirklich so, dass es keien direkte Übersicht an möglichen Schriften gibt.
```{r}
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
p1 +
theme(plot.title = element_text(family = "mono"),
plot.subtitle = element_text(family = "sans"),
axis.title.x = element_text(family = "Comic Sans MS"),
axis.title.y = element_text(family = "AppleGothic"),
axis.text.x = element_text(family = "Optima"),
axis.text.y = element_text(family = "Luminari"))
```
Am Ende gibt es noch die Möglichkeit eine `mono`, `sans` oder `serif` Schriftfamilie auszuwählen. Dann wird die Standardschrift deines Betriebssystems genommen. Das mag sich dann wiederum unterscheiden, aber dafür hat dann die Schrift die Eigenschaften, die du möchtest.
::: panel-tabset
## `mono`
Mit der Option `"mono"` erhälst du eine nichtproportionale Schrift.
```{r}
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
p1 +
theme(text = element_text(family = "mono"))
```
## `sans`
Mit der Option `"sans"` erhälst du eine serifenlose Schrift.
```{r}
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
p1 +
theme(text = element_text(family = "sans"))
```
## `serif`
Mit der Option `"serif"` erhälst du eine Serifenschrift.
```{r}
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
p1 +
theme(text = element_text(family = "serif"))
```
:::
Dann möchte ich hier am Ende nochmal auf ein paar Blogs und Hilfeseiten verlinken, die sich alle mit dem Thema Schriftarten in `{ggplot}` beschäftigen. Vielleicht ist ja einer der Links exakt das, was du jetzt nochmal nachschauen wolltest. Es ist dann eher ein kompliziertes Thema, was dich einiges an Zeit kosten kann. Die Installation und die Suche der Schriften in einem Betriebssystem ist nervig.
- [Adding Custom Fonts to ggplot in R](https://gradientdescending.com/adding-custom-fonts-to-ggplot-in-r/) stellt das [R Paket `{extrafont}`](https://github.com/wch/extrafont) vor. Ich habe mich erstmal hier dagegen entschieden nochmal das Paket zu zeigen. Ich stelle es aber weiter unten nochmal vor.
- [Changing fonts in ggplot2](https://stackoverflow.com/questions/34522732/changing-fonts-in-ggplot2) geht nochmal auf die Probleme mit dem R Paket `{extrafont}` ein.
- [Using different fonts with ggplot2](https://statisticaloddsandends.wordpress.com/2021/07/08/using-different-fonts-with-ggplot2/) ist die Basis dieses Abschnitts hier. Dort wird nochmal erklärt wie die Schriftarten eingebaut werden können.
- [Using systemfonts to handle fonts in R](https://systemfonts.r-lib.org/articles/systemfonts.html) ist die Vignette von `{ggplot}` wo es nochmal um die Verwendung von Systemschriften in `{ggplot}` geht.
Wenn ich noch was tolles finde, dann werde ich es hier ergänzen. Leider ist es mir nicht gegeben alle Betriebssysteme hier einmal vollumfänglich darzustellen. Das macht ja auch so keinen Sinn. Wenn es für mich klappt, dann muss es auch erstmal reichen.
::: {layout="[15,85]" layout-valign="top"}
{fig-align="center" width="100%"}
> *"Ich persönlich habe eigentlich nie die Schriftarten in `{ggplot}` geändert. Häufig haben wir dann nur die Abbildungen genommen und dann in PowerPoint nochmal die Legenden angepasst, wenn die Schriftarten nicht gepasst haben. Ich halte es eher für ein vorgeschobenes Problem um nicht R und Co. nutzen zu müssen sondern alles in Excel zu machen." --- Jochen Kruppa-Scheetz, meiner bescheidener Meinung nach.*
:::
### Theme anpassen
Am meisten Spaß macht es dann doch nochmal das Thema der `{ggplot}` Abbildung anzupassen. Da geht so einiges wie wir in der Vignette [Modify components of a theme](https://ggplot2.tidyverse.org/reference/theme.html) nachlesen können. Ich zeige hier mal die häufigsten Fragen und dann nochmal mein Thema, welches ich hier im Buch verwende.
#### Position der Legende {.unnumbered .unlisted}
Wo soll die Legende hin? Vielleicht einmal nach oben?
```{r}
#| eval: false
theme(legend.position = "top")
```
Oder soll die Legende gar nicht angezeigt werden?
```{r}
#| eval: false
theme(legend.position = "none")
```
Wenn ein einzelnes `geom_*` nicht angezeigt werden soll, dann kannst du auch den Eintrag in die Legende ausschalten.
```{r}
#| eval: false
geom_point(show.legend = FALSE)
```
#### Eigenes Theme {.unnumbered .unlisted}
Wie du in der Vignette [Modify components of a theme](https://ggplot2.tidyverse.org/reference/theme.html) nachlesen kannst, geht immer eine Menge, wenn es um die Änderung der Abbildung geht. Ich nutze dann immer folgendes Template, wenn es dann um meine Abbildungen geht. Daher verwende ich dann statt `theme_minimal()` in meinen Abbildungen immer `theme_modeling()`, wenn es um Modelle geht. Ich habe dann noch andere Themen in anderen Kapiteln definiert, aber das hier ist dann mein Template.
```{r}
theme_modeling <- function() {
theme_minimal() +
theme(panel.grid.minor = element_blank(),
plot.title = element_text(size = 16, face = "bold"),
plot.subtitle = element_text(size = 12, face = "italic"),
plot.caption = element_text(face = "italic"),
axis.title = element_text(size = 12, face = "bold"),
axis.text = element_text(size = 12),
strip.text = element_text(face = "bold"))
}
```
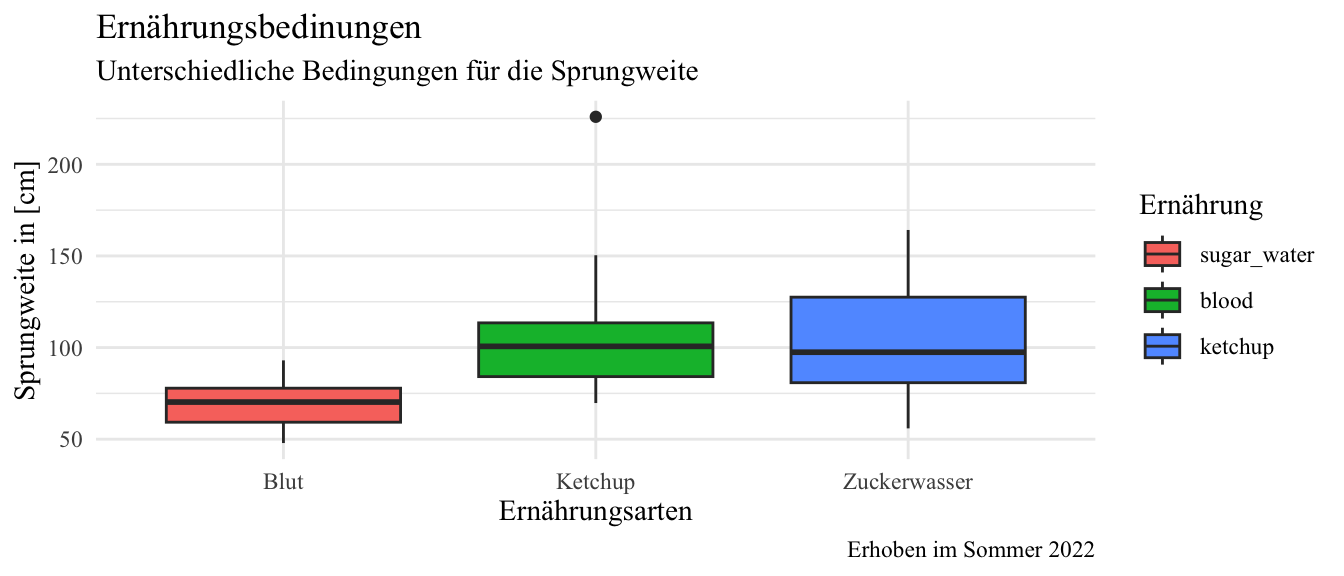
Dann können wir uns auch einmal das Template `theme_modeling()` in der folgenden Abbildung anschauen. Ich habe hier einfach etwas angepasst, so dass ich die Abbildung am Ende schöner fand. Das Farbthema ändere ich dann meistens auch noch, aber das ist dann weiter unten Thema.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-own-theme
#| fig-align: center
#| fig-height: 4
#| fig-width: 7
#| fig-cap: "Beispielhafte Anwendung des Templates `theme_modeling()`. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_modeling() +
geom_boxplot() +
scale_x_discrete(labels = c("Blut", "Ketchup", "Zuckerwasser")) +
labs(title = "Ernährungsbedinungen",
subtitle = "Unterschiedliche Bedingungen für die Sprungweite",
caption = "Erhoben im Sommer 2022",
x = "Ernährungsarten", y = "Sprungweite in [cm]",
fill = "Ernährung")
```
### Abbildungen beschriften
Manchmal willst du auch Abbildungen beschriften. Ich mache das ja hier die ganze Zeit. Alle meine Abbildungen sind in der zweiten Runde dann auf `{ggplot}` umgestellt worden. Ich haben nur noch in alten Kapiteln meine Handzeichnungen drin. Dennoch ist es immer so eine Sache. Dazu dann ein Wort von mir nochmal zum Ende. Hier einmal die beispielhafte Abbildung mit einem Text aus der Funktion `annotate()`. Die Funktion ist sehr mächtig, den wir können so ziemlich jedes `geom_*` auch annotieren. Mehr zur Beschriftung einer `{ggplot}` Abbildung findest du auch im [R Graphics Cookbook](https://r-graphics.org/CHAPTER-ANNOTATE.html).
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-beschriften
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Anwendung der Funktion `annotate()` für einen Text und ein Label. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = weight, y = jump_length,
color = feeding)) +
theme_minimal() +
geom_point(alpha = 0.5) +
annotate("text", x = 10, y = 100, label = "text", size = 5,
fontface = 3) +
annotate("label", x = 10, y = 200, label = "text", size = 5,
fontface = 2)
```
Wir haben mit der `fontface`-Option in `element_text()` die Möglichkeit auch die Schriften anzupassen. Dabei ist es dann die folgende Reihenfolge oder eben der Begriff.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-labels-beschriften-0
#| fig-align: center
#| fig-height: 0.75
#| fig-width: 7
#| fig-cap: "Zuordnung der Zahlen und Begrifflichkeiten in der `fontface`-Option. *[Zum Vergrößern anklicken]*"
df <- data.frame(x = 1:4, fontface = c("plain", "bold", "italic", "bold.italic"))
ggplot(df, aes(x, 1)) +
geom_text(aes(label = fontface, fontface = fontface),
size = 5) +
xlim(0.5, 4.5) +
theme_minimal() +
theme(panel.grid = element_blank(),
axis.text.y = element_blank(),
axis.title = element_blank(),
axis.text.x = element_text(size = 14))
```
Manchmal zeichne ich dann auch gerne mal eine Kurve oder eine Linie ein. Dafür haben wir dann die Funktionen `geom_curve()` und `geom_segment()`. Das sind dann aber schon eher die Besonderheiten in `{ggplot}`. Statt der Funktionen mit `geom` können wir dann auch die Funktionen mit `annotate()` anwählen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-beschriften-mitpfeil
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Darstellung eines Pfeils und einer Linie. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = weight, y = jump_length,
color = feeding)) +
theme_minimal() +
geom_point(alpha = 0.5) +
geom_curve(aes(x = 12, y = 100, xend = 15, yend = 150),
arrow = arrow(length = unit(0.3, "cm"), type = "closed"),
color = "blue",
size = 0.75,
curvature = 0.3) +
geom_segment(aes(x = 5, xend = 25, y = 50, yend = 100), color = "black")
```
Die Beschriftung von Abbildungen ist in dem Buch [ggplot2: Elegant Graphics for Data Analysis (3e)](https://ggplot2-book.org/annotations.html) super beschrieben. Aber auch hier muss man dann immer überlegen, ob sich der Aufwand dann lohnt. Das [R Graphics Cookbook](https://r-graphics.org/CHAPTER-ANNOTATE.html) liefert hier auch nochmal Inspirationen. Manchmal ist der Zeitaufwand aber sehr hoch und du könntest es auch schneller mit PowerPoint lösen. Das mag in einer Abschlussarbeit mal eine gute Idee sein.
::: {layout="[15,85]" layout-valign="top"}
{fig-align="center" width="100%"}
> *"Macht es überhaupt Sinn? Wenn du nur ein paar Abbildungen mit einem Pfeil versehen willst, dann würde ich dir immer den Weg über PowerPoint empfehlen. Dann kannst du dort deine Abbildung exportieren und im Zweifel zuschneiden. Das geht viel schneller als sich hier mit der Annotation in `{ggplot}` zu quälen. Aber ja, es ist sehr befriedigend, wenn es dann geklappt hat..." --- Jochen Kruppa-Scheetz, meiner bescheidener Meinung nach.*
:::
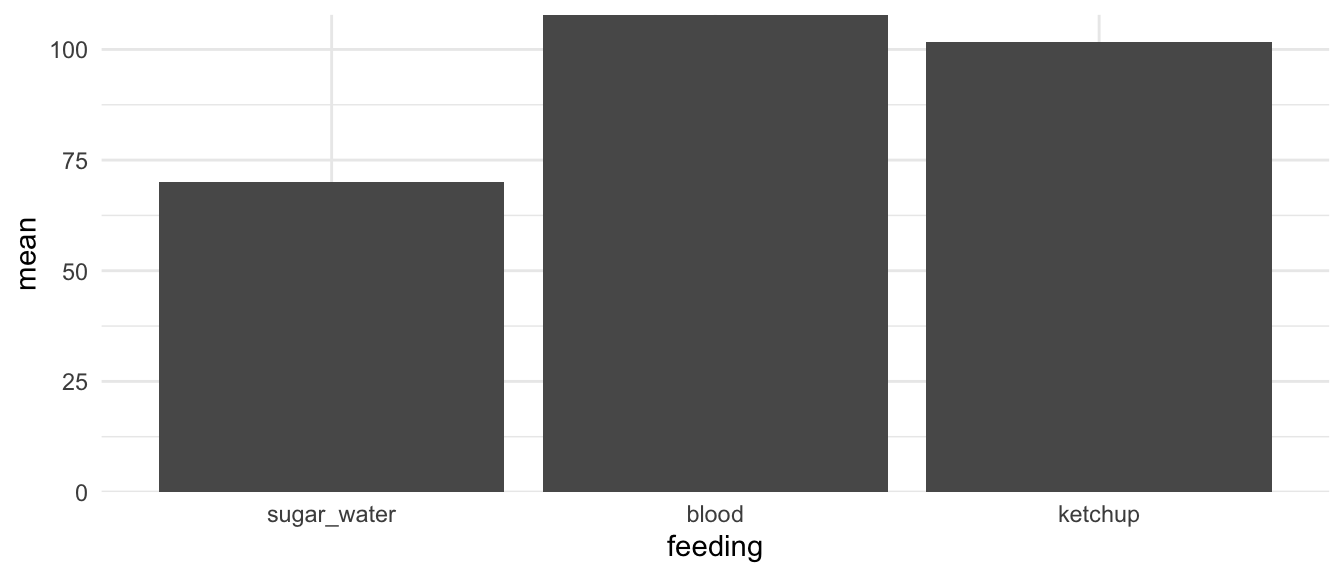
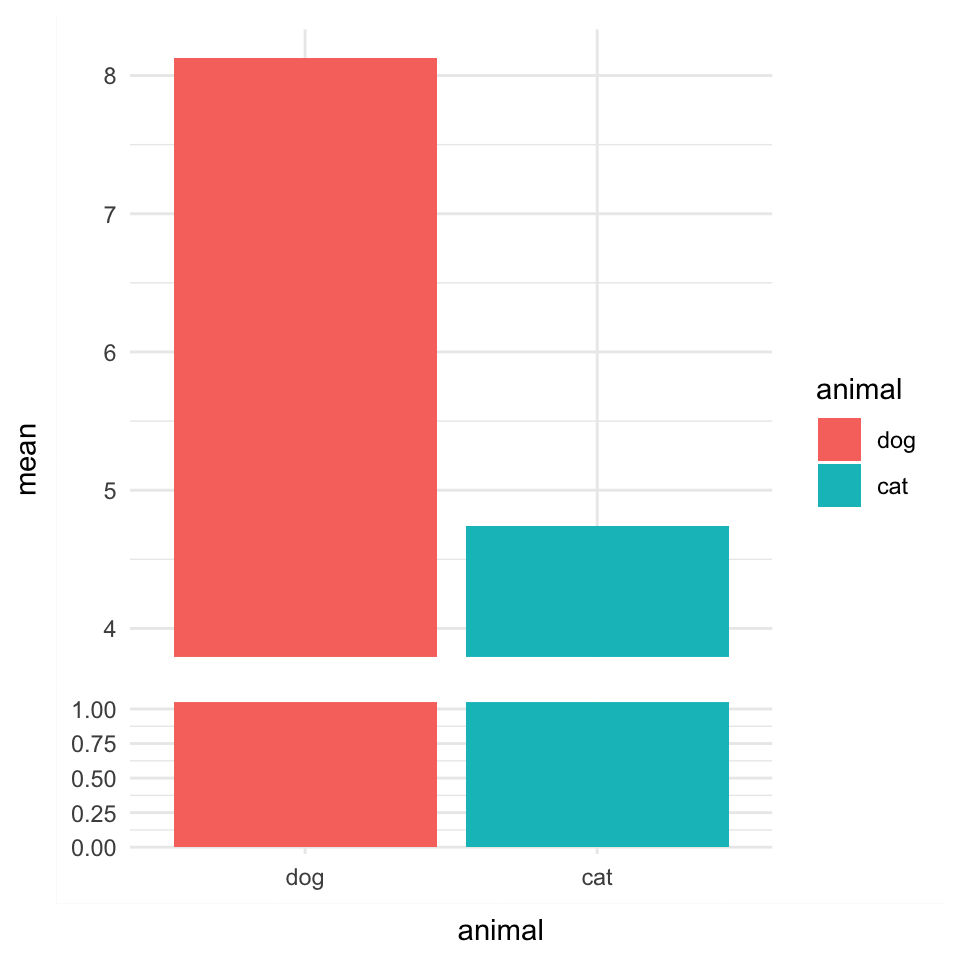
### Keine Lücke auf der y-Achse
Wenn du dir einen Barplot baust, dann hast du das Problem, dass auf der y-Achse eine kliene Lücke zu der x-Achse vorliegt. Das mag unangenehm sein. Deshalb können wir diese Lücke auch entfernen. Zuerst nochmal schnell die Daten zusammengefasst.
```{r}
stats_tbl <- ggplot_tbl |>
group_by(feeding) |>
summarise(mean = mean(jump_length))
```
Dann können wir uns auch schon die Abbildung bauen und dabei dann die Option `expand = expansion(0)` setzen. Damit haben wir dann keine Lücke mehr in unserer Abbildung.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-zero
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafter ohne Lücke auf der y-Achse zu der x-Achse. *[Zum Vergrößern anklicken]*"
ggplot(stats_tbl, aes(x = feeding, y = mean)) +
theme_minimal() +
geom_bar(stat = "identity") +
scale_y_continuous(expand = expansion(0))
```
### Abbildungen nebeneinander {#sec-abbildung-nebeneinander}
Das [R Paket `{patchwork}`](https://patchwork.data-imaginist.com/articles/patchwork.html) erlaubt es mehrere `ggplot` Abbildungen nebeneinander oder in einem beliebigen Layout miteinander zu verbinden. Das tolle ist, dass die Idee sehr intuitiv ist. Wir nutzen wieder das `+` um verschiedene Plots miteinander zu verbinden. Im Folgenden erschaffen wir uns zwei `ggplots` und speichern die Plots in den Objekten `p1` und `p2`. Das ist wie wir es bisher kennen, nur das jetzt keine Abbildung erscheint sondern beide Plots in zwei Objekten gespeichert sind.
```{r}
#| warning: false
#| echo: true
#| message: false
p1 <- ggplot(data = ggplot_tbl,
aes(x = weight, y = jump_length,
color = feeding)) +
geom_point() +
scale_color_okabeito() +
theme_minimal()
p2 <- ggplot(data = ggplot_tbl,
aes(x = feeding, y = jump_length,
color = feeding)) +
geom_point() +
scale_color_okabeito() +
theme_minimal()
```
Wie können wir nun die beiden Abbildungen nebeneinander zeichnen? Wir nutzen einfach das `+` Symbol.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: true
#| label: fig-labels-patchwork-1
#| fig-align: center
#| fig-height: 3
#| fig-width: 9
#| fig-cap: "Beispielhafte Abbildung der zweier Plots nebeneinander für die Sprungweite und zwei Einflussvariablen. **(A)** Zusammenhang zwischen dem Gewicht und der Sprungweite. **(B)** Zusammenhang der Ernährungsart und der Sprungweite. *[Zum Vergrößern anklicken]*"
p1 + p2 +
plot_layout(ncol = 2) +
plot_annotation(tag_levels = 'A', tag_prefix = '(', tag_suffix = ')') &
theme(plot.tag = element_text(size = 16, face = "bold"))
```
Auf der Seite des [R Paket `{patchwork}`](https://patchwork.data-imaginist.com/articles/patchwork.html) findest du viel mehr Möglichkeiten das Layout anzupassen und auch die einzelnen [Subplots zu beschriften](https://patchwork.data-imaginist.com/articles/guides/annotation.html).
### Abbildungen abspeichern
Wenn du eine Abbildung abspeichern willst, dann musst du nur nach dem `ggplot`-Code die Funktion `ggsave()` setzen. Wie du im hier im Folgenden siehst, speichere ich die Abbildung der Boxplots der Hunde- und Katzenflöhe einmal in der Datei `flea_dog_boxplot.png` ab. Dabei wähle ich eine Breite `width` und eine Höhe `height` von jeweils 5. Du musst dann immer etwas spielen, je größer die Zahlen, desto größer die Abbildung und die Auflösung.
```{r}
#| warning: false
#| echo: true
#| eval: false
#| message: false
#| label: fig-save
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abspeicherung einer Abbdilung mit `ggsave()`. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl,
aes(x = feeding, y = jump_length)) +
geom_boxplot()
## Abspeichern des obigen ggplots
ggsave("feeding_boxplot.png", width = 5, height = 5)
```
Wie immer hilft auch die [Hilfeseite von `ggsave()`](https://ggplot2.tidyverse.org/reference/ggsave.html) weiter, wenn es um mehr Optionen und Qualität der Abbildungen geht und natürlich das Kapitel im Buch [ggplot2: Elegant Graphics for Data Analysis (3e)](https://ggplot2-book.org/themes.html#sec-saving) wo wir nochmal über verschiedene Formate wie PDF und PNG aufgeklärt werden.
### Mathematische Ausdrücke {#sec-eda-tex}
Häufig wollen wir nicht nur einfache Achsenbeschriftungen haben, sondern auch irgendwie komplexere Einheiten wie Eisendüngergehalt im Boden in $[kg\, ha]^{-1}$ darstellen. Jetzt soll die Einheit auch in dieser Form mit in die Achsenbeschriftung. Wir können dafür zwei Wege wählen. Einmal über das R Paket `{latex2exp}` und die Funktion `TeX()` oder aber die Funktion `expression()`, wofür wir dann kein eigenes R Paket brauchen. Beide Wege haben Vor- und Nachteile. Wir gehen aber beide mal durch. Mehr Informationen durch das Tutorium [Using latex2exp](https://cran.r-project.org/web/packages/latex2exp/vignettes/using-latex2exp.html) oder aber eben der Klassiker mit [Plot math expression](https://stat.ethz.ch/R-manual/R-patched/library/grDevices/html/plotmath.html).
Wir können die Funktion `expression()` nutzen um uns mathematische Formeln zu bauen. Leider ist das Ganze etwas frickelig und auch ich brauche immer drei Anläufe, bis die Formel dann passt. Im Folgenden aber einmal zwei Beispiel für mathematische Formeln und Ausdrücke. Beachte, dass du jedes Leerzeichen durch eine Tilde `~` abbilden musst. Ich nutze die Funktion `expression()` sehr selten und nur wenn die Formel wirklich sehr einfach ist. Da wir aber schon mit eckigen Klammern Probleme kriegen und diese so nervig mit `"` einklammern müssen, nutze ich dann das Paket `{latex2exp}` was ich im Folgenden vorstellen werde.
Hier aber erstmal zwei Beispiele für eine Formel mit der Funktion `expression()`. Wenn du mehr über die Möglichkeiten wissen willst, dann schauen auch einmal auf die Hilfeseite von [Plot math](https://stat.ethz.ch/R-manual/R-patched/library/grDevices/html/plotmath.html) oder du googelst dir die Lösung wie ich früher zusammen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| fig-align: center
#| fig-height: 2
#| fig-width: 5
plot(expression(Eisendüngeform~und~-höhe~"[kg ha]"^-1), cex = 1.5, main = "")
plot(expression(Fe-Gehalt~"["~mg%.%(kg~TM)^-1~"]"), cex = 1.5, main = "")
```
Für mich ausdrücklich einfacher geht es mit dem R Paket `{latex2exp}` und der Funktion `TeX()` sowie die Helferfunktion `r"()"`. Ja, hier muss man dann noch eine andere Programmiersprache kennen, aber wie immer, du wirst nur schlauer. Die Informationen zur Matheumgebung in $\LaTeX$ kommen dann nochmal extra zwischen zwei Dollarzeichen `$`. Ja, das ist etwas wirr für einen Anfänger, aber wir nutzen hier auch zwei Programmiersprachen zusammen. Zum einen $\LaTeX$ um die Mathesymbole sauber darzustellen und dann R um die Abbildungen in `ggplot()` zu bauen. Mehr Informationen zu der Matheumgebung in $\LaTeX$ findest du einmal in der [LaTeX Mathehilfe I](https://de.wikipedia.org/wiki/Hilfe:TeX) sowie der [LaTeX Mathehilfe II](https://en.wikibooks.org/wiki/LaTeX/Mathematics).
Wie bauen wir uns also unseren mathematischen Ausdruck? Als erstes brauchen wir die Funktion `Tex()`, die sagt einfach nur aus, dass jetzt $\LaTeX$-Code kommt. Dann wollen wir noch einen String brauen in dem der $\LaTeX$-Code für unseren mathematischen Ausdruck drin steht. Diesen String bauen wir mit `r"()"`. Achtung, hier ist das Gänsefüßchen oben und unten vor und nach der runden Klammer sehr wichtig. In den Ausdruck können wir dann Text schreiben `Eisengehalt` oder aber einen mathematischen Ausdruck abgrenzt von zwei Dollarzeichen `$` wie `$[kg\, ha]^{-1}$`. $\LaTeX$ kann nämlich nicht nur mathematische Ausdrücke sondern ist eigentlich ein Textverarbeitungsprogramm. Deshalb musst du hier *nochmal* zwischen Text und mathematischen Ausdruck unterscheiden.
Hier nochmal aufgeschlüsselt wie der Code aussieht. Wir schreiben den Code nachher in einer Zeile, aber zum Verständnis ist es besser, wenn wir den Code einmal aufgeklappt sehen.
```{r}
#| eval: false
TeX(
r"(
Eisengehalt $[kg\, ha]^{-1}$
)"
)
```
Wir wollen uns das Ergebnis einmal in einem simplen `plot()` anschauen. Wir nutzen die Funktionalität natürlich später in `ggplot`, aber hier ist es so einmal einfacher zu sehen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| fig-align: center
#| fig-height: 2.5
#| fig-width: 5
plot(cex = 2, main = "",
TeX(r"(
Eisengehalt $[kg\, ha]^{-1}$
)")
)
```
Auch können wir sehr viel komplexere Formeln erstellen. Beachte auch hier, dass wir zwei Matheumgebungen in $\LaTeX$ vorliegen haben.
```{r}
#| warning: false
#| echo: true
#| message: false
#| fig-align: center
#| fig-height: 2.5
#| fig-width: 5
plot(cex = 2, main = "",
TeX(r"(
A $\LaTeX$ formula: $\frac{2hc^2}{\lambda^5}\frac{1}{e^{\frac{hc}{\lambda k_B T}} - 1}$
)")
)
```
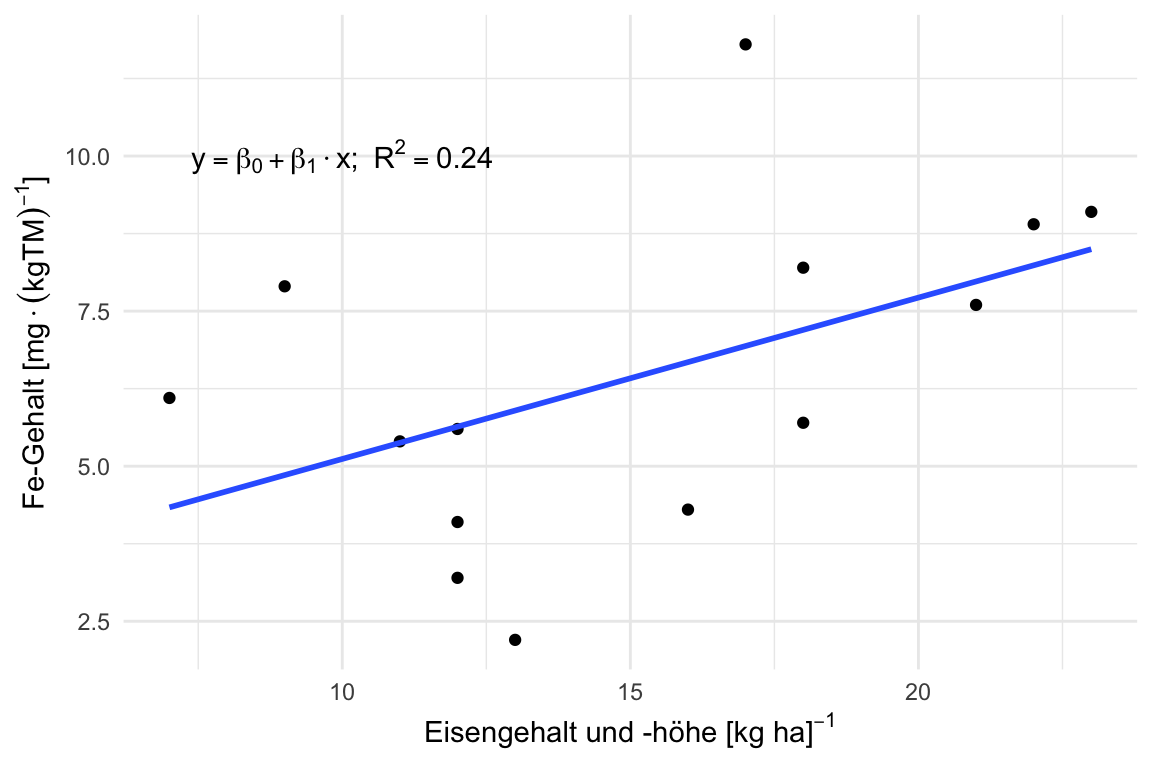
In der @fig-scatter-flea-1 dann nochmal die Anwendung in einem `ggplot` in dem wir die Achsen entsprechend beschriften und dann auch noch eine ausgedachte Regressionsgeleichung zu der Abbildung ergänzen.
```{r}
#| echo: true
#| warning: false
#| message: false
#| label: fig-scatter-flea-1
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Zusammenhang zwischen dem Eisengehalt und -höhe im Boden und dem Eisengehalt in Salat. Zusätzlich ergänzt eine Regressiongleichung und ein ausgedachtes Bestimmtheitsmaß. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = weight, y = jump_length)) +
geom_point() +
stat_smooth(method = "lm", se = FALSE) +
theme_minimal() +
labs(x = TeX(r"(Eisengehalt und -höhe $[kg\, ha]^{-1}$)"),
y = TeX(r"(Fe-Gehalt $[mg \cdot (kg TM)^{-1}]$)")) +
annotate("text", x = 10, y = 200,
label = TeX(r"($y = \beta_0 + \beta_1 \cdot x;\; R^2 = 0.24$)"))
```
Wenn du dann mal die Funktion `Tex()` in `geom_text()` verwenden willst, dann musst du einmal etwas anpassen. Dann klappt es aber auch hier. Das hat mich mal echt Nerven und Zeit gekostet, deshalb lagere ich die Information mal hier für mich.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: false
#| fig-align: center
#| fig-height: 1
#| fig-width: 2
ggplot() +
theme_void() +
geom_text(aes(0, 0,
label = TeX(r'($\alpha x^\alpha$, where $\alpha \in 1\ldots 5$)',
output = "character")), parse = TRUE)
```
### Farbpaletten
Die [Farben](http://www.cookbook-r.com/Graphs/Colors_(ggplot2)/) können weitreichend angepasst werden. Weiter unten findest du dann noch verschiedene Farbpaletten. Wenn du deine Farben selber wählst, dann kann es manchmal etwas kunterbund aussehen. Neben den klassischen Farben im R Paket `{ggplot2}` gibt es noch weit, weit mehr Farbpaletten. Wir nutzen in der Folge immer wieder die Okabe-Ito Farbpalette aus dem R Paket `{see}`. Die Okabe-Ito Farbpalette ist speziell so gebaut, dass die Farben sich gut für farbenblinde Personen unterscheiden. Mehr zum R Paket `{see}` auf der [Hilfeseite des Paketes](https://easystats.github.io/see/index.html). Der Kontrast zwischen den Farben ist sehr gut. Wenn du eine andere Farbpalette nutzen willst, findest du hier noch andere [Color Scales](https://easystats.github.io/see/articles/seecolorscales.html).
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-see-0
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung der Okabe-Ito Farbpalette für Boxplots. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl,
aes(x = feeding, y = jump_length,
fill = feeding)) +
geom_boxplot() +
scale_fill_okabeito() +
theme_minimal()
```
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-see-1
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung der Okabe-Ito Farbpalette für Punkte. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl,
aes(x = feeding, y = jump_length,
fill = feeding)) +
geom_point() +
scale_color_okabeito() +
theme_minimal()
```
Das Ganze geht dann auch händisch mit dem folgenden Code für die [jeweiligen Farben](http://www.cookbook-r.com/Graphs/Colors_(ggplot2)/#a-colorblind-friendly-palette). Anbei einmal die Farbpalette dargestellt.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-ggplot-okabeito
#| fig-align: center
#| fig-height: 0.75
#| fig-width: 6
#| fig-cap: "Farbpalette nach dem Okabe-Ito-Schema ebenfalls für farbblinde Personen erstellt. *[Zum Vergrößern anklicken]*"
df <- data.frame(
x = 1:9,
y = 0.5,
z = factor(1:9)
)
ggplot(df, aes(x, y)) +
geom_tile(aes(fill = z), colour = "grey50") +
scale_fill_okabeito() +
theme_void() +
theme(legend.position = "none")
```
Die Farben sind dann in der Reihenfolge wie folgt kodiert.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: false
cbbPalette <- c("#E69F00", "#56B4E9", "#009E73",
"#F0E442", "#0072B2", "#D55E00",
"#CC79A7", "#999999", "#000000")
```
Wenn wir Boxplots einfärben wollen dann nehmen wir den folgenden Code.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: false
scale_fill_manual(values = cbPalette)
```
Und das hier ist die Ergänzung für Punkte und Linien.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: false
scale_colour_manual(values = cbPalette)
```
Neben der Okabe-Ito Farbpalette liefert das [R Paket `{duke}`](https://cran.r-project.org/web/packages/duke/vignettes/duke.html) noch eine andere Möglichkeit eine Farbpalette für Farbblinde zu generieren.
```{r}
#| warning: false
#| echo: false
#| message: false
#| label: fig-ggplot-duke
#| fig-align: center
#| fig-height: 0.75
#| fig-width: 6
#| fig-cap: "Farbpalette nach dem Duke-Schema ebenfalls für farbblinde Personen erstellt. *[Zum Vergrößern anklicken]*"
df <- data.frame(
x = 1:9,
y = 0.5,
z = factor(1:9)
)
ggplot(df, aes(x, y)) +
geom_tile(aes(fill = z), colour = "grey50") +
scale_duke_fill_discrete() +
theme_void() +
theme(legend.position = "none")
```
Die Farben sind dann in der Reihenfolge wie folgt kodiert.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: false
dukePalette <- c("#012169", "#C84E00", "#00539B", "#339898",
"#A1B70D", "#E89923", "#FFD960", "#262626")
```
Die Funktionen hier sind `scale_duke_color_discrete()` sowie `scale_duke_continuous()` und `scale_duke_fill_discrete()`.
Manchmal benötigen wir auch Farbverläufe. In R heißen diese Farbverläufe dann Farbpaletten. Eine Einführung liefert das Tutorium [Using RColorBrewer palettes](http://www.sthda.com/english/wiki/colors-in-r#using-rcolorbrewer-palettes). Ich selber nutze gerne das R Paket `{wesanderson}` welches sehr schöne Farbverläufe hat. Mehr kannst du auf der GitHub Seite [Wes Anderson Palettes](https://github.com/karthik/wesanderson) erfahren. Wir können die Paletten ganz einfach mit der Funktion `wes_palette()` laden.
```{r}
#| fig-height: 0.75
#| fig-width: 6
wes_palette("Zissou1")
```
Das schöne ist hier, dass wir einfach wie folgt Farbverläufe erstellen können. Wir wollen hier 21 Farbwerte haben und das Ganze dann als kontinuierlichen Verlauf.
```{r}
#| fig-height: 0.75
#| fig-width: 6
wes_palette("Zissou1", 21, type = "continuous")
```
### Lange Namen auf der x-Achse
Manchmal haben wir das Problem, dass unsere Namen der Level der Faktoren zu lang werden oder wir zu viele Namen eben abbilden wollen. Der Blogpost [Quick and easy ways to deal with long labels in `{ggplot}`](https://www.andrewheiss.com/blog/2022/06/23/long-labels-ggplot/) hilft hier einmal. Ich habe mich dann meistens für die folgende Lösung entschieden.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-discrete-longnames
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit gestapelten Gruppen auf der x-Achse. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
scale_x_discrete(labels = c("Blutkonserven aus Rinderblut",
"Ketchup aus einer Flasche",
"Zuckerwasser mit Fruktose"),
guide = guide_axis(n.dodge = 2))
```
Das Rotieren um 45° mag auch eine Lösung sein, aber ich fand es dann immer schwerer zu lesen. Wenn es aber wirklich viele Gruppen sind, dann mag es dann doch eine der besseren Lösungen sein. Du kannst ja dann noch den Winkel entsprechend anpassen. Ich muss immer mit `hjust` spielen, bis es dann gut passt.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-discrete-longnames-rotate
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit einer 45° Drehung der Gruppen auf der x-Achse. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
scale_x_discrete(labels = c("Blutkonserven aus Rinderblut",
"Ketchup aus einer Flasche",
"Zuckerwasser mit Fruktose")) +
theme(axis.text.x = element_text(angle = -45, hjust = 0))
```
Am Ende sei dann noch auf die Funktion `label_wrap()` aus dem Paket `{scales}` verwiesen, die dir dann einen automatischen Zeilenumbruch mit einbaut. Da musst du dann spielen, ob du wirklich einen Zeilenbruch vertreten kannst oder nicht. Manchmal willst du dann die Namen zusammenhaben und nicht auseinandergerissen.
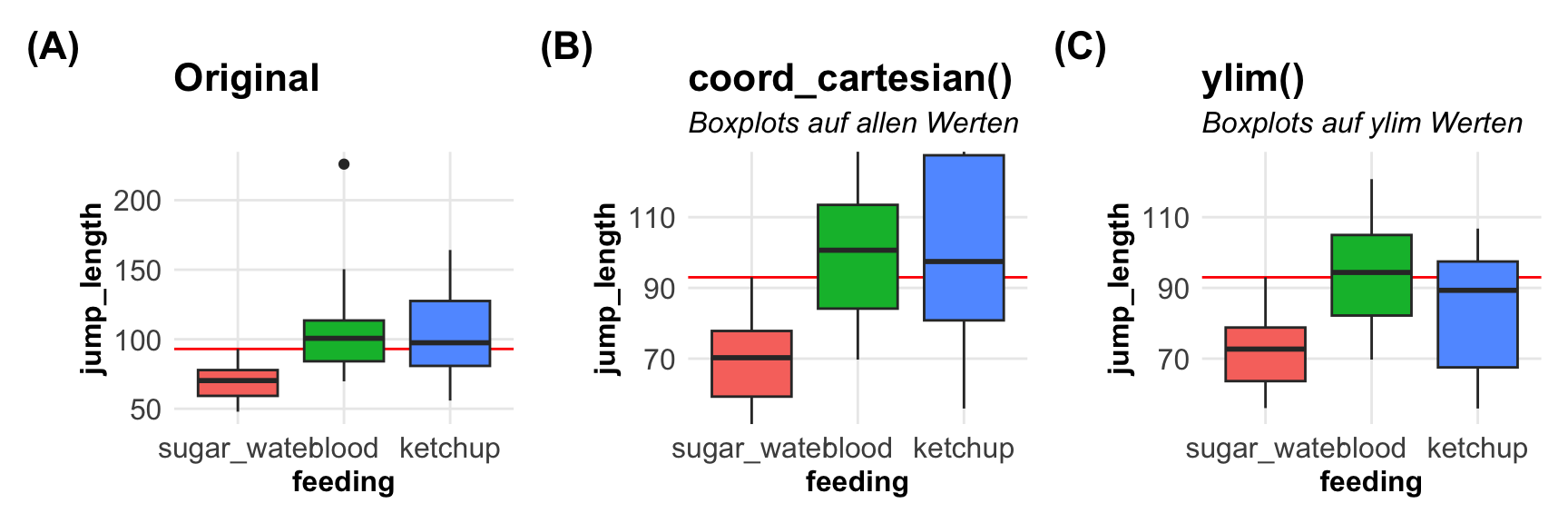
### Zoomen
Mir ist dann noch vor kurzem über den Weg gelaufen, dass wenn wir `ylim()` nutzen um in eine Abbildung zu zoomen, wir automatisch auch die Abbildungen wie einen Boxplot verändern. Wir entfernen mit `ylim()` wirklich die Werte aus der Berechnung der Abbildung. Wenn wir Zoomen wollen, also wirklich die Abbildungen auf allen Werten aber näher dran, dann müssen wir die Funktion `coord_cartesian()` nutzen. In dem folgenden Beispiel siehst du einmal die Anwendung. Dazu dann noch mein eigenes Theme mit `theme_modeling()` von weiter oben sowie die Anwendung des R Pakets `{patchwork}` um die Abbildungen nebeneinander darzustellen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-zoom
#| fig-align: center
#| fig-height: 3
#| fig-width: 9
#| fig-cap: "Problematisches Verhalten der Option `ylim()` beim Zoomen in eine Abbildung. Besser hierfür die Funktion `coord_cartesian()` verwenden. *[Zum Vergrößern anklicken]*"
p1 <- ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_modeling() +
geom_hline(yintercept = 93, colour = "red") +
geom_boxplot() +
theme(legend.position = "none")
p1 + ggtitle("Original") +
p1 + coord_cartesian(ylim = c(55, 125)) +
ggtitle("coord_cartesian()", subtitle = "Boxplots auf allen Werten") +
p1 + ylim(55, 125) +
ggtitle("ylim()", subtitle = "Boxplots auf ylim Werten") +
plot_layout(ncol = 3) +
plot_annotation(tag_levels = 'A', tag_prefix = '(', tag_suffix = ')') &
theme(plot.tag = element_text(size = 16, face = "bold"))
```
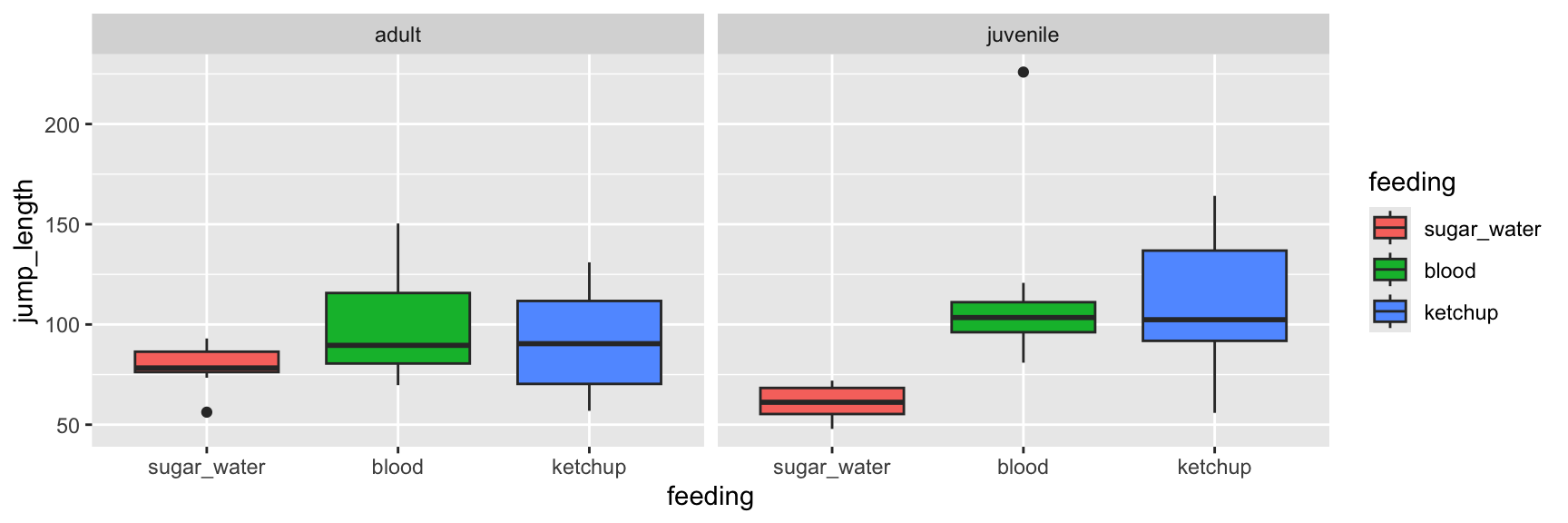
### Abbildungen aufteilen
Die Funktion `facet_wrap()` erlaubt es dir nach einem Faktor die Abbildungen einmal aufzuteilen. Wir werden später noch das R Paket `{ggh4x}` kennenlernen, welches noch besser mit der Aufteilung klarkommt. Hier aber einmal das simpelste Beispiel. Wir wollen die Ernährungsformen einmal getrennt für die beiden Entwicklungsstände darstellen. Wir haben auch hier die Möglichkeit festzulegen, dass die Einheiten auf der x- und y-Achse eben auch frei sein können. Dazu nutzen wir die Option `scales = "free_x"` oder `scales = "free_y"`. Ich persönlich nutze die Option hier nur bei der explorativen Datenanalyse. Wenn ich dann meine Abbildungen habe, würde ich eher über `{patchwork}` mit die einzelnen Abbildungen zusammenkleben als über die Funktion `facet_wrap()`. Aber das ist dann Geschmackssache.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-facet-wrap
#| fig-align: center
#| fig-height: 3
#| fig-width: 9
#| fig-cap: "Aufteilung der Ernährungsformen nach den beiden Entwicklungsständen. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
geom_boxplot() +
facet_wrap(~ stage)
```
### Gebrochene $y$-Achse
Das [R Paket `{ggbreak}`](https://cran.r-project.org/web/packages/ggbreak/vignettes/ggbreak.html) erlaubt es dir in die $x$-Achse oder aber $y$-Achse Lücken einzusetzen oder aber die Achsen eben gebrochen darzustellen. Zur Demonstration bauen wir uns nochmal den `stat_tbl` für die Hunde- und Katzenflöhe. Wir berechnen hier dann die Mittelwerte und nicht mehr die Standardabweichung, da es sonst nicht so gut mit der Darstellung mit der gebrochenen $y$-Achse für dieses Beispiel klappt.
```{r}
stat_tbl <- ggplot_tbl |>
group_by(feeding) |>
summarise(mean = mean(jump_length))
```
In der @fig-labels-ggbreak-1 siehst du einmal die Abbildung der Mittelwerte der Sprungweiten der Hunde- und Katzenflöhe als Barplots dargestellt. Ich habe hier einen Bruch auf der $y$-Achse mit der Funktion `scale_y_break()` bei 1 bis 4 eingefügt und den Abstand über die Option `space` etwas visuell vergrößert. Mit der Option `scales` könntest du dann noch die Skalierung der gebrochenen $y$-Achse anpassen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: true
#| label: fig-labels-ggbreak-1
#| fig-align: center
#| fig-height: 4
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung der Barplots mit gebrochener $y$-Achse. Die Fehlerbalken wurden aus Gründen der besseren Darstellung der zerbrochenen $y$-Achse entfernt. *[Zum Vergrößern anklicken]*"
ggplot(stat_tbl, aes(x = feeding, y = mean, fill = feeding)) +
theme_minimal() +
geom_bar(stat = "identity") +
scale_y_break(c(10, 60), space = 0.5)
```
## R Pakete von Interesse
Manchmal braucht es mehr, als nur `{ggplot}` zu bieten hat. Deshalb gibt es eine riesige Auswahl an zusätzlichen R Paketen, die sich nur mit der Erstellung von Abbildungen im Universum von `{ggplot}` beschäftigen. Hier will ich nicht alle vorstellen, dass macht auch überhaupt keinen Sinn. Es gibt aber immer wieder R Pakete, die finde ich so nützlich, dass ich diese Pakete gerne wiederfinden würde. Meistens brauche ich dann die Pakete aber doch nicht jeden Tag oder häufig. Dann vergesse ich eventuell den Namen oder gar das Paket. Daher ist diese Sammlung also auch für mich eine Art Bibliothek wo ich immer mal schauen kann was es so gibt. Wenn ich ein neues R Paket entdecke, dann ergänze ich das Paket mal eben hier.
::: callout-tip
## Woher habe ich alle Pakete her?
Wenn es einen Link zu einer Seite gibt, den ich immer mal wieder Besuche, ist es der folgende Link: [ggplot2 extensions](https://exts.ggplot2.tidyverse.org/gallery/). Du erfährst hier eine Menge neues an Paketen und es gibt immer wieder spannendes zu entdecken. Ein Besuch lohnt sich auf jeden Fall.
:::
Gehen wir also einmal die sicherlich nie vollständige Liste an R Paketen einmal durch. Die Pakete haben keine richtige Ordnung. Du musst schauen, ob dich was anspricht. Häufig musst du dann auch nochmal die Hilfeseiten hinter den Links besuchen. Hier gebe ich nur einen Einblick für die Anwendungen die ich eben gebraucht habe und glaube wieder zu brauchen.
### `{forcats}`
Das erste Paket hier hat eigentlich gar nicht so richtig was mit `{ggplot}` zu tun. Mit dem [R Paket `{forcats}`](https://forcats.tidyverse.org/) haben wir eigentlich nur eine bessere Art mit Faktoren umzugehen. Das brauchen wir dann auch für faktorielle Experimente, aber auch bei `{ggplot}` lohnt sich eine saubere Definition der kategorialen Variablen. Wir können mit der Funktion `fct()` sauber Faktoren erstellen und haben eine weitreichende Anwendungsmöglichkeiten. Hier empfehle ich dann das [Cheat sheet `{forcats}`](https://raw.githubusercontent.com/rstudio/cheatsheets/main/factors.pdf) um einemla einen Überblick zu erhalten. Hier einmal alle Funktionen durchzugehen sprengt dann das Kapitel. Ich nutze das R Paket immer mal wieder, aber hier hat es auf jeden Fall auch seine Nützlichkeit.
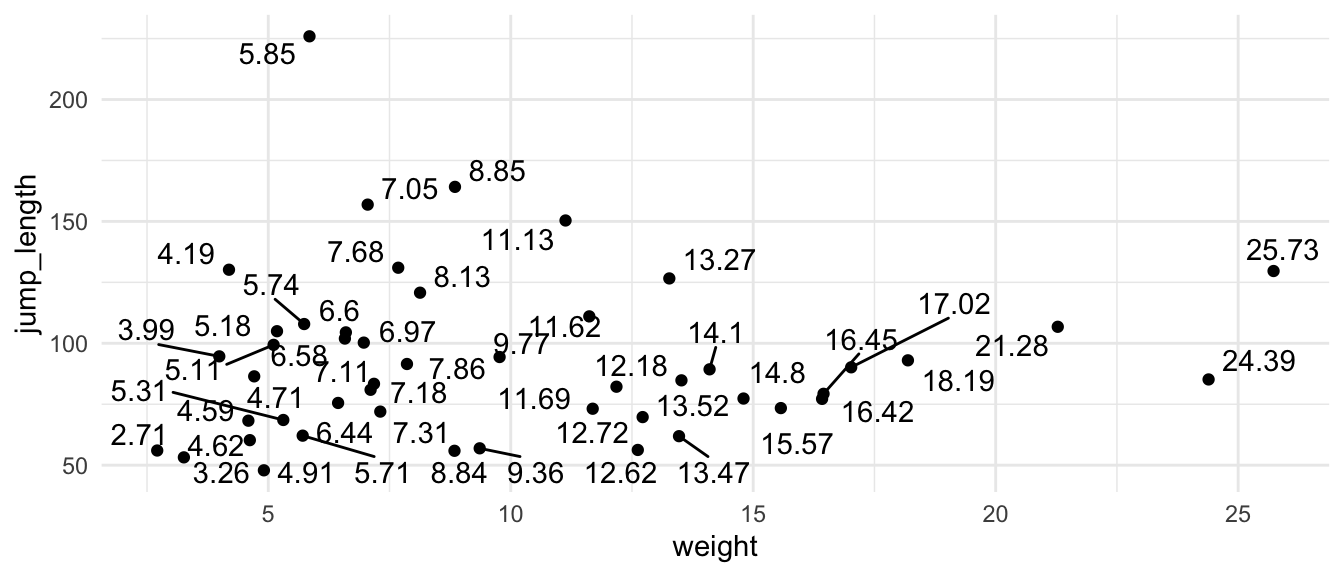
### `{ggrepel}`
Das [R Paket `{ggrepel}`](https://cran.r-project.org/web/packages/ggrepel/vignettes/ggrepel.html) ist die Antwort auf die Frage, ob es nicht irgendwie eine Möglichkeit gibt intelliegent einmal die Labels in einem Plot anzuordnen. Hier dann wiederum nur ein Beispiel, es gibt aber eine Menge [Beispiele für `{ggrepel}`](https://ggrepel.slowkow.com/articles/examples.html) zum Nachbauen und sich inspieren lassen. Die Idee ist eben, dass die LAbels so angeordnet werden, dass wir kein Überlappen der Worte haben und wenn dann doch, wird eben ein Pfeil oder Strich gezeichnet. Das ist super praktisch, wenn wir mal wirklich viele Punkte aufeinmal beschriften wollen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: true
#| label: fig-labels-ggrepel
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Anwendung des R Paketes `{ggrepel}` für die Beschriftung aller Punkte mit den entsprechenden Flohgewichten. *[Zum Vergrößern anklicken]*"
ggplot(ggplot_tbl, aes(x = weight, y = jump_length,
label = weight)) +
theme_minimal() +
geom_point() +
geom_text_repel()
```
### `{ggh4x}`
Das [R Paket `{ggh4x}`](https://teunbrand.github.io/ggh4x/articles/ggh4x.html) liefert nochmal eine Reihe von Funktionen in `{ggplot}` Sachen schöner zu machen. Der zentrale Teil ist aber `facet_wrap2()`. Mit dieser Funktion können wir dann die Aufteilung viel besser kontrollieren und somit auch schöner einen Faktor getrennt über einen anderen Faktor darstellen. Schaue dir dazu gerne einmal die ausführliche Vignette des Paketes an. Hier zeige ich dann nur einmal ein kurzes Beispiel zur Veranschaulichung.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-facet-wrap-gghx
#| fig-align: center
#| fig-height: 3
#| fig-width: 9
#| fig-cap: "Aufteilung der Ernährungsformen nach den beiden Entwicklungsständen. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = stage, y = jump_length,
fill = stage)) +
theme_minimal() +
geom_boxplot() +
facet_wrap2(~ feeding, axes = "all")
```
### `{ggtext}`
Was super nervt ist immer, wenn wir uns mit der Textformatierung rumschlagen müssen. Ich habe ja schon oben bei den Schriftarten geschrieben, dass es noch Lösungen gibt. Das [R Paket `{ggtext}`](https://wilkelab.org/ggtext/) ist mit der Funktion `element_markdown()` und den [Markdown theme elements](https://wilkelab.org/ggtext/articles/theme_elements.html) ein Paket davon. Es gibt aber noch weitere Funktionen und Möglichkeiten in dem R Paket, wie du in der Vignette [Plotting with markdown text](https://wilkelab.org/ggtext/articles/plotting_text.html) dir anschauen kannst. Im Prinzip ist das R Paket `{ggtext}` nochmal eine Möglichkeit alles was in Text in deiner Abbildung vokommt nochmal schhöner zu gestalten. Es gilt wie immer, das ist einigen zu viel udn auch nicht nötig. Manchmal möchtest du es dann aber wissen, dann geht es hier in den Kaninchenbau der Textformatierung. Du kannst mit den klassischen [Markdownbefehlen](https://quarto.org/docs/authoring/markdown-basics.html) deinen text in einer `{ggplot}` Abbildung formatieren. Ganz wichtig, du musst immer über `theme()` dann den entsprechenden Teil in deiner Abbdilung auf `element_markdown()` setzen.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-title-ggtext-0
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit Titel und geänderter Achsenbeschrittung mit Markdown. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
labs(x = "**Ernährungsarten**", y = "*Sprungweite in [cm]*") +
theme(legend.position = "none",
axis.title = element_markdown())
```
Neben klassischen Markdown kannst du auch [HTML Elemente](https://wiki.selfhtml.org/wiki/HTML/Elemente) verwenden. Das macht die Abbildiungen dann noch komplexer und noch kleinteiliger. Hier kannst du dann einzelne Worte oder aber Teile deiner Texte in den Abbildungen ändern. In der folgenden Tabelle dann einmal ein paar Beispiele für HTML Elemente, die du in deiner Abbildung nutzen kannst.
| HTML Element | Darstellung |
|----|----|
| `x<sup>2</sup>` | $x^2$ |
| `x<sub>2</sub>` | $x_2$ |
| `<span style = 'color:#0072B2;'> text </span>` | $\textcolor{#0072B2}{text}$ |
| `<br>` | Zeilenumbruch |
: Ausgewählte HTML Codes und deren Darstellung. {#tbl-html-beispiel}
Wenn wir die HTML Elemente dann anwenden wollen, dann müssen wir auch hier daran denken, dass wir die entsprechenden Teile in der Abbildung im `theme()` dann auch auf `element_markdown()` setzen. Wenn du das vergisst, dann ändert sich in den Abbildungen erstma rein gar nichts. Hier siehst du dann auch ganz schön, dass sich so gewisse mathematische Ausdrücke auch in HTML darstellen lassen. Auch hier kommt es dann auf die Komplexität an. Einfache Einheiten lassen sich hier so auf jeden Fall einfacher darstellen.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-title-ggtext-3
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung mit Titel und geänderter Achsenbeschrittung angepasst durch HTML Elemente. *[Zum Vergrößern anklicken]*"
ggplot(data = ggplot_tbl, aes(x = feeding, y = jump_length,
fill = feeding)) +
theme_minimal() +
geom_boxplot() +
labs(
x = "Horse power [E<sup>2</sup>]",
y = "Miles per gallon (MPG)",
title = "<span style = 'font-size:14pt; font-family:Helvetica;'>Transmission type impacts fuel efficiency</span><br>
MPG is higher for <span style = 'color:#0072B2;'>automatic</span>
than for <span style = 'color:#D55E00;'>manual</span> transmissions"
) +
theme(legend.position = "none",
plot.title = element_markdown(size = 11, lineheight = 1.2),
axis.title = element_markdown(size = 11))
```
Dann können wir auch noch die Abbildungen durch ein `facet_wrap()` schöner machen. Hier können wir dann natürlich auch noch das R Paket `{ggh4x}` mit nutzen um dann wirklich alles rauszuholen. Ich finde aber die Möglichkeit einmal die einzelnen Subplots schöner zu beschriften schon super. Am Ende nutze ich diese Option abr eher weniger sondern baue mir dann immer die einzelnen Plots und klebe diese dann über `{patchwork}` zusammen. Das ist dann aber wirklich nur noch eine Geschmacksfrage.
```{r }
#| warning: false
#| echo: true
#| message: false
#| label: fig-labels-scale-title-ggtext-1
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Abbildung für eine `facet_wrap()` Abbildung mit schöneren Subtiteln. *[Zum Vergrößern anklicken]*"
ggplot(ggplot_tbl, aes(x = stage, y = jump_length)) +
theme_minimal() +
geom_boxplot() +
facet_wrap(~ feeding) +
theme(
strip.background = element_blank(),
strip.text = element_textbox(
size = 12,
color = "white", fill = "#5D729D", box.color = "#4A618C",
halign = 0.5, linetype = 1, r = unit(5, "pt"), width = unit(1, "npc"),
padding = margin(2, 0, 1, 0), margin = margin(3, 3, 3, 3)
)
)
```
### `{ggdist}`
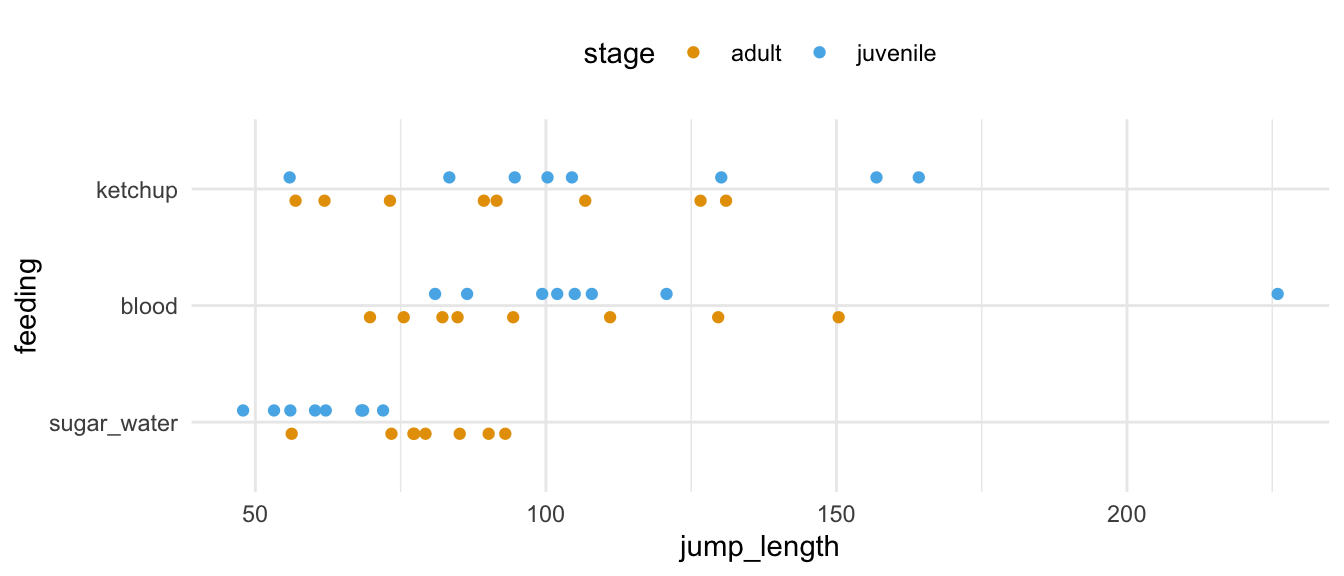
Das [R Paket `{ggdist}`](https://mjskay.github.io/ggdist/index.html) ist ein mächtiges Paket, wenn es darum geht Verteilungen darzustellen. Dafür brauchen wir dann aber auf der anderen Seite auch einiges an Beobachtungen pro Gruppe. Ich würde mal sagen, dass wir hier schnell mal von mittleren zweistelligen Zahlen an Beobachtungen sprechen. Wir können hier aber trotzdem mal zeigen, was das R Paket so kann. Im Folgenden einmal die Verteilung der Sprungweiten auf der x-Achse und die Gruppen auf der y-Achse. Ich habe dann noch die einzlenen Beobachtungen nach dem Entwicklungsstand und der Ernährungsform aufgeteilt.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-ggdist-01
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Darstellung der Sprungweite in Abhängigkeit der Ernährungsform und dem Entwicklungsstand. Hier einmal gedereht für die x- und y-Achse um gleich die Verteilungen etwas besser zeigen zu können. *[Zum Vergrößern anklicken]*"
ggplot_tbl |>
ggplot(aes(x = jump_length, y = feeding, color = stage)) +
theme_minimal() +
geom_point(position = position_dodge(0.4)) +
scale_color_okabeito() +
theme(legend.position = "top")
```
Dann können wir einmal ein lineares Modell schätzen um uns gleich mal die Koeffizienten mit den entsprechenden Fehlern als Verteilung anzuschauen.
```{r}
jump_feeding_fit <- lm(jump_length ~ feeding * stage, data = ggplot_tbl)
```
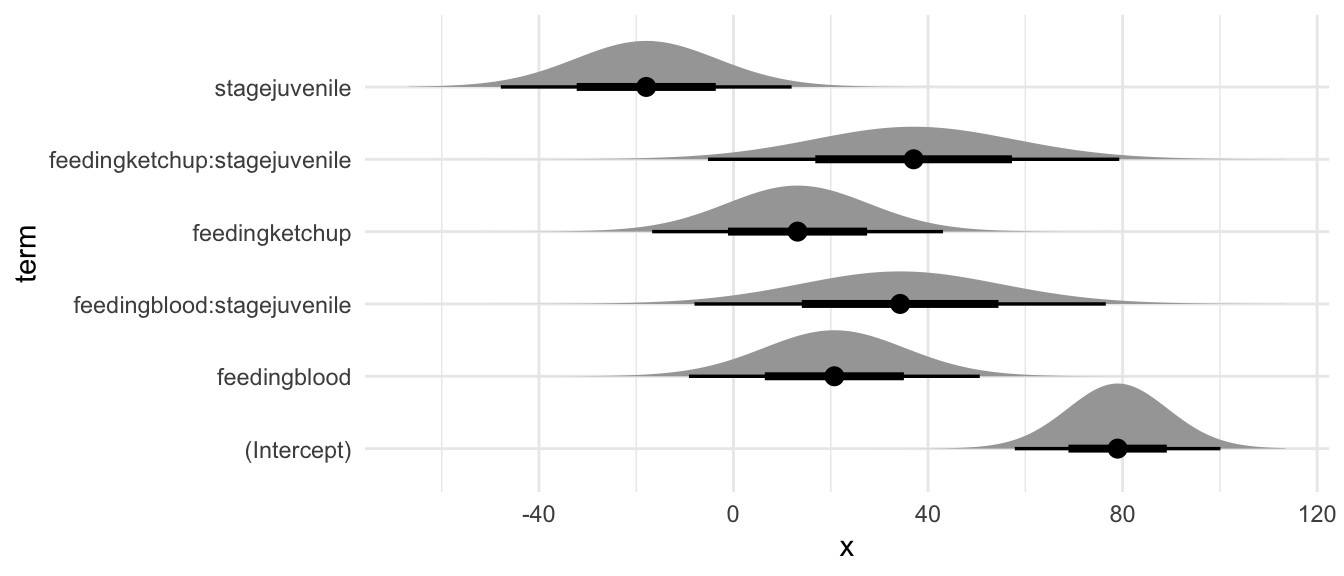
In der folgenden Abbildung siehst du dann einmal die Koeffizienten und deren Fehler als Verteilung einmal dargestellt. Hier siehst du sehr schön, dass die Koeffizienten als Punktschätzer eigentlich aus einer Verteilung stammen, die natürlich auch einen Fehler hat. Der Koeffizient ist eben dann doch nichts anders als der Wert wo die Verteilung ihren Schwerpunkt hat. Wir sehen hier im rinzip die Visualisierung der `summary()` Ausgabe eines linearen Modells.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-ggdist-02
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Darstellung der Koeffizienten aus einer linearen Regression zusammen mit dem Fehler und der Verteilung aus der der Punktschätzer stammt.. *[Zum Vergrößern anklicken]*"
jump_feeding_fit |>
tidy() |>
ggplot(aes(y = term)) +
theme_minimal() +
stat_halfeye(aes(xdist = dist_student_t(df = df.residual(jump_feeding_fit),
mu = estimate, sigma = std.error)))
```
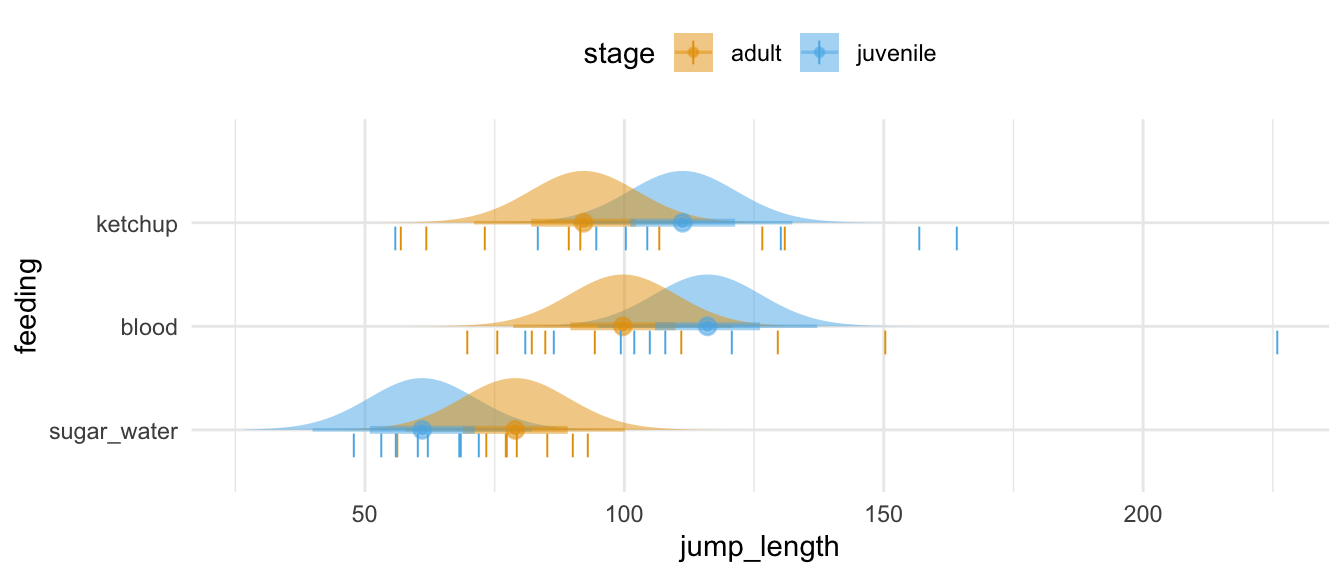
Jetzt können wir nochmal richtig ausholen und dann nochmal alles zusammenbringen. Wir schauen uns die Punktschätzer aus der linearen Regression mit den Koeffizienten an. Dann bringen wir noch die einzelnen Beobachtungen mit ein. Damit haben wir dann die Daten mit den theoretischen Koeffizienten zusammen. Da sehen wir dann auch gleich die Effekte der Ernährungsformen auf die Sprungweite und das sich die Effekte bei einer Zuckerernährung und dem Entwicklungsstand umdrehen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| label: fig-ggdist-03
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Darstellung der Sprungweite in Abhängigkeit von der Ernährungsform der Flöhe und dem Entwicklungstand. Die Verteilung der Koeffizienten ist zusammen mit den beobachteten Daten dargestellt. *[Zum Vergrößern anklicken]*"
expand(ggplot_tbl, feeding, stage) |>
augment(jump_feeding_fit, newdata = _, se_fit = TRUE) |>
ggplot(aes(y = feeding, color = stage)) +
theme_minimal() +
stat_halfeye(aes(xdist = dist_student_t(df = df.residual(jump_feeding_fit),
mu = .fitted, sigma = .se.fit),
fill = stage),
scale = 0.5, alpha = 0.5) +
geom_point(data = ggplot_tbl, aes(x = jump_length),
pch = "|", size = 3, position = position_nudge(y = -.15)) +
scale_color_okabeito() +
scale_fill_okabeito() +
theme(legend.position = "top")
```
Am Ende möchte ich dann nochmal auf das [R Paket `{distributional}`](https://pkg.mitchelloharawild.com/distributional/index.html) verweisen, was erlaubt auch noch andere Verteilungen für die Koeffizienten zu wählen. Häufig reicht hier die t-Verteilung oder Normalverteilung aus, aber es gibt eben dann noch mehr Möglichkeiten. Das Paket ist jedenfalls klasse, wenn es um Verteilungen geht.
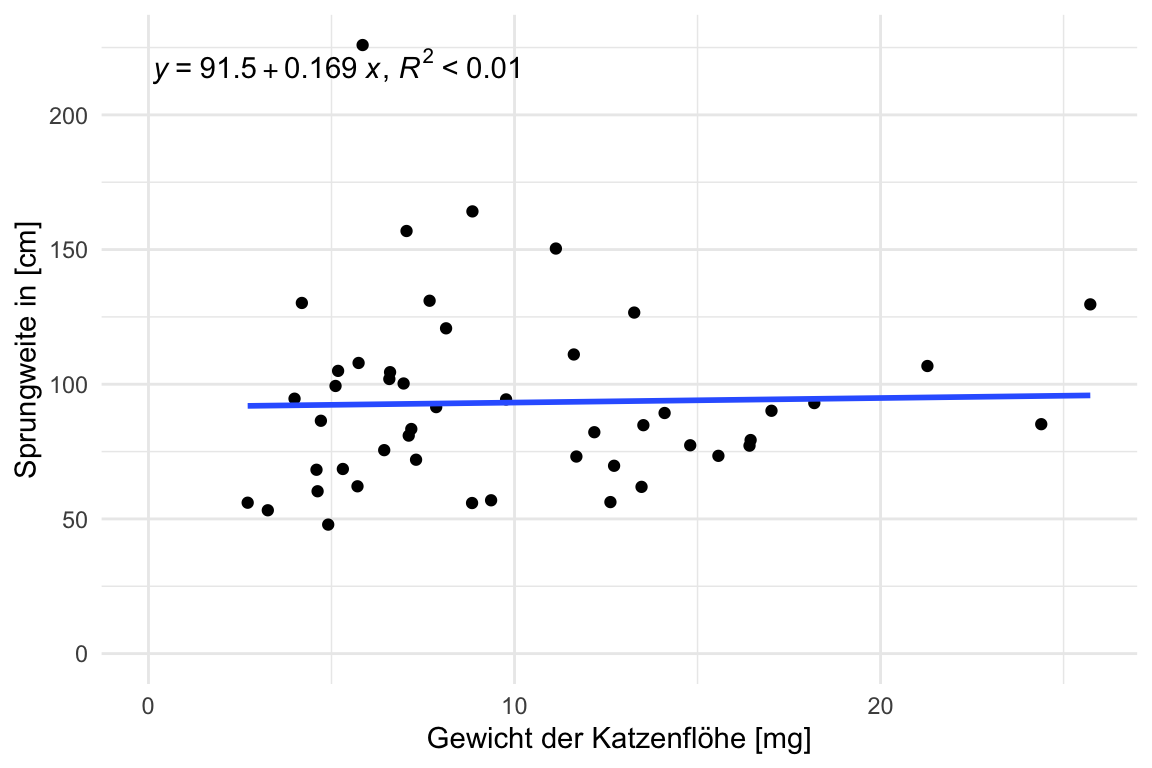
### `{ggpmisc}`
Ich nutze das [R Paket `{ggpmisc}`](https://cran.r-project.org/web/packages/ggpmisc/vignettes/user-guide.html) im Besonderen, wenn ich eine Grade durch eine Punktewolke legen will. Dafür ist das R Paket richtig super. Wir erhalten realtiv einfach die Gradengleichung und auch das Bestimmtheitsmaß angegeben. Wir haben also die Möglichkeit in einer explorativen Datenanalyse einmal zu spielen und dann gleich die Gradengleichung. Das ist jetzt aber ein sehr spezieller Fall. Deshalb hier nur der Link zu dem Paket. Ich zeige die Anwendung nochmal kurz bei dem Scatterplot in dem [Kapitel zur Visualisierung von Daten](#sec-eda-ggplot). Dort ist es auch besser aufgehoben. Sonst verteilt sich das Paket recht gleichmäßig immer mal wieder bei den Regressionsanalysen. Hier einmal ein Beispiel mit den Sprungweiten aus unseren Daten.
```{r }
#| echo: true
#| message: false
#| label: fig-cookbook-ppgmisc
#| fig-align: center
#| fig-height: 4
#| fig-width: 6
#| fig-cap: "Zusammenhang zwischen der Sprungweite in [cm] und Gewicht. Jeder Punkt stellt eine Beobachtung dar. Die Grade wird durch die Punkte gezeichnet und die Gradengleichung zusammen mit dem Bestimmtheitsmaß $R^2$ angegeben."
ggplot(ggplot_tbl, aes(x = weight, y = jump_length)) +
theme_minimal() +
geom_point() +
stat_poly_line(formula = y ~ x, se = FALSE) +
stat_poly_eq(formula = y ~ x, mapping = use_label(c("eq", "r2"))) +
labs(x = "Gewicht der Katzenflöhe [mg]", y = "Sprungweite in [cm]") +
ylim(0, NA) + xlim(0, NA)
```
### `{patchwork}`
Das [R Paket `{patchwork}`](https://patchwork.data-imaginist.com/) bietet die Möglichkeit Abbildungen nebeneinander darzustellen. Hier findest du einmal den Code, den ich hier in diesem Buch nutze. Dafür ist der Abschnitt auch nur da. Damit ich den Code kopieren kann.
```{r}
#| eval: false
p1 + p2 +
plot_layout(ncol = 2) +
plot_annotation(tag_levels = 'A', tag_prefix = '(', tag_suffix = ')') &
theme(plot.tag = element_text(size = 16, face = "bold"))
```
Hier gibt es dann nicht mehr zu sehen. Ausgeführten Code findest du dann weiter oben in dem [Abschnitt zu den Abbildungen nebeneinander](#sec-abbildung-nebeneinander). Das dient hier nur für mich um dann nochmal schnell auf meine Vorlagen zurückgreifen zu können.
### `{gganimate}`
Das [R Paket `{gganimate}`](https://gganimate.com) liefert die Möglichkeit animierte Abbildungen zu erstellen. Es gibt auf der Seite von [Datanovia - How to Create Plots with Beautiful Animation in R](https://www.datanovia.com/en/blog/gganimate-how-to-create-plots-with-beautiful-animation-in-r/) nochmal einen Blogpost dazu und auch das [Cheat sheet `{gganimate}`](https://rstudio.github.io/cheatsheets/gganimate.pdf) hilft weiter. Ich persönlich finde aber die Anwendung immer etwas sperrig. Oder andersherum, ich habe mich noch nicht so richtig in den Code und die Funktionen reinfuchsen können. Nicht jede Funktion klappt auf Anhieb und dann muss man fummeln bis es geht. Anbei einmal eine mögliche Darstellung. Es geht so viel mehr, aber aktuell fehlt mir die Zeit und der Wille mich tiefer mit `{gganimate}` zu beschäftigen.
```{r}
#| warning: false
#| echo: true
#| message: false
#| eval: true
#| label: fig-labels-animate
#| fig-align: center
#| fig-height: 3
#| fig-width: 7
#| fig-cap: "Beispielhafte Anwendung der Funktion `transition_reveal()` auf dem Flohgewicht. *[Zum Vergrößern anklicken]*"
#| cache: true
ggplot(ggplot_tbl, aes(x = weight, y = jump_length,
color = feeding)) +
theme_minimal() +
geom_point(aes(group = seq_along(feeding))) +
theme(legend.position = "top") +
transition_reveal(weight)
```
Am Ende bin ich dann gescheitert hier einmal anständig `geom_smooth()` zu ergänzen, was ja auch wirklich mal das Interessanteste wäre. Das geht auch leider nicht mit dem R Paket `{ggpmisc}` einfach so. Damit ist dann hier erst recht erstmal Pause für mich.
::: {layout="[15,85]" layout-valign="top"}
{fig-align="center" width="100%"}
> *"Das R Paket `{gganimate}` klingt super spannend und ich war auch begeistert es zu nutzen. Dann fand ich aber nicht so richtig eine Anwendung. Vielleicht bei Zeitreihen oder aber bei Verläufen. Aber so richtig nutzen tue ich `{gganimate}` nicht, so dass das Paket hier erstmal parkt, bis ich eine bessere Anwendung gefunden habe. Gut zu wissen das es `{gganimate}` gibt." --- Jochen Kruppa-Scheetz, meiner bescheidener Meinung nach.*
:::
::: callout-tip
## Mehr Pakete von Interesse
Manchmal finde ich irgendwo ein R Paket und denke, dass könnte von Interesse sein und irgendwie dann doch mit in Visualisierung passen. Daher schreibe ich mir das Paket erstmal hier auf und schaue später mal vorbei. Es sind eben dann auch Daten dabei, die man mal visualisieren könnte. Manchmal passen die Pakete hier auch nicht so richtig zur Visualisierung. Aber erstmal sammeln.
| Nr. | Link | Beschreibung |
|----|----|----|
| 1\. | [R Paket `{collapse}`](https://sebkrantz.github.io/collapse/index.html) | Das R Paket `{collapse}` ist ein umfangreiches C/C++-basiertes Paket für die Datenumwandlung und statistische Berechnungen in R. Es ist super spannend, aber mir fehlte bis jetzt die Zeit. Wenn deine Daten riesig werden, dann ist das hier deine Lösung. |
| 2\. | [R Paket `{visibly}`](https://m-clark.github.io/visibly/articles/intro.html) | Das Paket erlaubt dir schnell die passenden Farben für deine Analysen zu finden, die maximal verschieden sind. Daneben gibt es noch weitere Funktionen um Farben und Visualisierungen. |
| 3\. | [R Paket `{gapminder}`](https://github.com/jennybc/gapminder) | Hier noch ein Paket mit Daten von [Gapminder](https://www.gapminder.org/data/) und damit eine Möglichkeit nochmal auf große soziodemographische Daten zuzugreifen. Es ist eien super Quelle und man kann sich in den Daten verlieren. |
| 4\. | [R Paket`{emojifont}`](https://cran.r-project.org/web/packages/emojifont/vignettes/emojifont.html) | Nochmal etwas zu Emojis und die Möglichkeit diese dann in R darzustellen. Nur alleine in `{ggplot}` ist es eine Sache, das ganze dann aber mit Quarto und HTML eine andere. Ich habe es nicht weiter vertieft. |
| 5\. | [R Paket`{emo}`](https://github.com/hadley/emo) | Eine Möglichkeit nochmal anders Emojis darzustellen. Ich hatte das Paket gefunden aber nicht mehr ausprobiert. |
| 6\. | [R Paket `{ggpp}`](https://docs.r4photobiology.info/ggpp/index.html) | Eine weitere Möglichkeit Tabellen und verschiedene Abbildungen ineinander zu verschränken. Also Tabellen innerhalb eines `{ggplot}`s zu plotten oder aber noch wildere Sachen. Am Ende habe ich es nie angewendet. |
| 7\. | [R Paket `{ggsignif}`](https://github.com/const-ae/ggsignif) | Auch hier hast du nochmal die Möglichkeit einen statistischen Test direkt in deine Abbildung darstellen zu lassen. Fand ich interessant, habe es aber dann auch nie selber verwendet. |
: {tbl-colwidths="\[5,35,60\]"}
Diese Liste kann natürlich beliebig erweitert werden. Für mich ist es dann ein ewiger Arbeitsauftrag mich mal mit dieser Liste zu beschäftigen. Teilweise sind aber die Pakete auch hier für dich. Ich fand es interessant, habe das Paket dann aber doch (noch) nicht genutzt.
:::
## Links von Interesse
Kennst du das auch? Spannende Links, die man sich mal anschauen möchte? Aber noch nicht richtig weiß, wie und wo die Links mit reinpassen. Zwar haben alle was mit der Visualisierung zu tun, aber eventuell dann doch zu spezielle Themen. Auch diese Liste ist vermutlich nie vollständig und wird dann immer mal wieder von mir erweitert, wenn ich ein nettes Buch oder einen tollen Blogeintrag zu Thema hier finde.
| Nr. | Link | Beschreibung |
|----|----|----|
| 1\. | [ggplot2: Elegant Graphics for Data Analysis (3e)](https://ggplot2-book.org/) | Das Buch, wenn es um `{ggplot}` geht. Eigentlich ja schon ob mit in der Einleitung, aber dann hier nochmal zur Erinnerung. Hier geht es eher um die Struktur von `{ggplot}` und die Idee dahinter. |
| 2\. | [R Graphics Cookbook](https://r-graphics.org/) | Auch dieses Buch habe ich oben schon mal erwähnt. Es ist super, wenn du nach Rezepten für die Lösung deiner Probleme mit `{ggplot}` suchst. |
| 3\. | [Legend Attributes](https://github.com/tidyverse/ggplot2/wiki/Legend-Attributes) | Zwar wird das `{ggplot}`-Wiki nicht mehr unterstützt, aber dennoch findet sich hier einiges an Informationen zu den Legenden in einem Plot. Sehr schön aufgearbeitet. |
| 4\. | [Legenden in ggplot](https://www.tidyverse.org/blog/2024/02/ggplot2-3-5-0-legends/) | Hier erfahren wir dann nochmal mehr über die Legenden in einem `{ggplot}` und wie wir die Abbildung sauber beschriften. |
| 5\. | [Controlling legend appearance in ggplot2 with `override.aes`](https://aosmith.rbind.io/2020/07/09/ggplot2-override-aes/) | Ein Problem was mir gar nicht so aufgefallen war, wir haben zu viele Informationen auf der Legende oder wollen nicht alles in der Legende darstellen. Hier hilft dieser Blogpost nochmal, wenn du deine Legenden sauberer haben möchtest. |
| 7\. | [Exploratory Data Analysis Checklist](https://bookdown.org/rdpeng/exdata/exploratory-data-analysis-checklist.html) | Manchmal ist es ganz hilfreich sich nochmal klar zu machen wie eine explorative Datenanalyse ablaufen kann. Daher hier nochmal die Checkliste, die du dann abarbeiten kannst. Fand ich als Orientierung ganz hilfreich. |
| 8\. | [Principles of Effective Data Visualization](https://www.cell.com/patterns/fulltext/S2666-3899(20)30189-6) | Eine wunderbare Arbeit, die nochmal auf die Prinzipien einer guten Abbiuldung im Kontext einer wissenschaftlichen Fragestellung eingeht. Ich nutze die Arbeit auch in meiner Lehre und als Orientierung immer super. Besonders wenn du gerade mit der Thematik anfängst. (Hier auch einmal die PDF-Datei unter [Principles of Effective Data Visualization](https://www.cell.com/action/showPdf?pii=S2666-3899%2820%2930189-6)) |
| 9\. | [Emoji Unicode Tables](https://apps.timwhitlock.info/emoji/tables/unicode) | Manchmal wollen wir lustige Emoticons in unsere Abbildung bauen. Dann müssen wir auch wissen, welche es so gibt. Auf dem Handy ist es einfach hier in `{ggplot}` dann manchmal schwerer. Die Tabelle gibt aber erstmal eine Orientierung. |
: {tbl-colwidths="\[5,25,70\]"}
## Referenzen {.unnumbered}