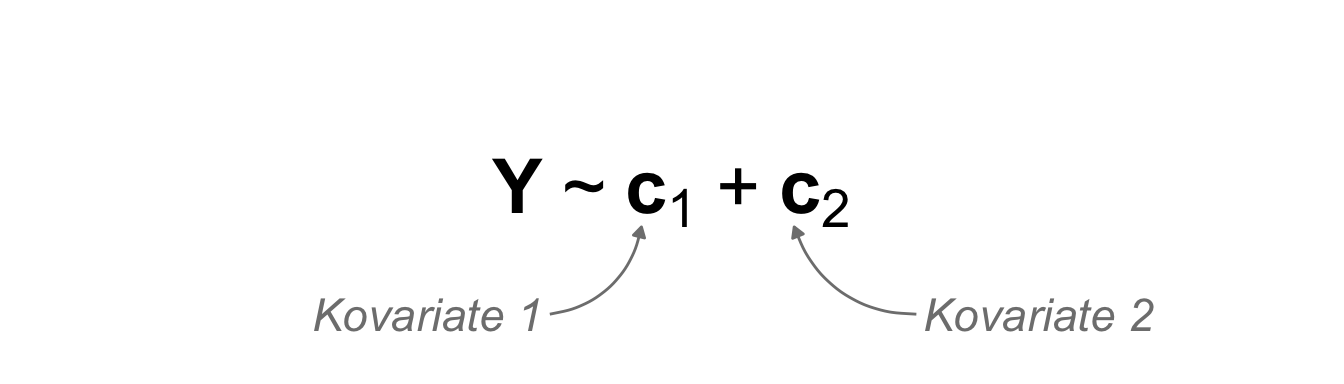21 Generierung von Daten
Letzte Änderung am 24. September 2025 um 13:13:38
“Je größer die Insel des Wissens, desto größer der Strand der Verzweiflung.” — unbekannt
In diesem Kapitel geht es um die Erstellung von zufälligen Daten. Oder anderherum, wir wollen uns künstliche Daten (eng. artificial data oder eng. synthetic data) generieren. Warum machen wir das? Eine Frage, die sich vermutlich kein Statistiker oder Date Scientist stellen würde, ist es doch Teil der wissenschaftlichen Arbeit simulierte Daten zu erstellen. Auf diesen simulierten Daten werden dann die neuen entwickelten Algorithmen getestet. Für Menschen außerhalb der Data Science mag es seltsam erscheinen, dass wir uns einfach Daten erschaffen können. Diese synthetischen Daten kommen dann aber den biologischen Daten manchmal recht Nahe. Daher kannst auch du dir deine Daten theoretisch einfach selber bauen, nachdem du dieses Kapitel gelesen hast.
“With great power comes great responsibility.” — Uncle Ben to Peter Parker in Spiderman
Keine Daten für die Abschlussarbeit erschaffen. Auch wenn die Daten mal nicht so passen, wie du dir das aus einem Experiment erhofft hast, ist es nicht erlaubt sich einfach die Daten selber zu simulieren. Simulierte Daten haben keinen wissenschaftlichen Wert abseits der Validierung von neuen Algorithmen.
Was du aber machen kannst, ist deine experimentellen Daten schon mal im Design vorbauen um deine Analyse in R vorzurechnen. Wenn du dann aus deinem Experiment die echten Daten vorliegen hast, kannst du dann schneller die Analysen rechnen. Du hast dann ja schon alles einmal geübt und vorgearbeitet.
Daher nutzen wir diesen Kapitel auch anders. Wir erschaffen uns ja Daten um Algorithmen zu verstehen. Wenn wir wissen wie die Daten aufgebaut sind, dann können wir auch besser verstehen was uns ein Modell oder ein statistischer Test wiedergibt. Dafür brauchen wir dann die synthetischen Daten. Eine andere Möglichkeit ist, dass du einmal ausprobieren möchtest, wie eine potenzielle Analyse ablaufen würde. Dann macht es auch Sinn einmal über künstliche Daten nachzudenken. Ich komme dann immer mal wieder auf dieses Kapitel zurück, wenn ich in anderen Kapiteln mal künstliche Daten brauche. Wir schauen uns verschiedene Wege künstliche Daten zu erschaffen einmal an und es geht auch in Excel relativ einfach.
21.1 Allgemeiner Hintergrund
“We test our framework both on synthetic data, as the only source of ground truth on which we can objectively assess the performance of our algorithms, as well as on real data to demonstrate its real-world practicability.” — Jackson et al. (2009)
Wenn wir Daten generieren wollen, dann müssen wir auch wissen, was wir genieren wollen. Dafür müssen wir dann verstehen, wovon wir sprechen. Da wir es bei der Datengenerierung auch immer mit statistischen Modellen zu tun haben, müssen wir auch die Fachsprache der statistischen Modellierung nutzen. Dann wollen wir verstehen, was wir eigentlich simulieren. Klar, wir simulieren Daten, aber was brauchen wir dafür an statistischen Maßzahlen? Ich betrachte dann nochmal die Einschränkungen dieses Kapitels, denn wir können natürlich hier nicht alles simulieren, was möglich ist. Faktisch konzentrieren wir uns hier auf häufige Datenstrukturen in den Agrawissenschaften. Dann zeige stelle ich dir nochmal kurz die zwei R Pakete vor, die wir dann hier nutzen können.
Sprachlicher Hintergrund
“In statistics courses taught by statisticians we don’t use independent variable because we use independent on to mean stochastic independence. Instead we say predictor or covariate (either). And, similarly, we don’t say”dependent variable” either. We say response.” — User berf auf r/AskStatistics
Wenn wir uns mit dem statistischen Modellieren beschäftigen wollen, dann brauchen wir auch Worte um über das Thema reden zu können. Statistik wird in vielen Bereichen der Wissenschaft verwendet und in jedem Bereich nennen wir dann auch Dinge anders, die eigentlich gleich sind. Daher werde ich mir es hier herausnehmen und auch die Dinge so benennen, wie ich sie für didaktisch sinnvoll finde. Wir wollen hier was verstehen und lernen, somit brauchen wir auch eine klare Sprache.
“Jeder nennt in der Statistik sein Y und X wie er möchte. Da ich hier nicht nur von Y und X schreiben will, führe ich eben die Worte ein, die ich nutzen will. Damit sind die Worte dann auch richtig, da der Kontext definiert ist. Andere mögen es dann anders machen. Ich mache es eben dann so. Danke.” — Jochen Kruppa-Scheetz, meiner bescheidener Meinung nach.
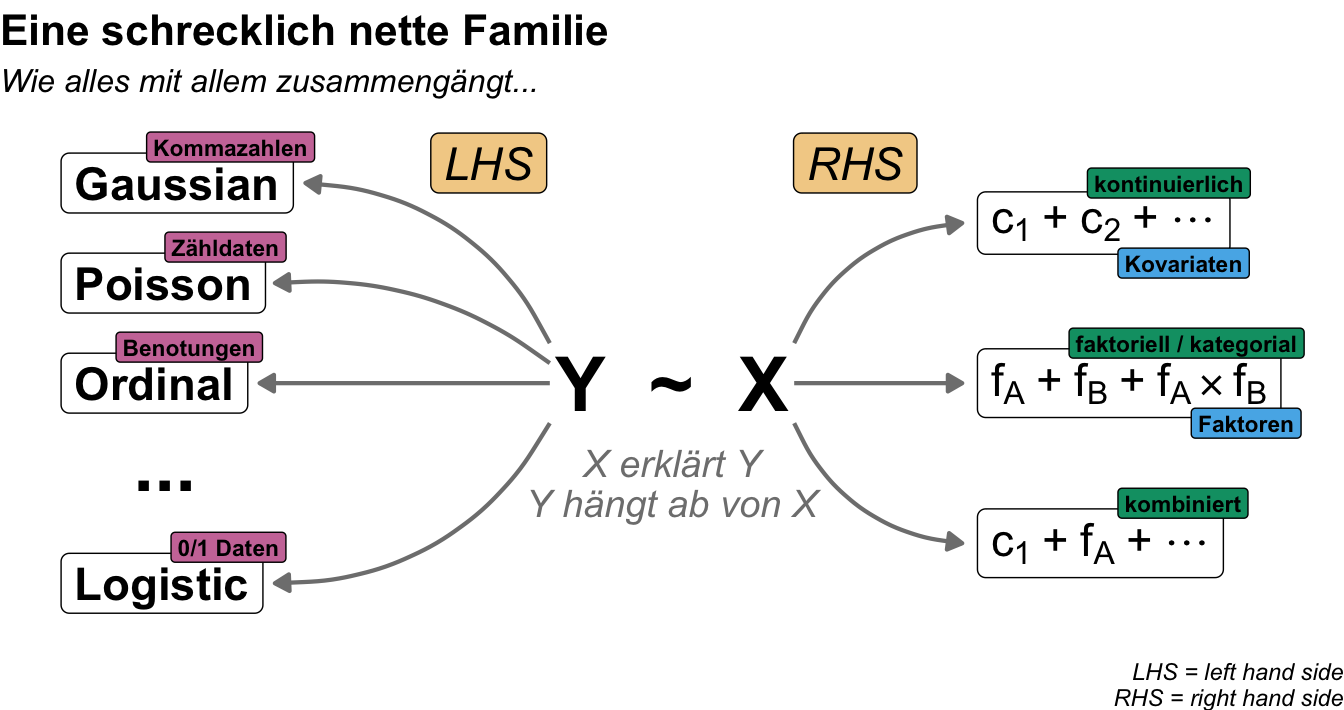
In dem folgenden Kasten erkläre ich nochmal den Gebrauch meiner Begriffe im statistischen Modellieren als Grundlage für die Generierung von Daten. Es ist wichtig, dass wir hier uns klar verstehen. Zum einen ist es angenehmer auch mal ein Wort für ein Symbol zu schreiben. Auf der anderen Seite möchte ich aber auch, dass du dann das Wort richtig einem Konzept im statistischen Modellieren zuordnen kannst. Deshalb einmal hier meine persönliche und didaktische Zusammenstellung meiner Wort im statistischen Modellieren. Du kannst dann immer zu dem Kasten zurückgehen, wenn wir mal ein Wort nicht mehr ganz klar ist. Die fetten Begriffe sind die üblichen in den folgenden Kapiteln. Die anderen Worte werden immer mal wieder in der Literatur genutzt.
“Die Grenzen meiner Sprache bedeuten die Grenzen meiner Welt.” — Ludwig Wittgenstein
Hier kommt einmal die Tabelle mit den wichtigsten Begriffen im statistischen Modellieren und wie ich die Worte benutzen werde. Damit wir uns verstehen und du was lernen kannst. In anderen Büchern und Quellen findest du teilweise die Worte in einem anderen Sinnzusammenhang. Das ist gut so dort. Bei mir ist es anders.
| Symbol | Deutsch | Englisch | |
|---|---|---|---|
| LHS | \(Y\) / \(y\) | Messwert / Endpunkt / Outcome / Abhängige Variable | response / outcome / endpoint / dependent variable |
| RHS | \(X\) / \(x\) | Einflussvariable / Erklärende Variable / Fester Effekt / Unabhängige Variable | risk factor / explanatory / fixed effect / independent variable |
| RHS | \(Z\) / \(z\) | Zufälliger Effekt | random effect |
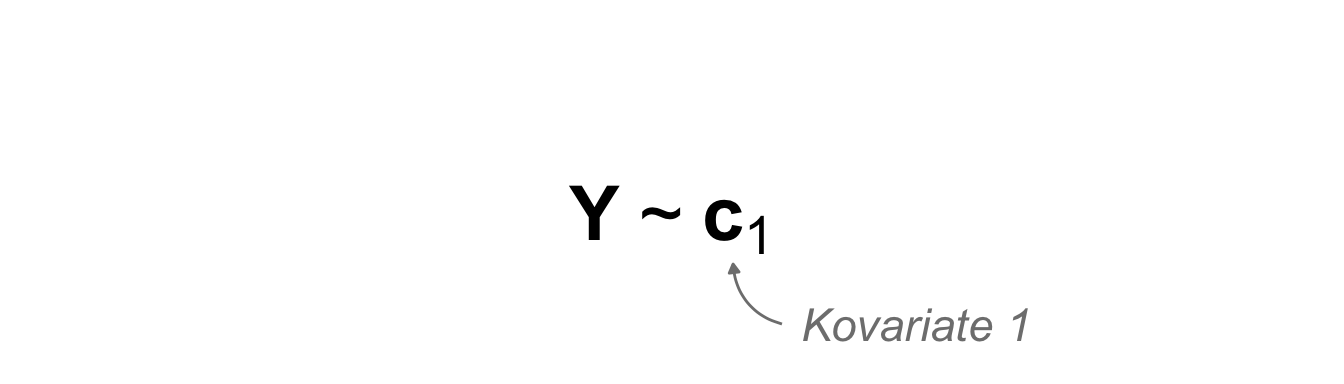
| \(X\) ist kontinuierlich | \(c_1\) | Kovariate 1 | covariate 1 |
| \(X\) ist kategorial | \(f_A\) | Faktor \(A\) mit Level \(A.1\) bis \(A.j\) | factor \(A\) with levels \(A.1\) to \(A.j\) |
Am Ende möchte ich nochmal darauf hinweisen, dass wirklich häufig von der abhängigen Variable (eng. dependent variable) als Messwert und unabhängigen Variablen (eng. independent variable) für die Einflussvariablen gesprochen wird. Aus meiner Erfahrung bringt die Begriffe jeder ununterbrochen durcheinander. Deshalb einfach nicht diese Worte nutzen.
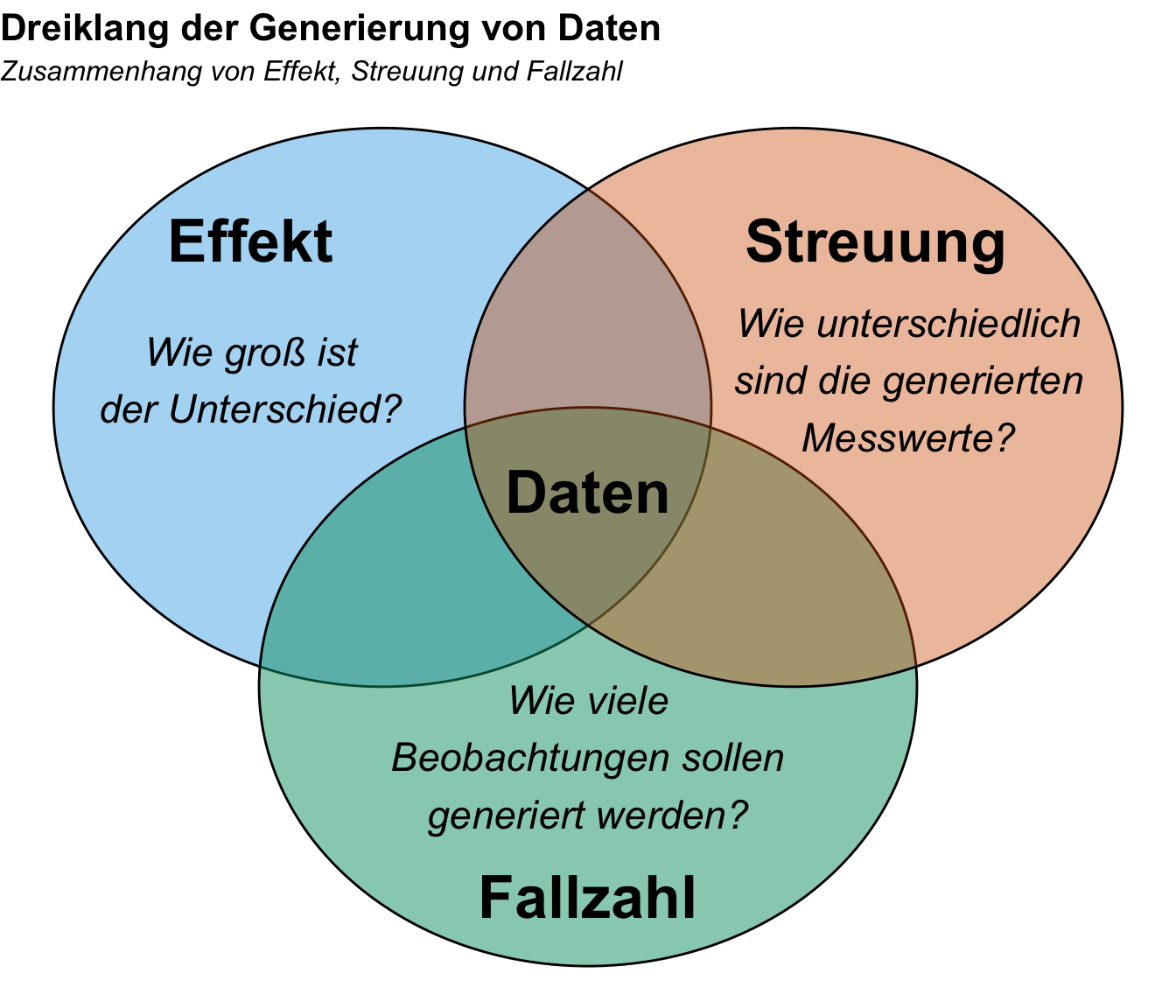
Effekt, Streuung und Fallzahl
Was brauchen wir um Daten zu generieren? Wir brauchen einen Effekt, eine Streuung der generierten Daten um den Mittelwert sowie die Fallzahl als Anzahl an Beobachtungen. Dann müssen wir noch entscheiden aus welcher Verteilung unsere Daten kommen sollen. In dem folgenden Venndiagramm habe ich dir nochmal den Dreiklang der drei statistischen Eigenschaften für die Generierung von Daten zusammengefasst. Wir müssen uns also klar werden, welchen Effekt wir simulieren wollen, welche Streuung in den Daten vorliegen soll und natürlich wie viele Beobachtungen wir in den generierten Daten vorfinden wollen.
Wir machen wir das jetzt praktisch in R. Dazu haben wir verschiedene Möglichkeiten, wir beginnen hier einmal mit der simpelsten Form. Mit der Funktion rnorm() können wir uns zufällige Daten aus einer theoretischen Normalverteilung ziehen. Es gibt natürlich noch viel mehr r*-Funktionen für die Generierung von Daten. Die Seite Probability Distributions in R zeigt nochmal einen großen Überblick. Im Kapitel zu den Verteilungen findest du dann auch nochmal mehr Informationen zu den einzelnen Funktionen.
Fangen wir aber einmal mit der einfachen Funktion für die Normalverteilung an. Hier genieren wir uns also einmal fünf Beobachtungen aus einer Normalverteilung. Wir brauchen dafür die Fallzahl (n), die wir generieren wollen. Dann den Mittelwert der theoretischen Verteilung (mean) sowie die Streuung (sd). Dann erhalten wir fünf Zahlen, die in der Gesamtheit den vorgestellten Zahlen entsprechen.
R Code [zeigen / verbergen]
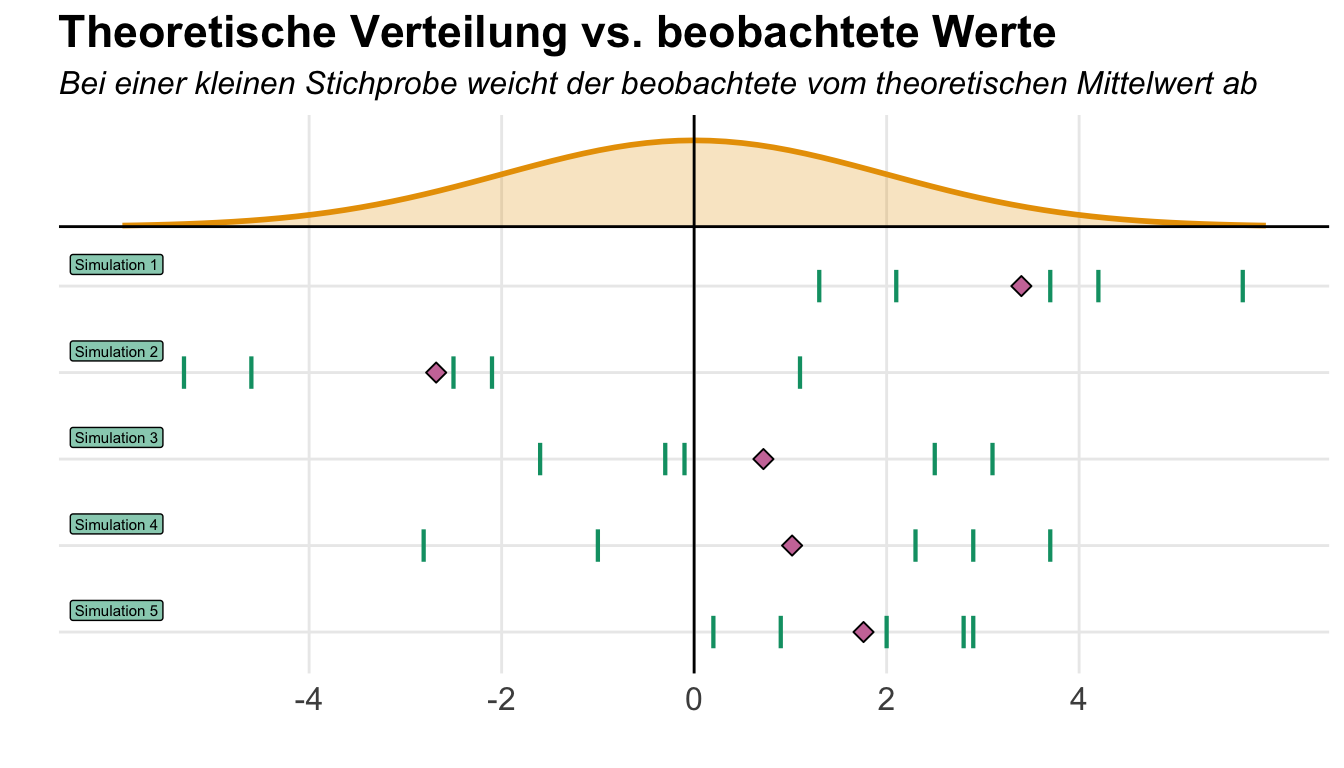
rnorm(n = 5, mean = 0, sd = 1) |> round(2)[1] 0.75 0.02 0.77 -1.17 0.39Wenn wir jetzt den Mittelwert der fünf Zahlen berechnen, dann erhalten wir aber gar nicht einen Mittelwert von \(0\) und auch nicht eine Standardabweichung von \(1\) wieder. Wie kann das sein? Häufig stolperst du hier ein wenig. Wieso Daten generieren, wenn die Daten dann nicht die Eigenschaften haben, die wir wollen? Hier kommt die Grundgesamtheit und die Stichprobe ins Spiel. In der Grundgesamtheit hast du einen Mittelwert von \(2\) und eine Standardabweichung von \(1\) vorliegen. Du ziehst aber nur eine kleine Stichprobe wie ich dir einmal in der folgenden Abbildung zeige. Zwar liegt der theoretische Mittelwert auf der Null, aber die Mittelwerte der einzelnen Simulationen können stark variieren.
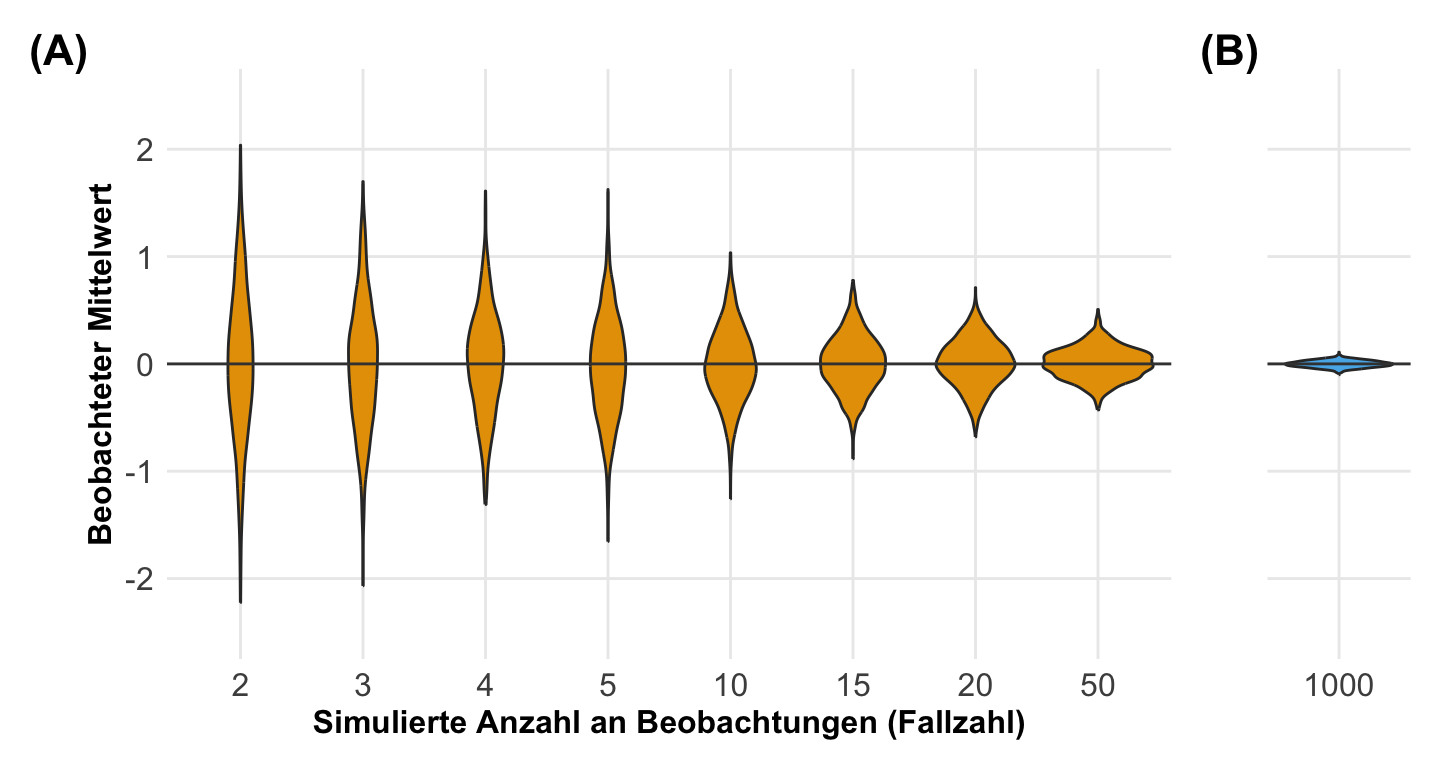
Der Zusammenhang wird dir vielleicht dann nochmal in der folgenden Abbildung klarer. Ich habe hier einmal eine kleine Simulation laufen lassen. Dabei habe ich immer Daten aus einer Standardnormalverteilung mit einem Mittelwert von \(0\) und einer Standardabweichung von \(1\) generiert. Was ich dabei geändert habe ist die Fallzahl. Wenn ich nur zwei Beobachtungen generiere, dann finde ich eine große Breite an beobachteten Mittelwerten. Wenn ich die Fallzahl erhöhe, dann komme ich immer näher an den erwarteten Mittelwert in meinen beobachteten Daten heran. Bei eintausend Beobachtungen habe ich faktisch kaum noch eine Abweichung vorliegen. Hier siehst du eben, es kommt auch stark auf die Fallzahl an. Wenn du dir zu wenig Beobachtungen anschaust, dann wirst du nicht immer den theoretischen Mittelwert oder Effekt in den generierten Daten wiederfinden.
Welche Möglichkeiten gibt es eigentlich?
Wenn wir Daten generieren wollen, dann haben wir im Prinzip zwei Möglichkeiten. Wir bauen uns die Daten selber per Hand in Excel oder aber nutzen die entsprechenden R Pakete. Ich stelle hier die zwei größeren R Pakete vor sowie die Lösungen per Hand in Excel. Wir haben natürlich auch die Möglichkeit die Daten in R ohne Pakete zu erstellen. Das macht in einem einfacheren Setting auch mehr Sinn.
- Das R Paket
{simstudy}liefert eigentlich alles was wir brauchen, wenn die Daten komplizierter werden. Insbesondere wenn wir keinen normalverteilten Messwert vorliegen haben oder aber abhängige Einflussvariablen. Wenn ich größere Datensätze generiere, dann nutze ich gerne dieses Paket. Hier gibt es aber die Einschränkung, dass sich faktorielle Experimente nicht gut darstellen lassen. In diesem Fall ist die händische Erstellung manchmal einfacher. - Das R Paket
{simdata}hat den Vorteil große, komplexe Korrelationsstrukturen abzubilden. Wir können hier Netzwerkeffekte gut darstellen und dann auch in einen Datensatz übersetzen. Auch Transformationen von Daten lassen sich hier gut integrieren. Hier liegt aber nicht der Fokus auf einem faktoriellen Design mit Gruppeneffekten. - Wir schauen uns natürlich auch Excel an. Hier haben wir natürlich gleich die Möglichkeit uns die Daten dort zu bauen, wo wir die erhobenen Messwerte dann später nach dem durchgeführten Experiment auch abspeichern. Deshalb zeige ich auch hier einmal wie wir dann die Daten händisch aufbauen.
Abschließend muss ich noch erwähnen, dass faktorielle Designs, die wir dann häufig in den Agrarwissenschaften finden, wir am besten dann händisch in R zusammenbauen. Wir nutzen dann die Funktionen, die es schon in R gibt und erstellen uns dann selber den Datensatz. Das ist im Prinzip so ähnlich wie in Excel nur das wir etwas besser nachvollziehbare Wege haben.
21.2 Theoretischer Hintergrund
Neben dem allgemeinen Hintergrund können wir natürlich auch etwas tiefer in die Materie einsteigen. Zum einen müssten wir überlegen, welche Effekte es eigentlich gibt. Wir wollen ja Daten generieren, die nicht gleich sind. Daher wollen wir etwas bauen, wo wir auch einen Effekt sehen. Zwar ist es auch interessant sich Daten zu generieren wo die Nullhypothese wahr ist und nichts passiert, aber das ist eher nicht der Fokus der praktischen Arbeit. Wenn wir ein Experiment machen, dann wollen wir eigentlich auch einen Effekt oder Unterschied in den Gruppen oder allgemeiner den Daten sehen.
Welche Effekte gibt es?
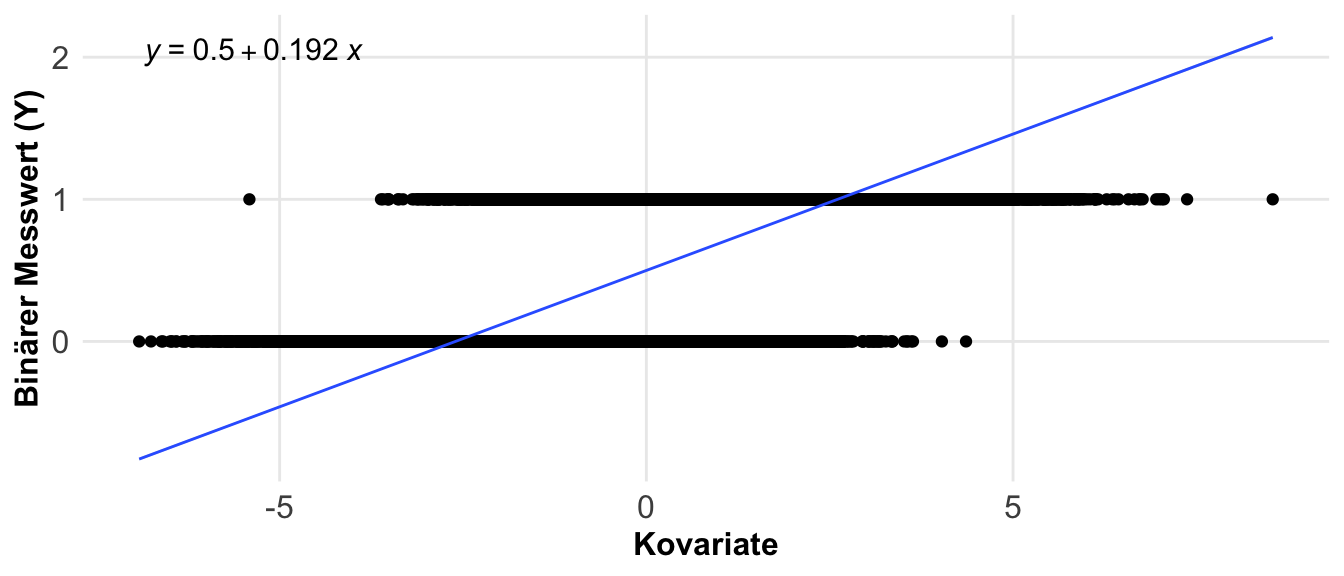
Wenn wir also Wissen, dass wir einen Effekt \(\Delta\), eine Streuung \(s\) und Fallzahl \(n\) definieren müssen um uns zufällige Daten zu generieren, was sind denn dann die Effekte? Die Streuung und die Fallzahl sind ja relativ einfach zu verstehen. Die Fallzahl legen wir fest und es ist eben die Anzahl an Beobachtungen, die wir generieren wollen. Im Prinzip also die Anzahl an Zeilen in unserem Datensatz. Die Streuung ist schon etwas schwerer zu fassen, ist aber am Ende nichts anderes als die Varianz in den Daten. Also zum Beispiel bei einer Normalverteilung, wie stark streuen meine Beobachtungen um den Mittelwert. Die Effekt sind schon etwas schwerer zu greifen, da wir hier erst mal definieren müssen, welche Effekte es gibt. Grob gesprochen gibt es im Rahmen der Generierung von Daten zwei Effekte von Bedeutung. Entweder sind wir an einer Mittelwertsdifferenz interessiert oder aber an einem Anteil.
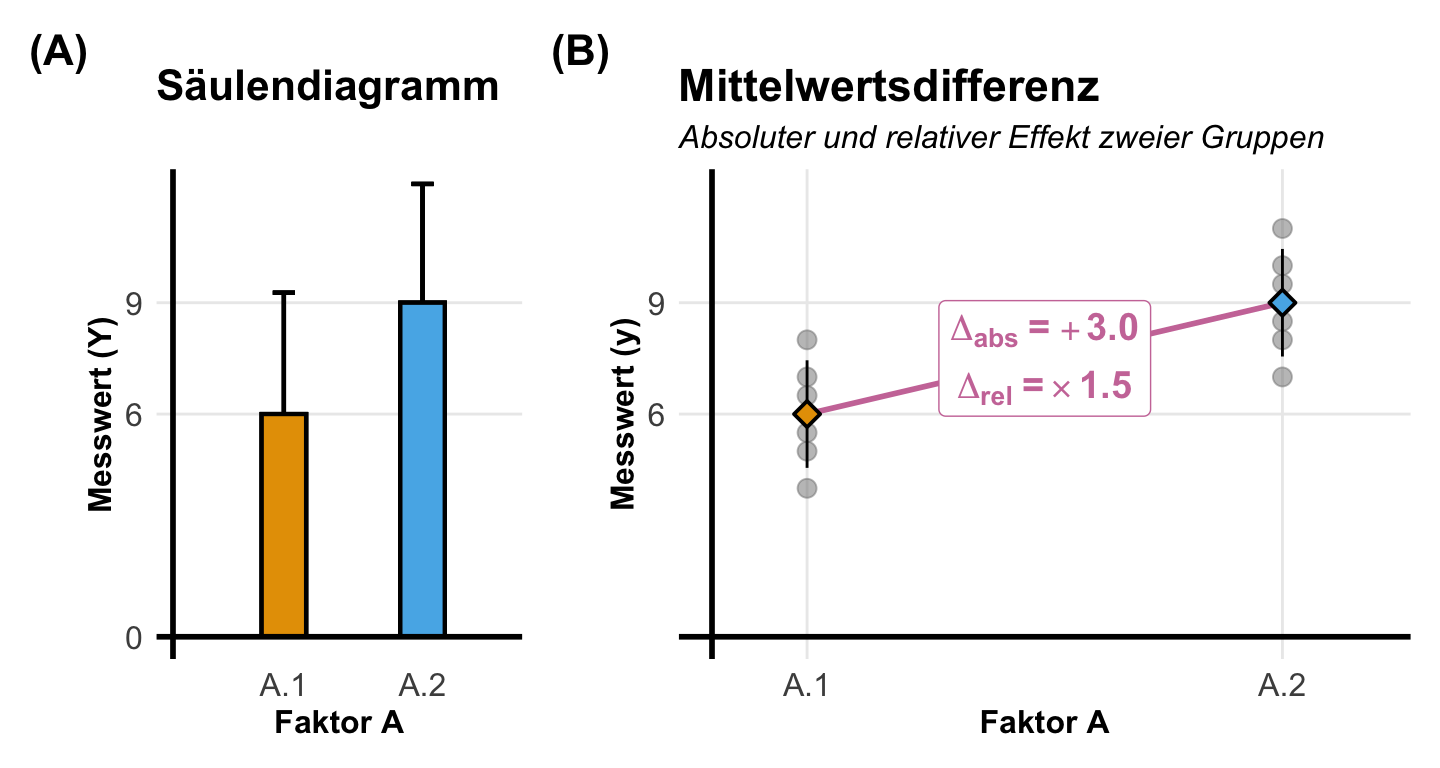
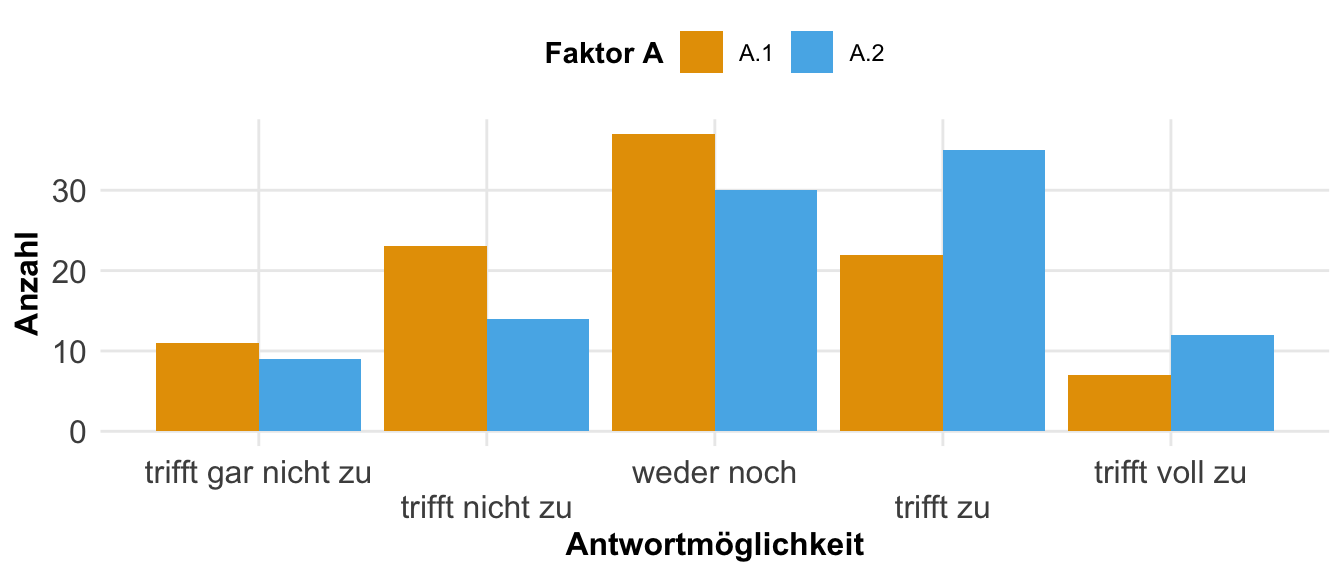
Die Mittelwertsdifferenz wird sehr häufig als Effektschätzer genutzt, wenn wir Daten erstellen wollen. Du kennst die Mittelwertsdifferenz schon aus der Darstellung der Mittelwerte von Gruppen anhand des Barplots. Hier können wir als Effekt entweder auf den absoluten Mittelwertsunterschied hinaus wollen oder aber die relative Veränderung angeben. Im Rahmen der Simulation nutzen wir die absolute Differenz sehr viel häufiger. In der folgenden Abbildung habe ich dir einmal für zwei Gruppen in einem Faktor einen Barplot sowie die Mittelwertsdifferenz mit Dotplots dargestellt.
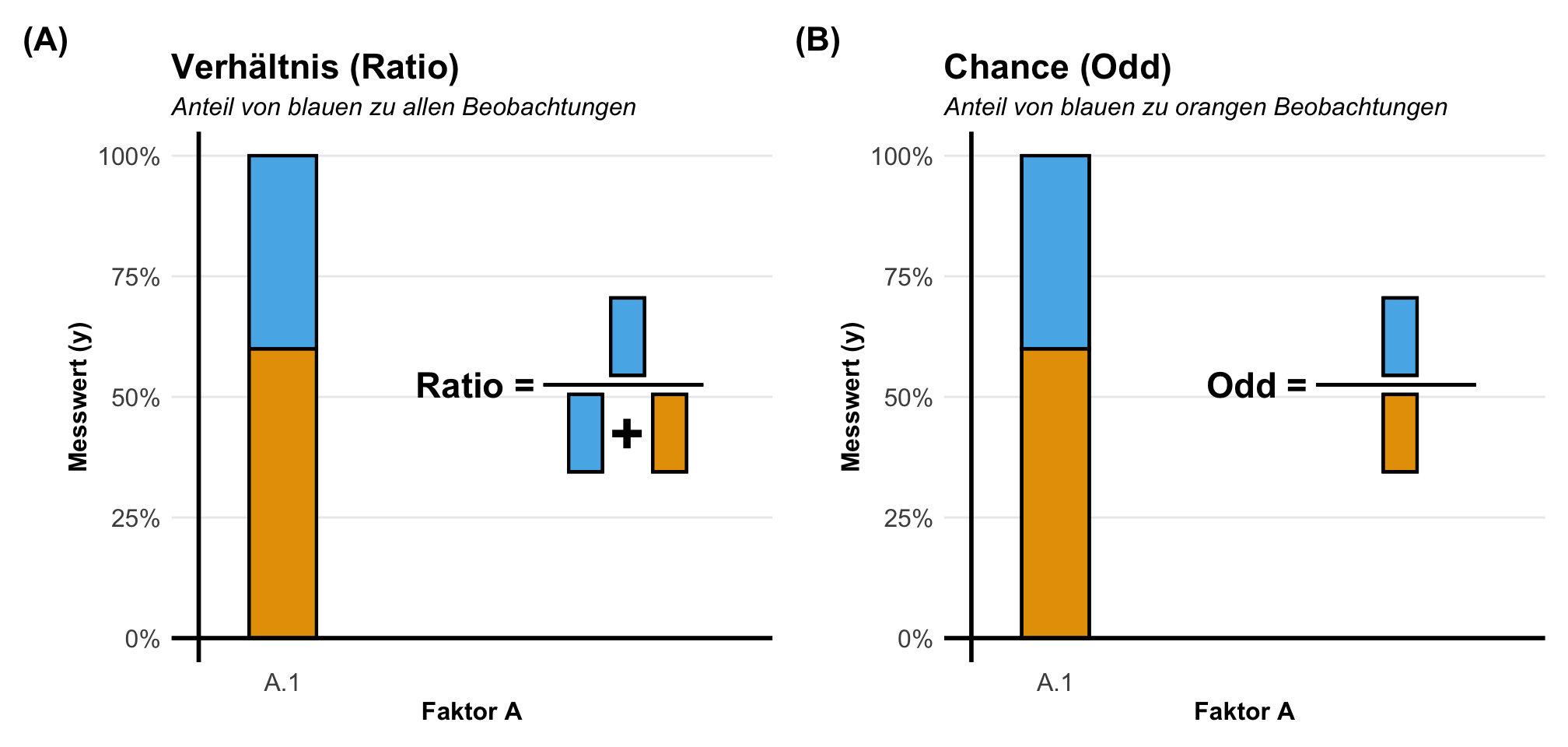
Eine andere Möglichkeit sind die Anteile. Hier fragen wir, wie häufig tritt ein Ereignis in der Population auf? Daher ist die Pflanze krank? Oder aber, liegen wir im Zielbereich? Wir sind hier an einem Anteilsverhältnis interessiert. Anteile werden in unserem Kontext eigentlich immer mit ja (1) oder nein (0) kodiert. Wenn wir also zwei Gruppen haben, die gesunden und die kranken Pflanzen, dann können wir die Anteile an gesunden und kranken Pflanzen auf zwei Arten berechnen. Entweder sind wir an dem Anteil von den kranken Pflanzen an allen Pflanzen interessiert, dann rechnen wir ein Verhältnis. Wenn wir aber den Anteil von kranken Pflanzen an gesunden Pflanzen wollen, dann berechnen wir die Chance. In der folgenden Abbildung habe ich dir den Zusammenhang nochmal dargestellt.
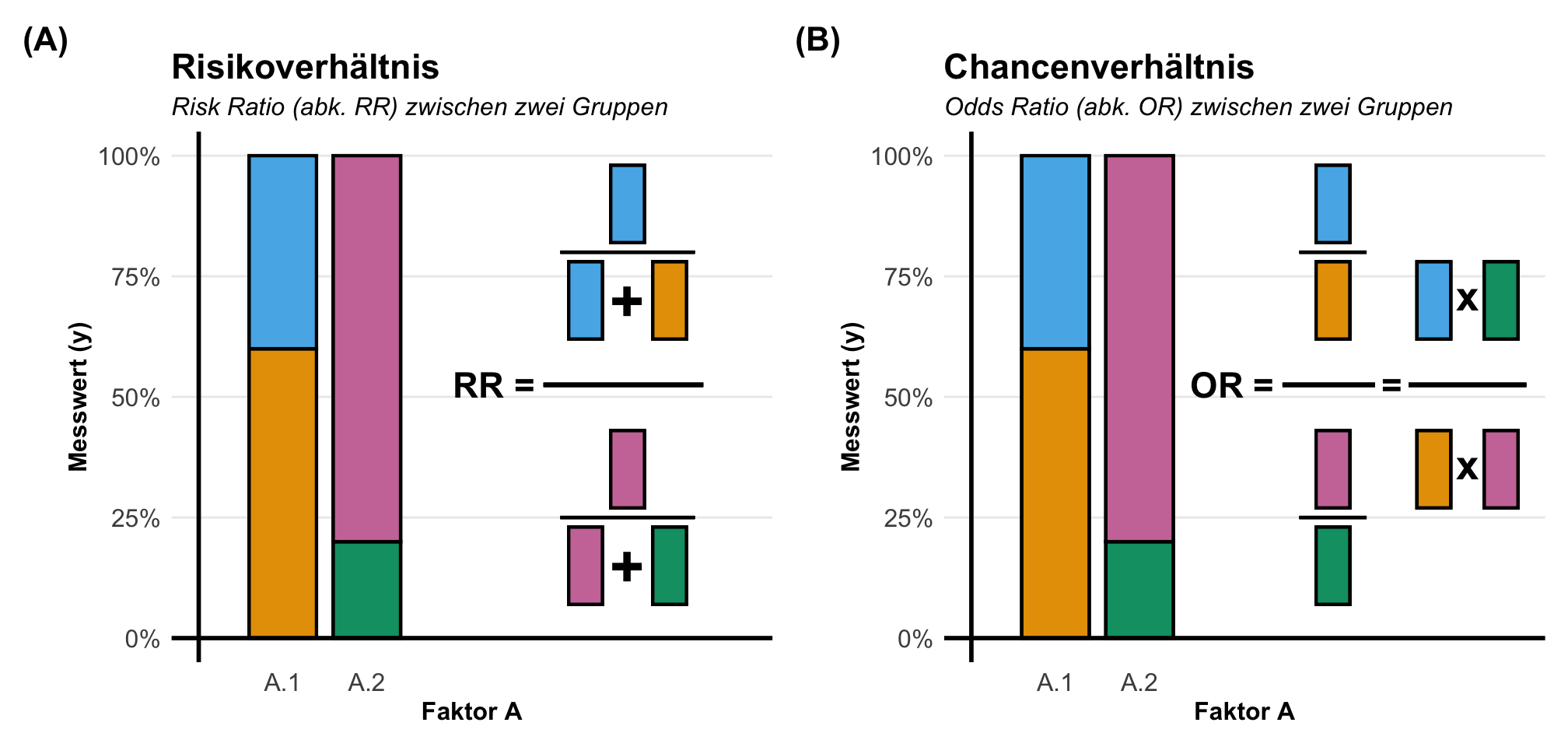
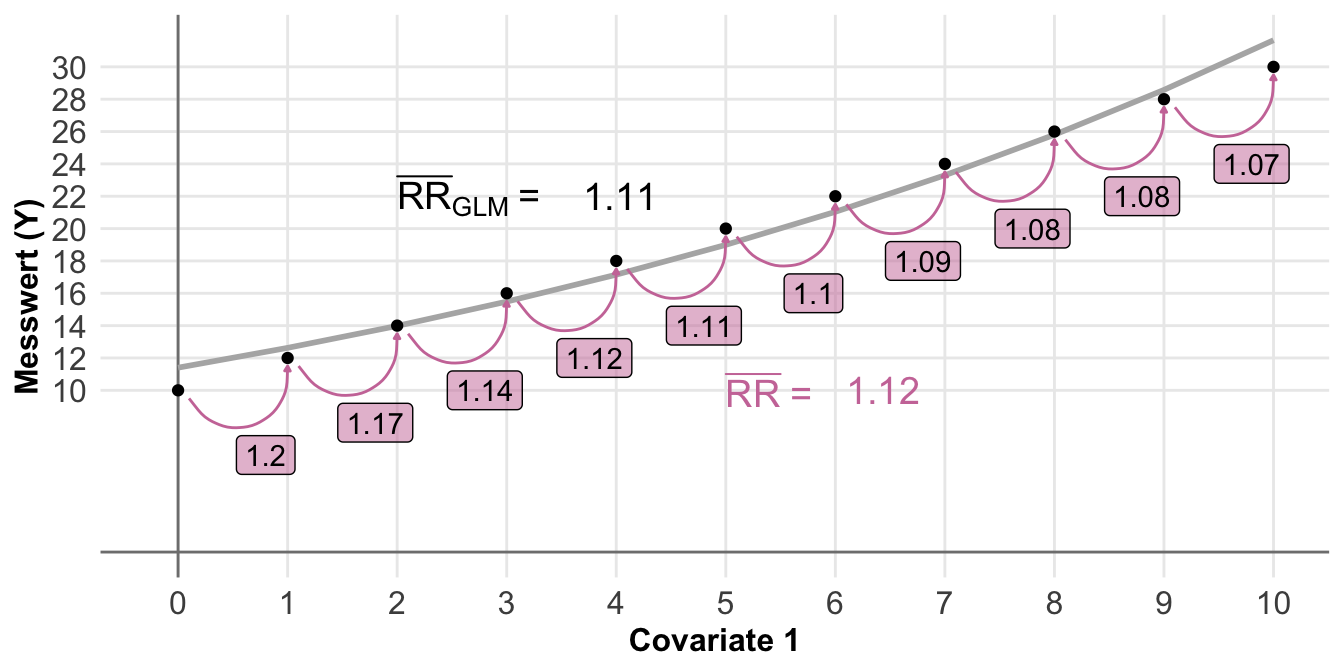
Etwas wilder wird es jetzt, wenn wir die Effekte zwischen zwei Gruppen mit Anteilen berechnen wollen. Hier nutzen wir dann nicht die Differenz sondern ebenfalls den Anteil. Daher haben wir es hier mit einem Anteil von einem Anteil zu tun. Daher berechnen wir hier dann das Risikoverhältnis (eng. risk ratio) oder aber das Chancenverhältnis (eng. odds ratio). Als reine Formel ist es dann meistens verwirrender, deshalb hier einmal als Abbildung visualisiert. In dem Kapitel zu den Effektschätzern kannst du noch mehr lesen und dein Wissen vertiefen.
Was nehmen wir hier mit?
- Die Mittelwertsdifferenz ist zu gebrauchen, wenn wir von einem normalverteilten Messwert ausgehen. Oder aber, wenn wir mit den mittleren Effekt leben können. Wir wollen also die mittlere Anzahl an Blattläusen anstatt die Anzahl vergleichen. Oder aber die mittlere Infektionsrate anstatt den Anteil der kranken Pflanzen.
- Anteilsverhätnisse nutzen wir, wenn wir einen Messwert vorliegen haben, der nicht normalverteilt ist und wir über die Anteile von Gruppen in unserem Messwert sprechen wollen. Daher wir wollen über den Anteil in den Boniturnotenstufen reden oder aber den Anteil an kranken Pflanzen modellieren.
Häufig reicht erstmal eine Genierung mit dem Mittelwert und der Mittelwertsdifferenz. Wir können uns vielen Fragstellungen erst mal mit einem Mittel annäheren und dann später nochmal differenzierter Auswerten. Beachte immer, das es hier ja um die Erstellung von künstlichen Daten geht, die selten die Komplexizität echter Daten abbilden können. Daher habe ich hier auch den Fokus auf der Normalverteilung und der Generierung von mittleren Differenzen.
Effekt und Streuung
Jetzt müssen wir noch den Zusammenhang zwischen Effekt und Streuung verstehen. Je höher deine Fallzahl ist, desto eher wirst du in den genierten Daten deine voreingestellten Effekte wiederfinden. Es gibt aber noch die Variable der Streuung. Wenn du eine Streuung mit ins Spiel bringst, dann werden natürlich auch deine Effekte wiederum verzerrt. Bei einer Streuung erhälst du nicht deine voreingestellten Werte genau so wieder. Das ist manchmal etwas verwirrend, aber auch häufig der Grund warum wir nicht nur einen Datensatz simulieren sondern viele, die wir dann eben mitteln. Das hat aber keine Relevanz, wenn du deine Daten generierst um deine Analysepipeline zu testen. Wir schauen uns jetzt einmal den Fall eines faktoriellen und eines kovariaten Designs an.
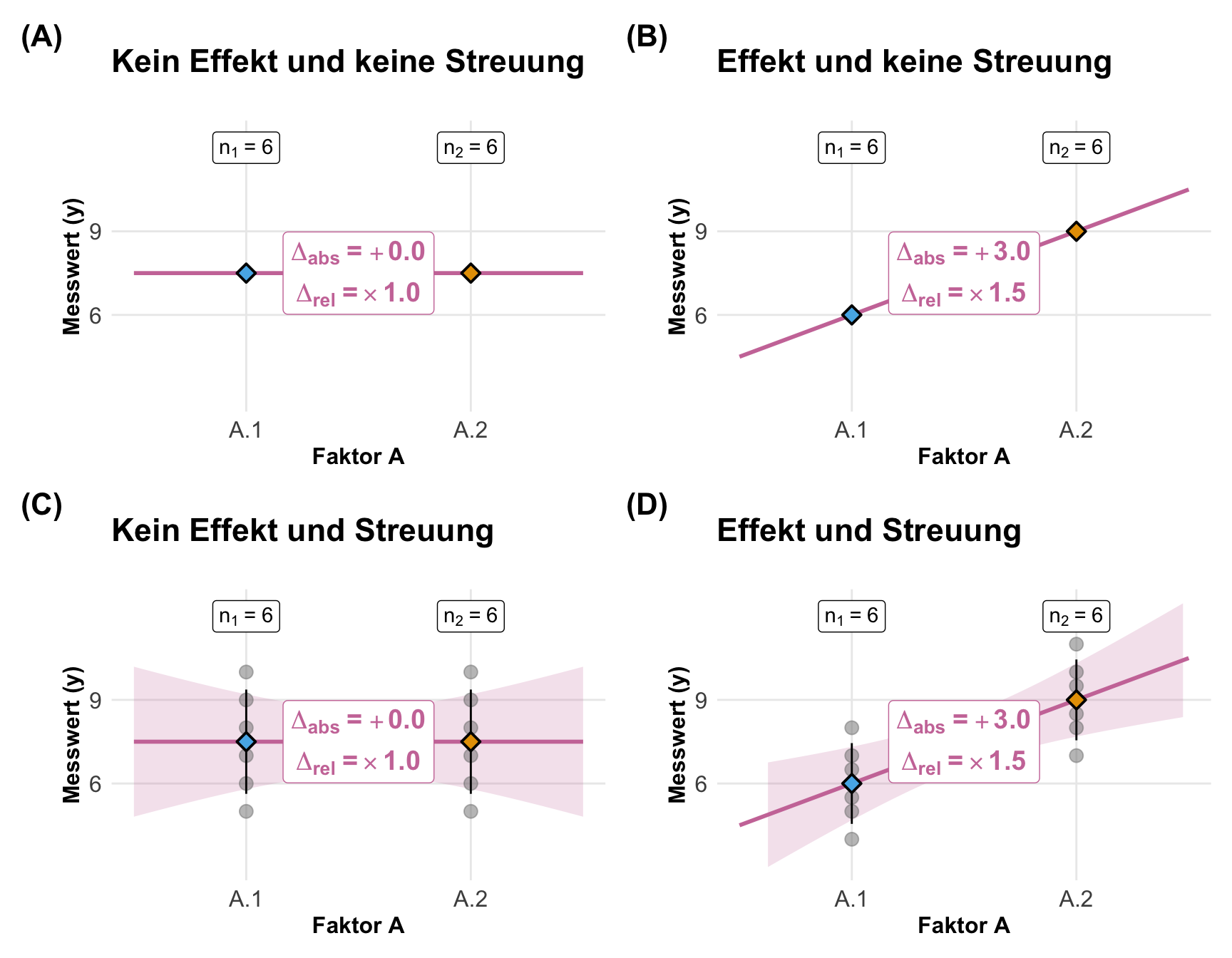
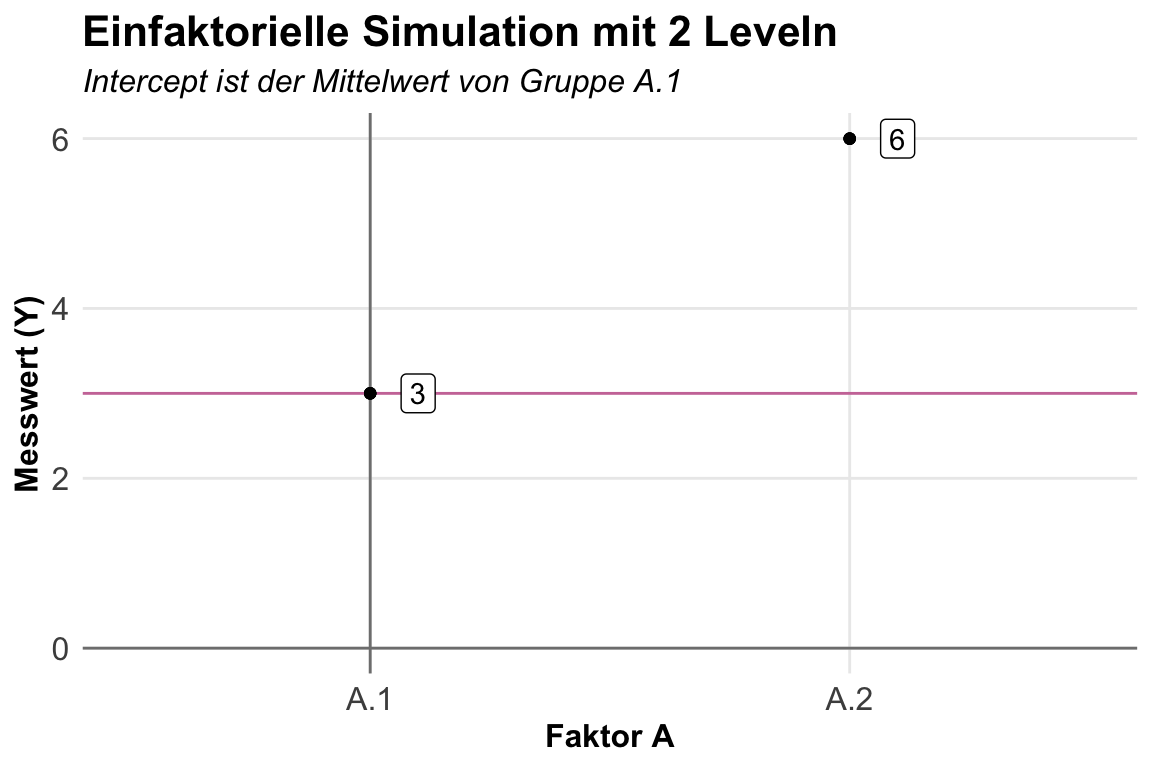
In dem folgenden Beispiel habe ich ein einfaktorielles Design mit nur zwei Leveln gewählt. Wir sehen recht gut, das wir bei keinem Effekt keinen Mittelwertsunterschied vorliegen haben. Ich habe hier eine Mittelwertsdifferenz von \(+3\) voreingestellt. Wenn wir dann auch keine Streuung vorliegen haben, dann liegen alle Beobachtungen auf dem Mittelwert als ein Punkt. Wenn wir dann Streuung vorliegen haben, dann spreizen die Punkte auf dem Faktorlevel. Durch die Streuung können wir dann auch nicht mehr exakt die voreingestellten Effekte wiederfinden. Je kleiner die Fallzahl, desto größer ist dann die Abweichung von den voreingestellten Effekten.
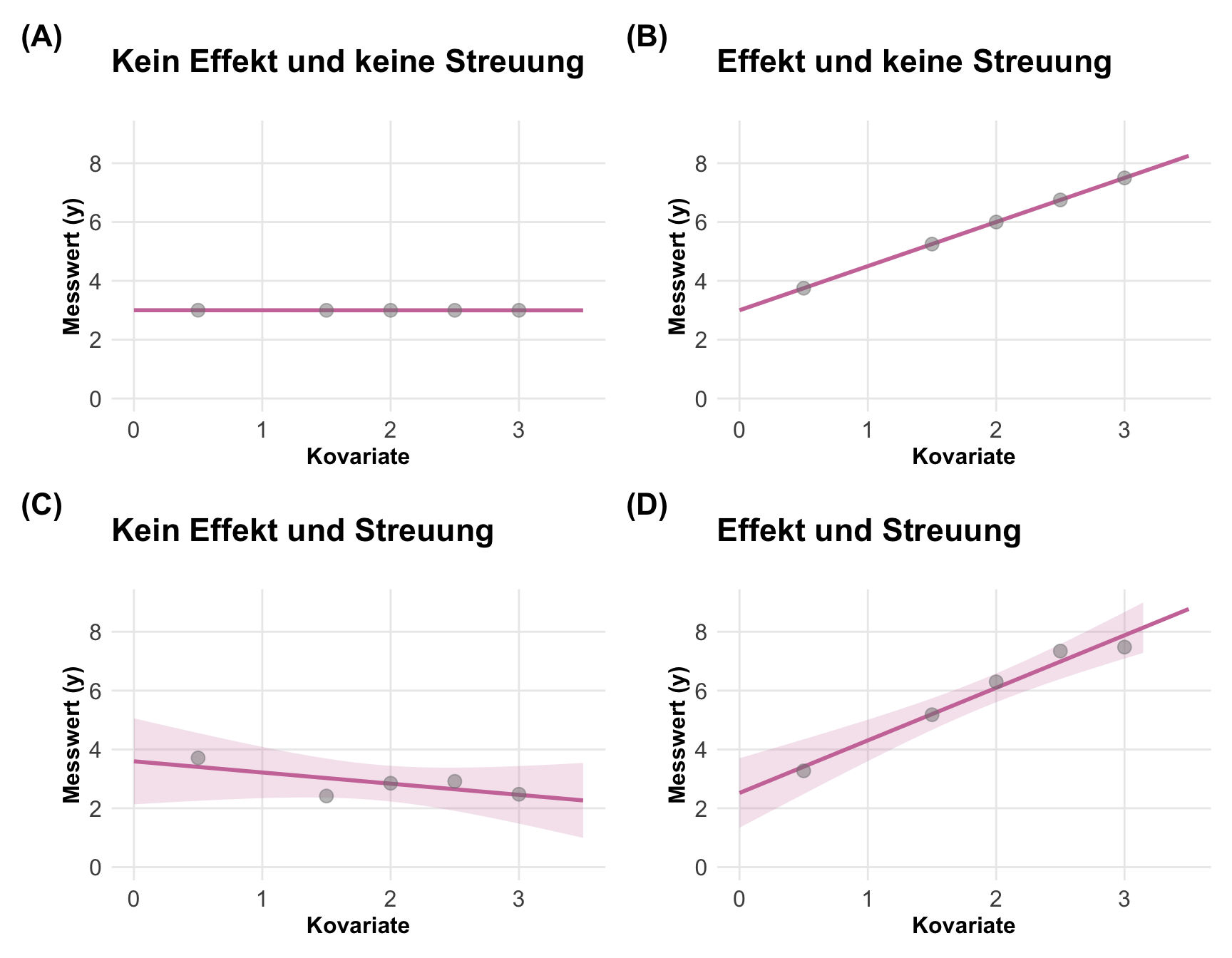
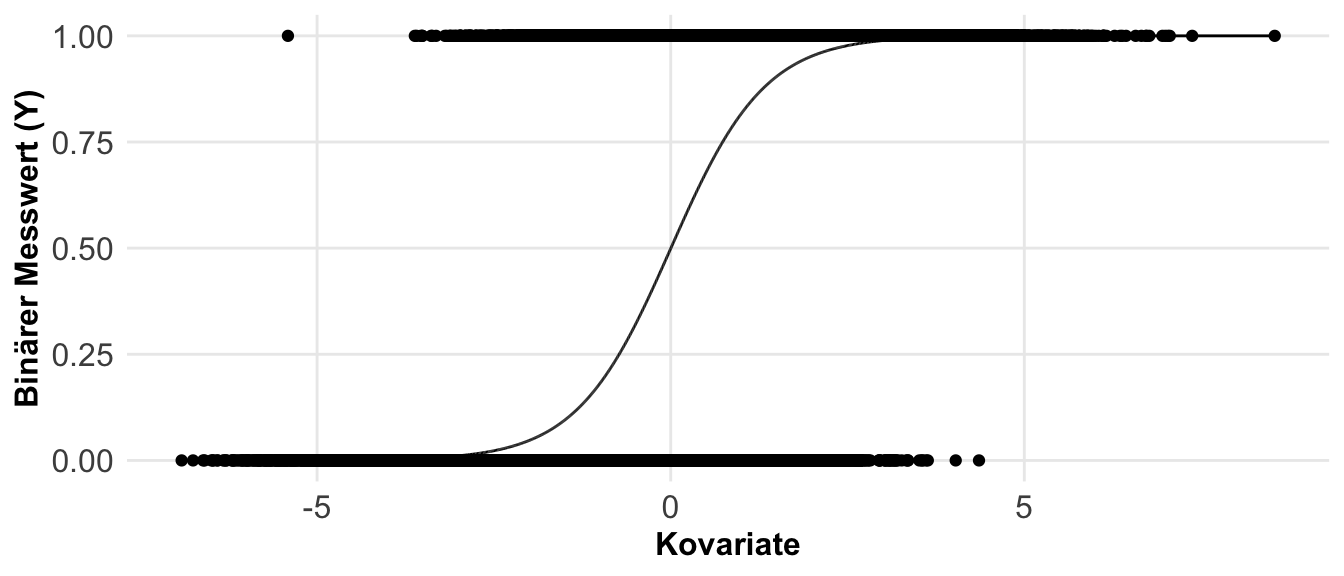
Haben wir ein kovariates Design vorliegen, dann wollen wir ja die Steigung in der Graden als Effekt einstellen. Ich habe hier eine Steigung von \(+1.5\) und einen Intercept von \(3\) voreingestellt. Wir brauchen immer etwas Streuung, sonst kann der Algorithmus keine Grade berechnen. Daher hier auch bei keiner Streuung ein winziger Wert, den wir dann eben auch in der Steigung wiederfinden. Ohne Streuung erhalten wir auch die voreingestellten Werte für die Steigung wieder. Haben wir keinen Effekt der Kovariaten auf den Messwert, dann haben wir auch keine Steigung. Sobald wir Streuung ins Spiel bringen, haben wir auch eine mehr oder minder starke Abweichung in den Daten. Insbesondere bei keinem Effekt und dann noch Streuung kann man schnell denken, dass wir doch was in den Daten hätten.
Einschränkungen bei der Generierung
Wenn wir über die Simulation von Daten sprechen, dann kommen wir nicht darum herum einmal über die schrecklich nette Familie der Verteilungen zu sprechen. Wir haben ja nicht nur auf der rechten Seite der Tilde unsere Einflussvariablen, die entweder Faktoren oder Kovariaten sind, sondern auch die Messwerte, die wir simulieren wollen. Diese Messwerte gehören aber einer Verteilungsfamilie an. Zwar ist die Normalverteilung (eng. gaussian) sehr häufig aber können wir mit der Normalverteilung nicht alle Messwerte abbilden. Daher brauchen wir auch die anderen Verteilungen. Daher hier einmal die Übersicht für dich. Das R Paket {simstudy} hat eine Vielzahl an Verteilungen implementiert auf die wir hier nicht tiefer eingehen werden.
Wir konzentrieren uns hier nur auf einen kleinen Teil der möglichen Simulationen und Datengenerierungen. Das hat neben der vielen möglichen Verteilungen auch den Grund, dass wir usn hier ja nicht mit der algorithmischen Modellentwicklung in der Statistik beschäftigen wollen. Wir wollen ja nicht neue Algorithmen bauen und diese dann auf Daten evaluieren. Daher konzentrieren wir uns hier auf wenige Verteilungen, die dann aber das abbilden, was wir häufig brauchen. Wenn du mehr über Verteilungen wissen willst, dann ist die Shiny App The distribution zoo einen Besuch wert. Als Literatur kann ich dir nur Dormann (2013) empfehlen, der sehr detailliert für Einsteiger auf Verteilungen eingeht.
- Die Normalverteilung erlaubt uns das Frischegewicht, Trockengewicht oder aber Chlorophyllgehalt zu simulieren. Also im Prinzip die Messwerte, die wir hauptsächlich in einem agrawissenschaftlichen Umfeld erheben.
- Die Possionverteilung erlaubt uns alles was wir Zählen zu simulieren. Also im Prinzip alle Anzahlen von Schädlingen oder aber Blütenstände sowie Blättern. Wir zählen hier eben etwas.
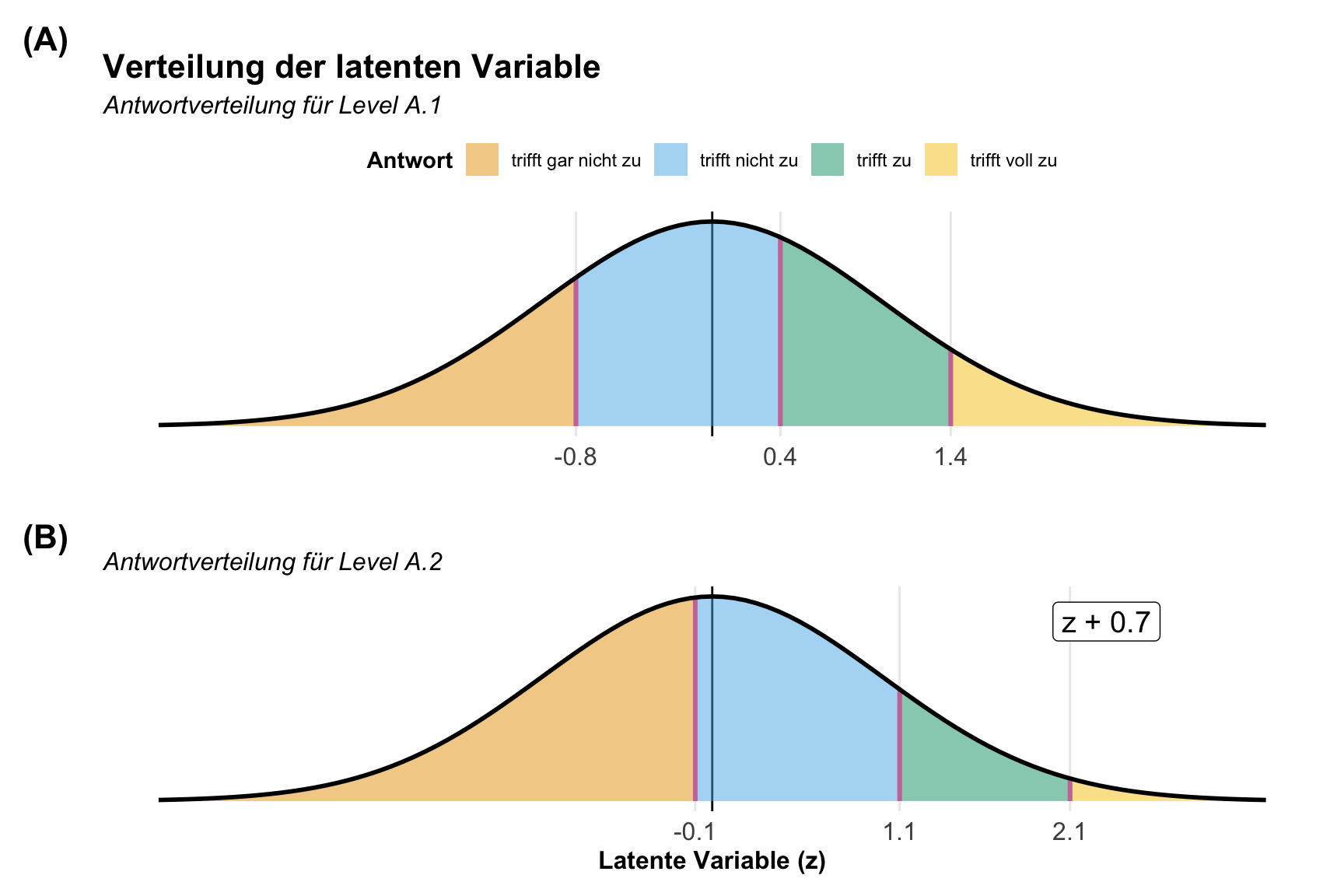
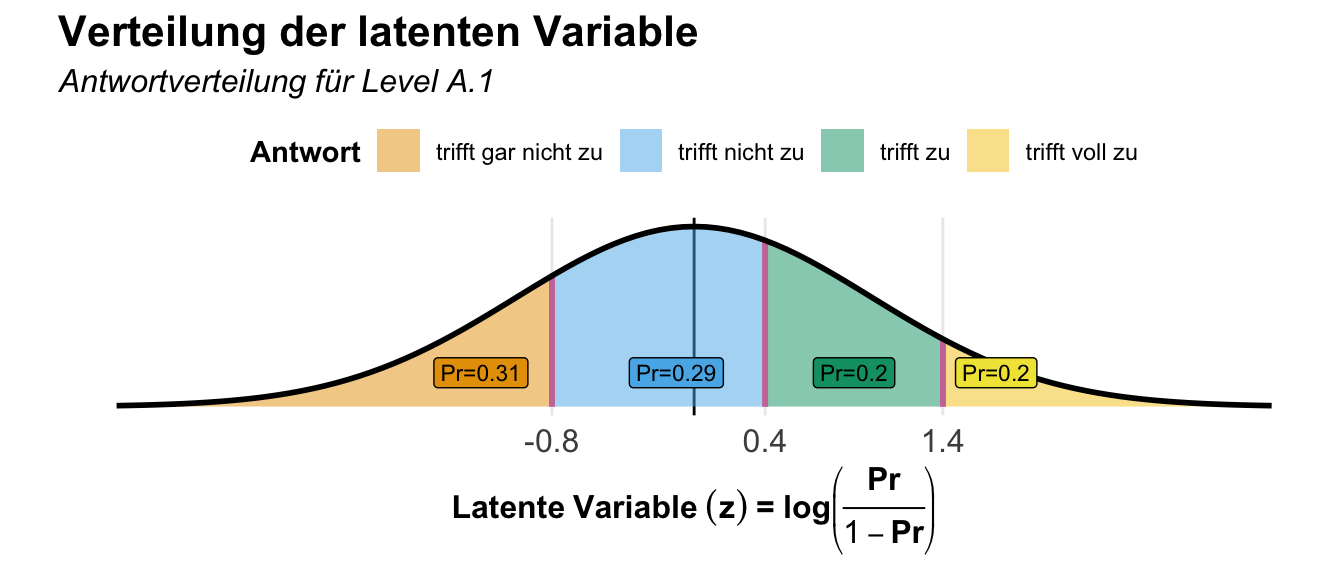
- Die Ordinalverteilung ist dafür gut, wenn wir Noten simulieren wollen. Daher brauchen wir die Ordinalverteilung um unsere Boniturnoten zu generieren. Prinzipiell aber auch die Antwortverteilungen in einem Fragebogen.
- Die Binomialverteilung ist die Verteilung, die wir nutzen, wenn wir nur zwei mögliche Messwerte haben. Also im prinzip gesunde Beobachtung oder kranke Beobachtung. Wir haben nur zwei Ausprägungen in dem Messwert vorliegen.
Neben den vorgestellten Verteilungen schauen wir uns dann noch die Simulation von pseudo Zeitreihen an. Wir haben hier dann verschiedene Messwerte über einen zeitlichen Verlauf vorliegen. Teilweise dann in einer etwas wirren Form, da wir eventuell keinen linearen Zusammenhang mehr vorliegen haben. Daher müssen wir dann hier etwas mehr schauen, dass wir die Daten gut simuliert kriegen.
Dann mache ich hier noch die Einschränkung, dass ich prinzipiell alles unter der Annahme der Varianzhomogenität generiere. Daher addiere ich über alle Faktorkombinationen oder Gruppen immer den gleichen Fehler auf. Im Prinzip kannst du natürlich auch für einzelne Faktorkombinationen dann einen anderen Fehler addieren und dir dafür einen Vektor mit unterschiedlichen Fehlertermen bauen, aber darauf gehe ich nur einmal kurz bei dem einfakoriellen Design ein. Und dann noch die Ergänzung, dass wir uns immer nur balancierte Designs erstellen. Das heißt wir haben keine fehlenden Werte in unseren Daten und die Gruppen in unseren Faktoren haben auch immer die gleiche Fallzahl.
Selbst gebaute Verteilungen
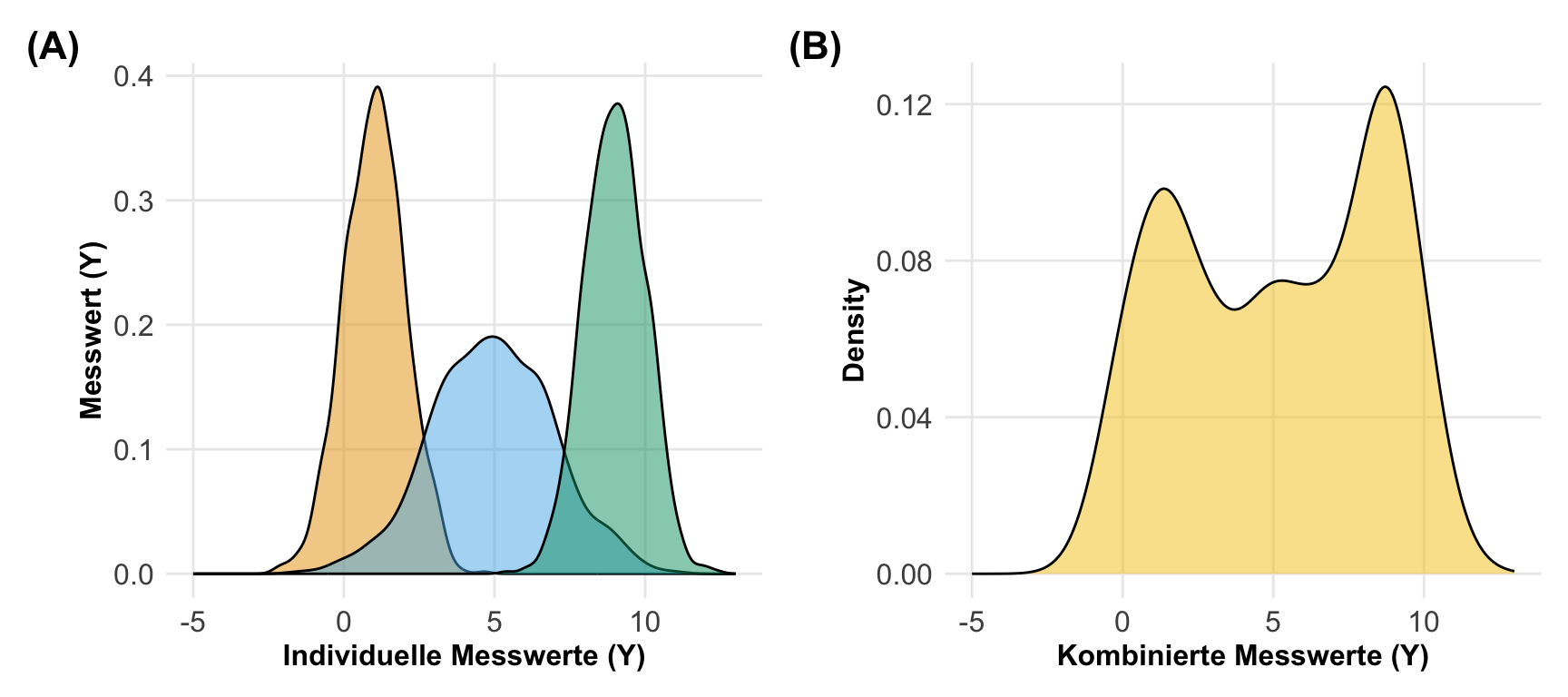
Am Ende möchte ich noch auf eine andere Art der Simulation von Daten eingehen. Wir können uns auch eine wilde Verteilung selber bauen und dann aus dieser wilden Verteilung dann unsere Beobachtungen ziehen. In dem Tutorium Wilcoxon is (almost) a one-sample t-test on signed ranks wird einmal gezeigt wie wir das machen könnten. Wir bauen uns dazu erst mal eine Verteilung, die aus drei Verteilungen besteht. Wir haben hier aber ein paar Einschränkungen. Zum einen ist unsere Verteilung um die Null angeordnet. Das macht es einfacher verschiedene Verteilungen miteinander zu verbinden.
R Code [zeigen / verbergen]
null_dist_tbl <- tibble(y = c(rnorm(10000),
exp(rnorm(10000)),
runif(10000, min=-3, max=-2)),
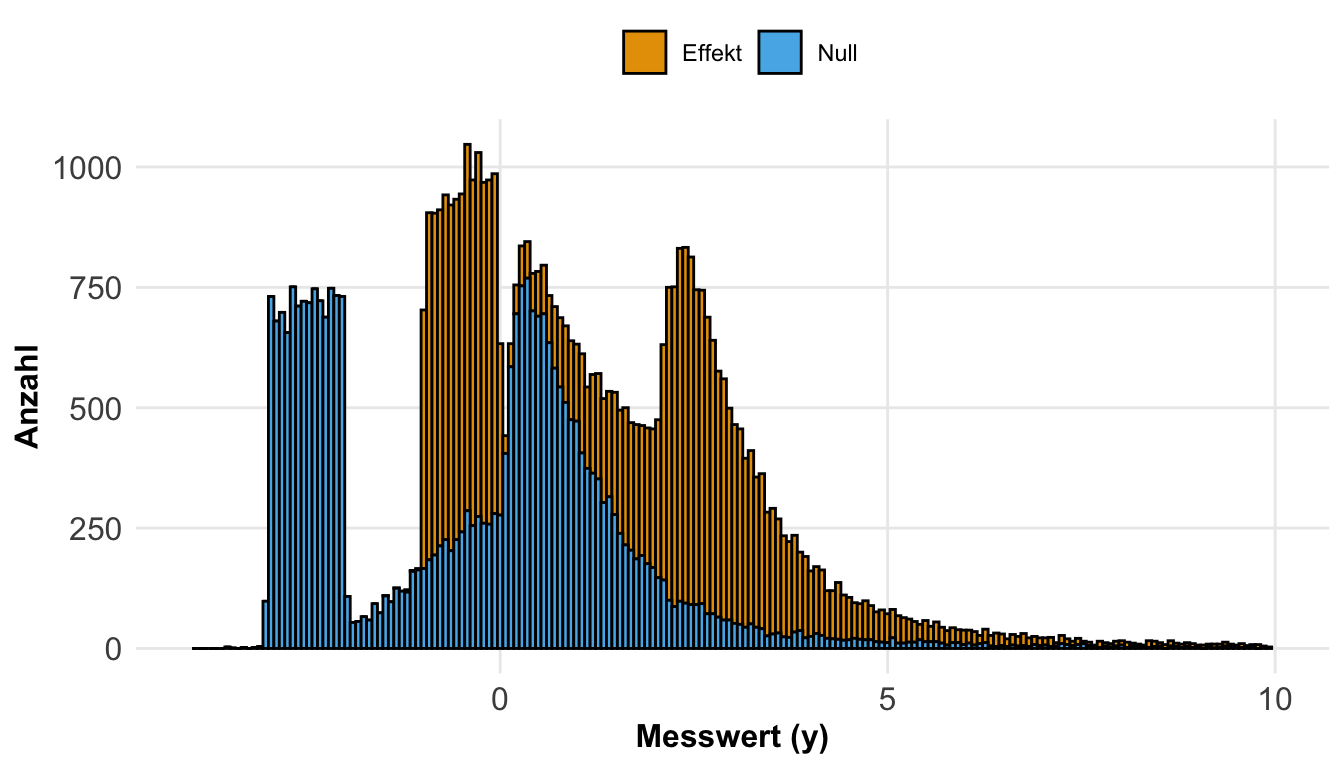
type = "null")Dann siehst du hier einmal die Verteilung dargestellt. Wenn wir nur aus dieser Verteilung Beobachtungen ziehen, dann haben wir natürlich einen Effekt zwischen potenziellen Gruppen.
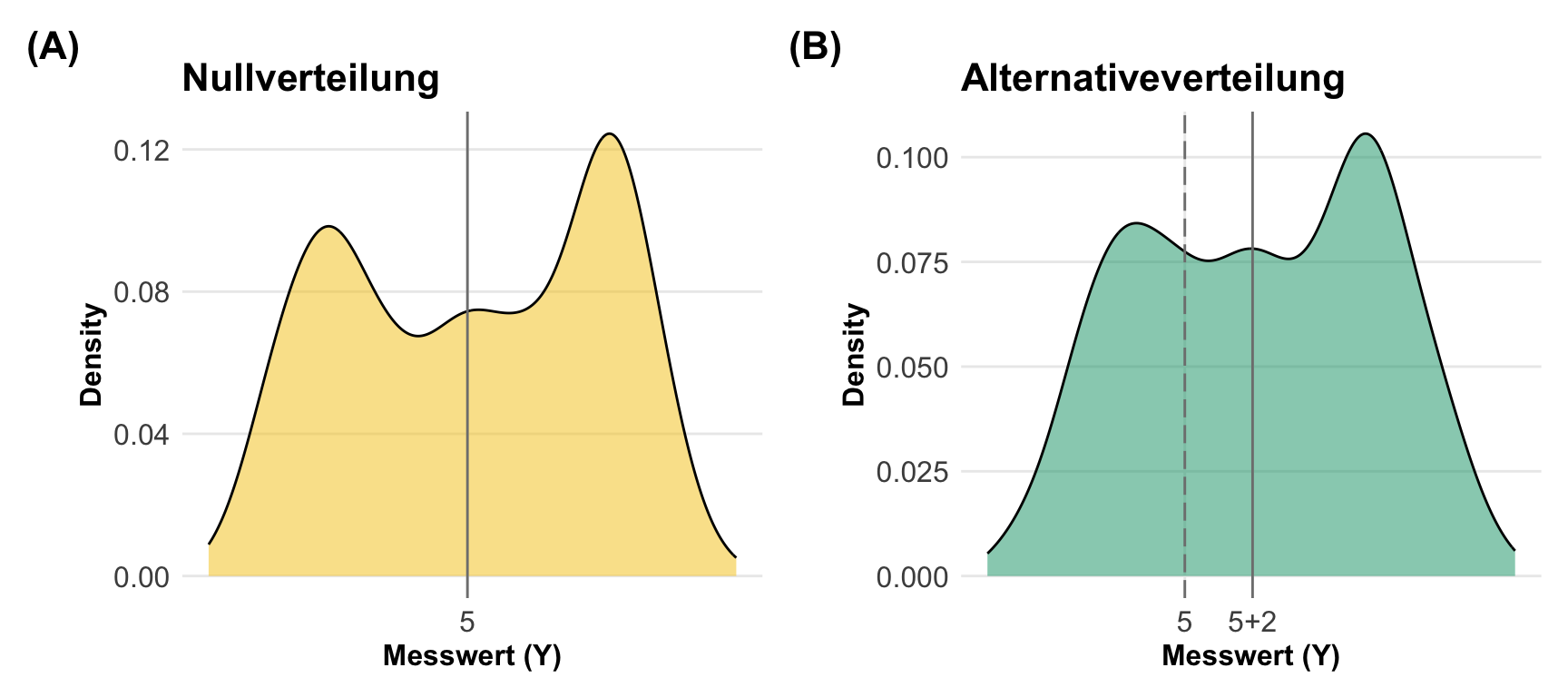
Wenn wir jetzt noch einen Effekt haben wollen, dann können wir natürlich die beiden Gruppen nicht aus der gleichen Verteilung ziehen. Daher können wir uns auch noch eine Alternative Verteilung bauen indem wir einfach die Messwerte um einen fixen Effekt erhöhen. Hier wähle einen Effekt von \(+2\). Du kannst natürlich auch die Nullverteilung erstmal aus dem negativen herausschieben und dann nochmal einen Effekt addieren.
R Code [zeigen / verbergen]
eff_dist_tbl <- tibble(y = c(rnorm(10000),
exp(rnorm(10000)),
runif(10000, min=-3, max=-2)),
type = "alt") |>
mutate(y = y + 2) |>
bind_rows(null_dist_tbl)In der folgenden Abbildung siehst du dann einmal wie sich die beiden Verteilungen mit den unterschiedlichen Effekten ergeben. Bitte beachte, dass die Alternative erst mal neu generiert wurde und nicht die verschobene Nullhypothese ist. Daher hat die Alternative natürlich auch eine andere Verteilung als die Nullhypothese.
Für die Generierung deiner Daten würdest du dann mit slice_sample() dir dann die gewünschten Anzahlen aus den beiden Verteilungen rausziehen. Hier hilft die tolle Funktion group_by(), die dir dann alles in einem Abwasch macht. Ich will hier pro Gruppe dann nur drei zufällig ausgewählte Beobachtungen haben.
R Code [zeigen / verbergen]
eff_dist_tbl |>
group_by(type) |>
slice_sample(n = 3)# A tibble: 6 × 2
# Groups: type [2]
y type
<dbl> <chr>
1 -0.794 alt
2 2.86 alt
3 2.83 alt
4 -0.697 null
5 6.27 null
6 -1.13 null Bei den selbstgebauten Verteilungen ist die Effektzuweisung aber überhaupt nicht einfach und ab zwei Gruppen wird es wirklich nicht mehr schön. Daher zeige ich dir hier nur die Idee, in der Anwendung zeige ich dir etwas robustere Varianten, die einfacher zu bedienen sind. Wenn es aber wirklich mal was wirres sein soll, dann ist diese Art der Verteilungserstellung wirklich eine Möglichkeit um auf künstliche Daten zu kommen.
Wir immer geht natürlich mehr als ich hier Vorstellen kann. Du findest im Folgenden Tutorien, die mich hier in dem Kapitel inspiriert haben. Ich habe mich ja in diesem Kapitel auf die Durchführbarkeit in R und die allgemeine Verständlichkeit konzentriert.
- Einen guten Überblick liefert Brad Duthie in seinem Tutorium Creating simulated data sets in R. Hier kannst du dich dann nochmal in die Grundlagen einlesen.
- Auch Excel kann künstliche Daten generieren. Dafür schaue ich dann immer in die statistischen Funktionen (Referenz) Seite nach. Die Seite ist leider etwas schwer zu lesen.
- Getting started simulating data in R: some helpful functions and how to use them gibt nochmal einen guten Überblick über die Standardfunktionen in R um sich schnell Daten zu generieren.
- The distribution zoo sowie Probability Distributions in R oder aber auch Plotting Continuous Probability Distributions In R With ggplot2 gben nochmal einen Einblick in die Verteilungen. Dabei würde ich den Distribution Zoo als Shiny App erstmal vorziehen. Die anderen beiden Quelle liefern nochmal mehr mathematischen Hintergrund.
21.3 Genutzte R Pakete
Wir wollen folgende R Pakete in diesem Kapitel nutzen.
R Code [zeigen / verbergen]
pacman::p_load(tidyverse, simstudy, simdata, modelr,
ggpmisc, janitor, emmeans, correlation,
data.table, ordinal, marginaleffects, conflicted)
set.seed(20250820)
conflicts_prefer(ggplot2::annotate)An der Seite des Kapitels findest du den Link Quellcode anzeigen, über den du Zugang zum gesamten R-Code dieses Kapitels erhältst.
21.4 R Paket {simstudy}
Der Fokus in diesem Kapitel liegt auf dem R Paket {simstudy} und deren Funktionen. Wir werden in dem Abschnitt zu der Normalverteilung nochmal auf die Standardfunktionen in R eingehen und die Daten etwas mehr händisch genierieren. Wennn die Verteilungen aber komplizierter werden, dann wird es auch mit dem Standard schwerer. So ist die Poissonverteilung, die Ordinalverteilung und auch die Binomialverteilung in einem mehrfaktoriellen Design schon nicht mehr schön selber zu bauen. Insbesondere wenn die Einflussvariablen noch untereinander miteinander in Beziehung stehen.
In der folgenden Tabelle siehst du einmal die drei Variablen für das Alter, das Geschlecht und die Sprungweite von Katzenflöhen. Wir sagen, dass der Effekt des Alters immer fix \(10\) ist. Das Geschlecht des Flohs hängt hier absurderweise vom Alter ab. Die Sprungweite ist dann eine Funktion des Alters und des Geschlechts. Dabei haben nicht alle Verteilungen eine Varianz wie die Normalverteilung. Deshalb haben wir hier auch Null in der Varianz bei den anderen Verteilungen stehen.
{simstudy}. Jede Zeile beschreibt eine Variable in dem Datensatz und deren mathematischen Zusammenhang oder den Effekt untereinander. Dazu kommt noch die Varianz und die entsprechende Verteilung der Variable.
| varname | formula | variance | dist | link |
|---|---|---|---|---|
| age | 10 | 2 | normal | identity |
| female | -2 + age * 0.1 | 0 | binary | logit |
| jump_length | 1.5 - 0.2 * age + 0.5 * female | 0 | poisson | log |
Dann können wir die obige Tabelle auch einmal in {simstudy} nachbauen. Wir müssen dazu die Funktion defData() nutzen und dann immer erweitern. Dabei ist natürlich die Ordnung wichtig, da wir erst Variablen erschaffen müssen bevor wir mit den Variablen weiterrechnen können. Das verlangt etwas Übung und auch ein gewissen Gefühl für die Datenerstellung. Mir ist es am Anfang auch etwas schwerer gefallen, aber diese Art der Genierung hat echte Vorteile, wenn die Daten komplexer werden.
R Code [zeigen / verbergen]
def <-
defData(varname = "age", dist = "normal",
formula = 10, variance = 2) |>
defData(varname = "female", dist = "binary",
formula = "-2 + age * 0.1", link = "logit") |>
defData(varname = "jump_length", dist = "poisson",
formula = "1.5 - 0.2 * age + 0.5 * female", link = "log")Dann können wir schon die Funktion genData() nutzen um uns dann entsprechend viele Beobachtungen mit der entsprechenden Datendefinition zu erschaffen. Hier habe ich einmal fünf Beobachtungen gewäht, später nutze ich dann eher eintausend Beobachtungen.
R Code [zeigen / verbergen]
dt <- genData(n = 5, def)
dtKey: <id>
id age female jump_length
<int> <num> <int> <int>
1: 1 11.647845 0 0
2: 2 12.958943 0 1
3: 3 7.439934 0 1
4: 4 10.127518 1 0
5: 5 8.237693 0 0Manchmal ist es so, dass wir einem bestehenden Datensatz mit Vairablen eine Definition hinzufügen wollen. Das ist insbesondere bei Faktorenrastern der Fall. Dort bauen wir uns ja erst alle Faktorkombinationen und wollen dann nochmal den Messwert berechnen. Dafür können wir dann die Funktion defDataAdd() nutzen, die auch Variablennamen akzeptiert, die nicht noch nicht definiert wurden. Hier bauen wir uns eine binäre Sprungweite zusammen.
R Code [zeigen / verbergen]
def <- defDataAdd(varname = "jump_length_bin", formula = "-2 + 0.5*age + 0.5*female",
dist = "binary", link = "logit")Da wir bis jetzt nur die Defintion haben, müssen wir noch die Daten mit der Datendefinition zusammenbringen. Die Funktion addColumn() verbindet einen bestehenden Datensatz mit einer neuen Definition und erschafft dann die neue Variable. Wir werden dieses Vorgehen dann häufiger bei den faktoriellen Daten nutzen.
R Code [zeigen / verbergen]
addColumns(def, dt)Key: <id>
id age female jump_length jump_length_bin
<int> <num> <int> <int> <int>
1: 1 11.647845 0 0 1
2: 2 12.958943 0 1 1
3: 3 7.439934 0 1 0
4: 4 10.127518 1 0 1
5: 5 8.237693 0 0 1Wenn dich die Datengenerierugn und das Drumrum interessiert, dann kannst du auch auf dem Blog ouR data generation – meditations on data generating processes, R, and statistics, welches die Themen rund um das R Paket {simstudy} aufgreift einmal weiterlesen. Leider muss man sich mühsam durch die Chronik klicken, da es keine Sortierung nach Schlagworten oder ein Inhaltsverzeichnis gibt. Dafür ist da die eine oder andere spannende Idee darunter. Wenn ich was gefunden habe, habe ich es weiter unten in den Abschnitten verlinkt.
21.5 Gängige Messwerte
Im Folgenden wollen wir uns für die gängigen Verterilungsfamilien einmal die Generierung von Daten anschauen. Dabei bleibe ich länger bei der Normalverteilung und zeige dort einmal verschiedene Wege um auf künstliche Daten zu kommen. Dabei liegt auch hier der Fokus mehr auf dem faktoriellen Design, da wir es hier meistens mit etwas komplizierteren Generierungen zu tun bekommen. Nach der Normalverteilung gehe ich dann auf die Poissonverteilung und klassische Verteilung für Zähldaten ein. Dann kommt noch die ordinale und binomiale Verteilung dran. Dann haben wir die häufigsten Verteilungen besprochen. Mehr findet sich dann auch in der Vingette zu dem R Paket {simstudy}.
21.5.1 Normalverteilt
Beginnen wir mit der wichtigsten Art der Simulation von Messwerten. Wir fangen mit der Normalverteilung an. Das heißt, dass unser Messwert \(y\) normalverteilt ist und wir unsere Messwerte aus mehreren Normalverteilungen zusammenbauen. Jede der Faktoren oder Kovariaten ist normalverteilt und hat damit immer einen Mittelwert und eine Varianz als Streuung. Das macht es dann auch für uns einfacher einmal die Grundlagen zu verstehen. Am Ende kannst du dir auch mittlere Noten oder eine mittlere Anzahl generieren und dann eben etwas stärker Runden. Dazu mehr dann in den folgenden Verteilungen.
Faktorielles Design
Ein faktorielles Design heißt nichts anderes als das du einen Faktor mit Leveln vorliegen hast. Häufig ist dieser Faktor dann deine Behandlung und deine Level sind dann die Stufen der Behandlung. Wenn du nur zwei Behandlungen vorliegen hast, dann rechnest du dann später einen Zweigruppenvergleich wie eben den t-Test. Wenn du mehr als zwei Gruppen vorliegen hast, wirst du dann eine einfaktorielle ANOVA nutzen und den entsprechenden Post-hoc Test. Weil zwei Gruppen eben so schön einfach sind, fangen wir hier damit einmal an. An den zwei Gruppen kannst du dann immer schön die Grundlagen nachvollziehen.
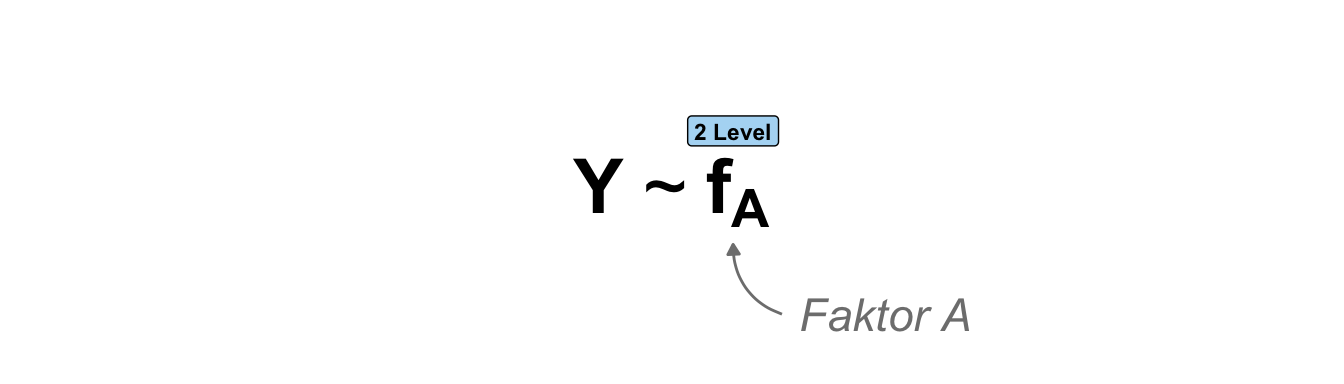
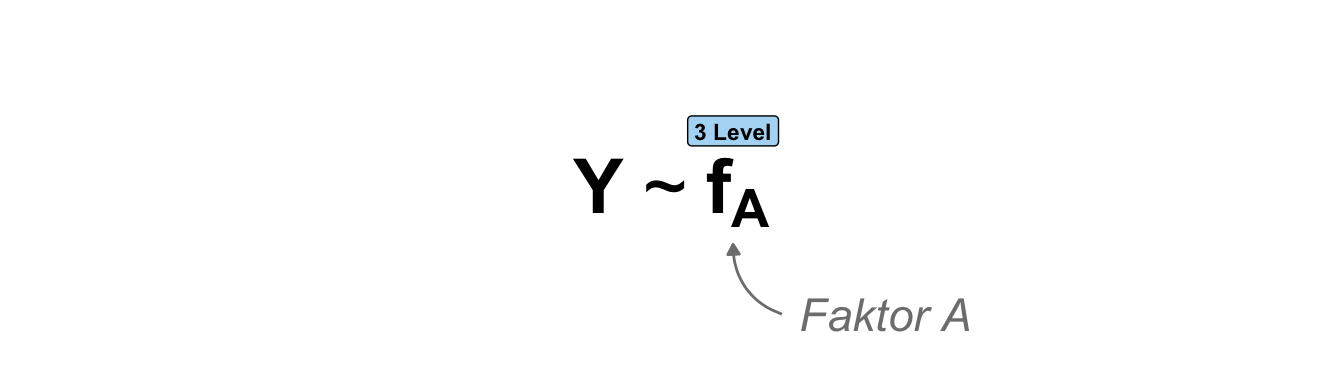
Einfaktoriell \(f_A\) mit 2 Leveln
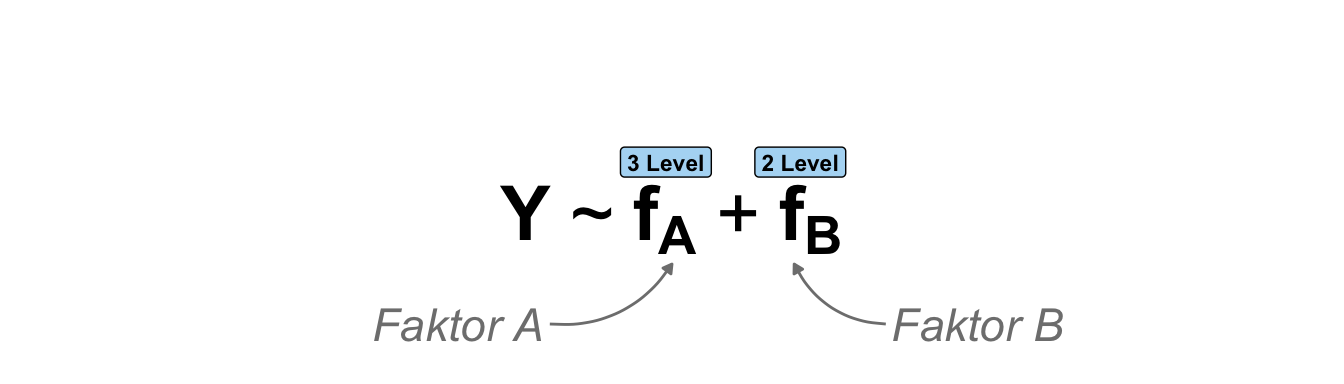
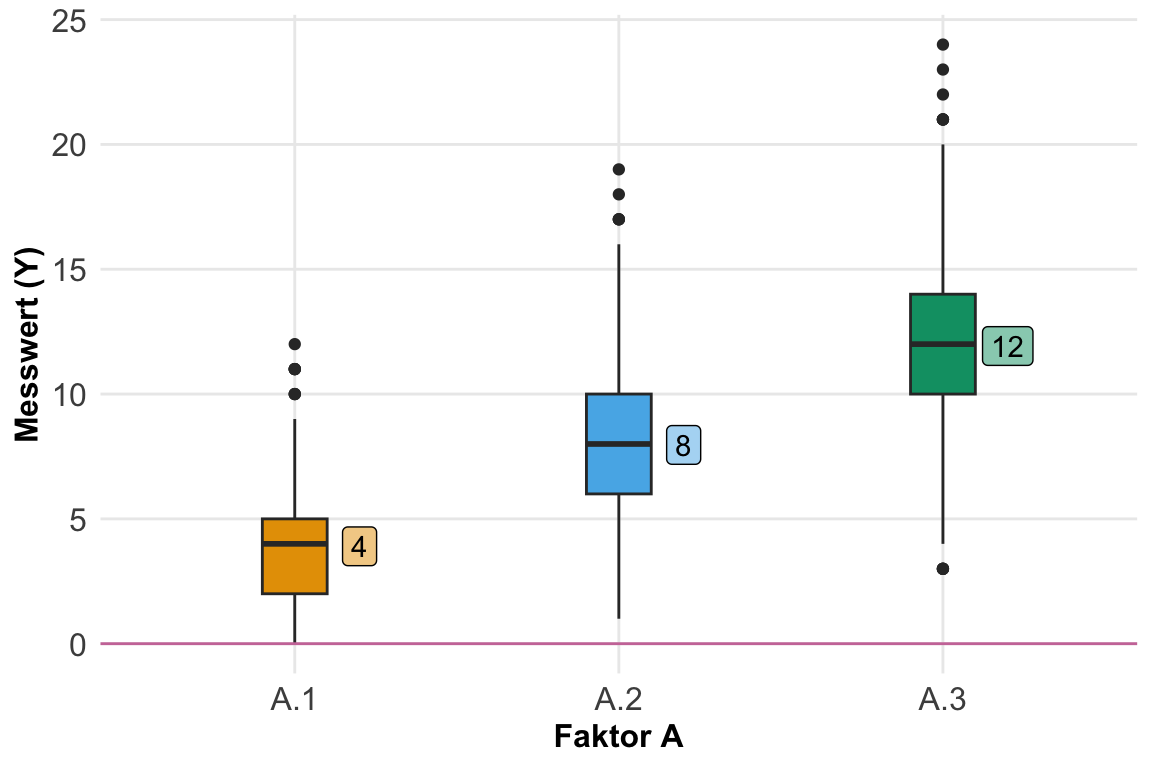
Beginnen wir also einmal mit dem einfaktoriellen Design mit nur zwei Gruppen. In der folgenden schematischen Darstellung ist einmal das Modell dargestellt. Wir haben einen Messwert und einen Faktor, der aus zwei Gruppen besteht. Das Modell ist sehr simple und soll uns nur helfen einmal die Simulation von Daten in R zu verstehen.
Wir haben jetzt unzählige Möglichkeiten die Daten zu generieren. Ich zeige hier einmal die zwei einfacheren Methoden. Einmal können wir die Daten getrennt in einem tibble() erstellen. Hierbei ist es wichtig, dass wir ein balanciertes Design vorliegen haben. Dann bauen wir uns das Long-Format über gather(). Wir schauen uns immer Gruppen mit gleicher Fallzahl an. Dann können wir auch die Spalten getrennt zusammenbauen und am Ende alles einmal aufaddieren. Wir haben hier einen Effekt von einer Mittelwertsdifferenz von \(+3\) vorliegen bei keiner Streuung.
Wenn wir wenig Gruppen haben, dann ist die getrennte Erstellung in einem tibble() sehr übersichtlich. Wir stellen ja den Effekt jeweils in den einzelnen Gruppen separat ein.
R Code [zeigen / verbergen]
fac1_tbl <- tibble("1" = rnorm(n = 5, mean = 3, sd = 0.001),
"2" = rnorm(n = 5, mean = 6, sd = 0.001)) |>
gather(key = "f_a", value = "y")Im additiven Zusammenbauen nutzen wir viele Funktionen um uns die Spalten mit den Faktoren, Effekten und Fehler aufzubauen. Am Ende addieren wir die Spalten zum Messwert auf.
R Code [zeigen / verbergen]
fac1_tbl <- tibble(f_a = gl(2, 5, labels = c("1", "2")),
eff_a = rep(c(3, 6), each = 5),
error = rnorm(length(f_a), 0, 0.001),
y = eff_a + error)Dann rechnen wir einfach einmal einen schnellen t-Test und schauen, ob wir die Differenz wieder erhalten. Wir sehen, dass wir die gleichen Mittelwerte wiedergeben bekommen und natürlich auch einen signifikanten Unterschied. Die Streuung ist ja super klein.
R Code [zeigen / verbergen]
t.test(y ~ f_a, data = fac1_tbl)
Welch Two Sample t-test
data: y by f_a
t = -4231.5, df = 7.0551, p-value < 2.2e-16
alternative hypothesis: true difference in means between group 1 and group 2 is not equal to 0
95 percent confidence interval:
-3.001890 -2.998542
sample estimates:
mean in group 1 mean in group 2
2.999996 6.000212 Wir können uns den Zusammenhang auch als eine simple lineare Regression darstellen lassen. Da ich die Streuung sehr gering eingestellt habe, können wir auch die Koeffizienten exakt so wieder erhalten. In der folgenden linearen Regression siehst du einmal wie die Mittelwertsdifferenz wieder der eingestellten Differenz entspricht.
R Code [zeigen / verbergen]
lm(y ~ f_a, fac1_tbl) |> coef() |> round(2)(Intercept) f_a2
3 3 In der folgenden Abbildung ist der Zusammenhang nochmal graphisch dargestellt. Wir haben den Mittelwert der ersten Gruppe als Intercept im Modell und die Steigung der Graden als Mittelwertsdifferenz. Wir sehen hier also nochmal den Zusammenhang zwischen der Datengenerierung und der linearen Regression.
Manchmal wollen wir uns auch Daten erstellen, die nicht die gleiche Varianz in allen Gruppen haben. In diesem Fall hilft am meisten die getrennte Erstellung. Wir nehmen dann einfach in jeder Gruppe eine andere Varianz und lassen dann die Daten erstellen. In dem folgenden Kasten siehst du nochmal ein Beispiel für die Generierung von Daten mit Varianzheterogenität. Ob wir das brauchen hängt dann davon ab, was du mit den simulierten Daten ausprobieren möchtest.
In der getrennten Erstellung von Daten können wir dann für jede Gruppe eine eigene Varianz festlegen. Hier habe ich in der einen Gruppe eine Standardabweichung von \(5\) und in der anderen Gruppe eine Standardabweichung von \(1\) angenommen. Ich brauche diese Art der Erstellung für den Welch t-Test oder aber in {emmeans} und dem multiplen Testen.
R Code [zeigen / verbergen]
fac1_tbl <- tibble("1" = rnorm(n = 5, mean = 3, sd = 5),
"2" = rnorm(n = 5, mean = 6, sd = 1)) |>
gather(key = "f_a", value = "y")Einfaktoriell \(f_A\) mit >2 Leveln
Kommen wir jetzt zu den etwas spannenderen Teil. Wir haben jetzt ein faktorielles Design mit einem Faktor und drei Leveln vorliegen. Ich habe dir einmal das Modell in der folgenden Abbildung einmal schematisch dargestellt. Wir würden hier also Daten bauen, die wir dann mit einer einfaktoriellen ANOVA oder aber dann mit einem Post-hoc Test wie {emmeans} auswerten würden. Der Messwert hier folgt daher einer Normalverteilung.
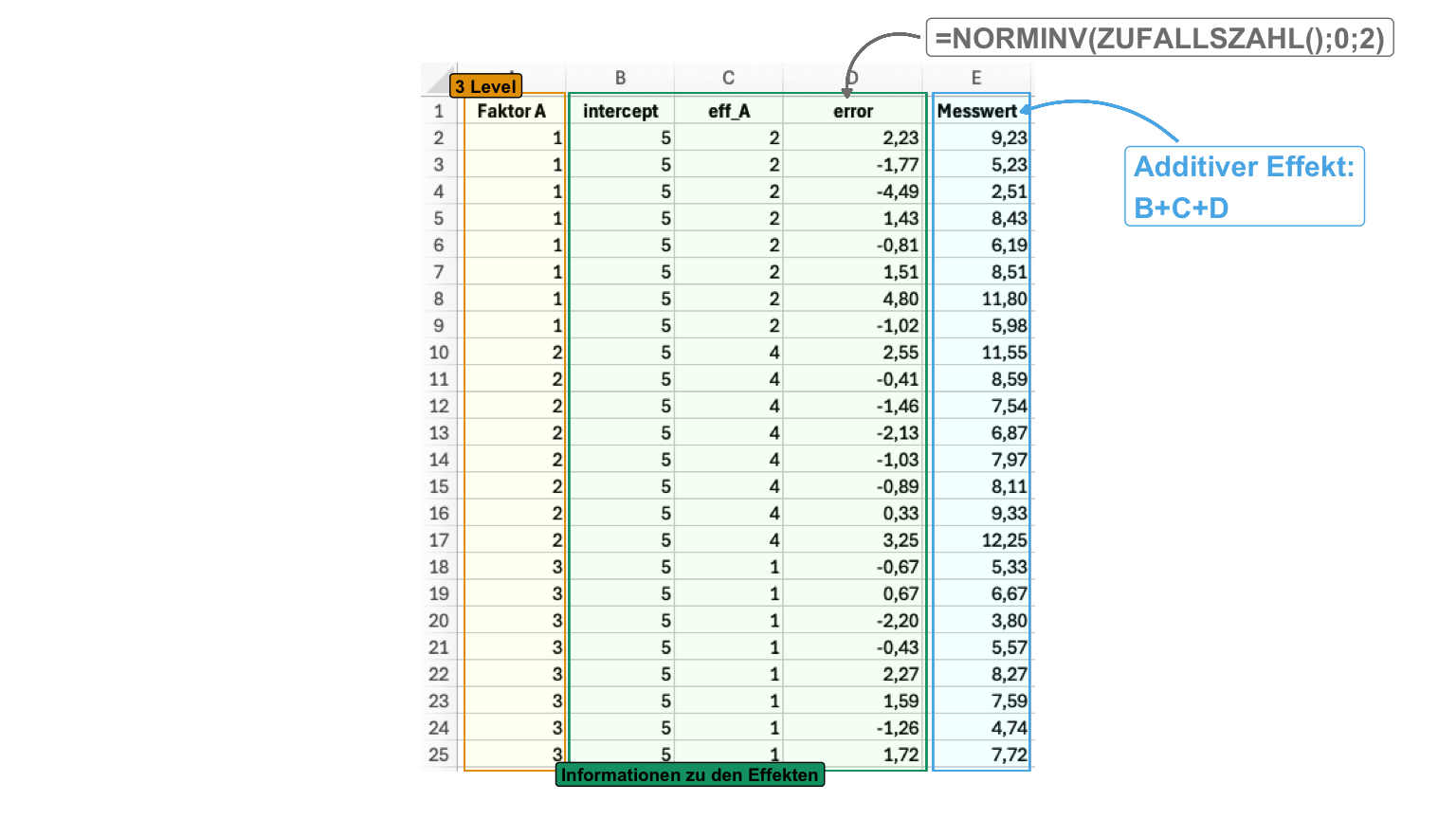
Wir können die Daten einmal getrennt bauen und hätten dann auch einfach die Möglichkeit die Varianzen auch unterschiedlich zu machen. Beachte immer, dass ich hier extrem geringe Varianzen mit \(0.001\) eingestellt habe um dann bei der geringen Fallzahl auch auf die voreingestellten Effekte zu kommen. Du kannst dir das Modell auch additiv bauen, dann wird es aber etwas komplexer und der Fehler ist dann homogen, wenn du nicht den Fehlervektor nochmal aufbrechen willst. Dann zeige ich dir die Generation der Daten auch nochmal in Excel.
Getrennt
Wenn wir wenig Gruppen haben, dann ist die getrennte Erstellung in einem tibble() sehr übersichtlich. Wir stellen ja den Effekt jeweils in den einzelnen Gruppen separat ein. Wir bauen hier alles um die Mittelwerte auf.
R Code [zeigen / verbergen]
fac1_tbl <- tibble("1" = rnorm(5, 2, 0.001),
"2" = rnorm(5, 6, 0.001),
"3" = rnorm(5, 4, 0.001)) |>
gather(key = "f_a", value = "y")Additiv
Im additiven Zusammenbauen nutzen wir viele Funktionen um uns die Spalten mit den Faktoren, Effekten und Fehler aufzubauen. Am Ende addieren wir die Spalten zum Messwert auf. Hier sind natürlich heterogene Varianzen schwerer darzustellen, da wir den Fehler für jedes Level einzeln generieren müssten. Auch hier wähle ich die Mittelwerte zuerst.
R Code [zeigen / verbergen]
fac1_tbl <- tibble(f_a = gl(3, 5, labels = c("1", "2", "3")),
eff_a = rep(c(2, 6, 4), each = 5),
error = rnorm(length(f_a), 0, 0.001),
y = eff_a + error)Effekt
Jetzt können wir aber das Ganze auch von der Effektseite aufziehen. Wir legen also fest, wie stark sich die Mittelwerte unterscheiden sollen anstatt die Mittelwerte selber. Daher eben der Effekt als Ausgangspunkt für unsere Datengenerierung. Als erstes bauen wir uns aber einmal den Datensatz nur für den Faktor. Die Spalte rep gibt an wie viele Wiederholungen wir pro Level haben.
R Code [zeigen / verbergen]
fac1_tbl <- expand_grid(f_a = 1:3, rep = 1:5) |>
mutate(f_a = as_factor(f_a))Dann nutzen wir den Faktor um eine Dummykodierung zuerstellen. Mit der Dummykodierung können wir dann den Leveln des Faktors die Effekte zuordnen.
R Code [zeigen / verbergen]
fac1_mat <- fac1_tbl |>
model_matrix(~ f_a) |>
as.matrix()Jetzt brauchen wir die Effekte. Wir haben hier den Mittelwert der ersten Gruppe in den Intercept und dann die Effekte danach. Daher hat das zweite Level einen um 4 höheren Mittelwert. Das dritte Level ist dann nur um 2 größer als der Intercept.
R Code [zeigen / verbergen]
eff_vec <- c(intercept = 2,
f_a2 = 4, f_a3 = 2)Dann kombinieren wir die Modellmatrix mit den Information wo der Effekt angewendet werden soll mit dem Vektor der Effekte. Wir erhalten dann unsere Messwerte in dem Vektor y_raw. Hier sind natürlich pro Faktor die Werte immer gleich. Wir haben ja noch keine Streuung hinzugefügt.
R Code [zeigen / verbergen]
y_vec <- fac1_mat %*% eff_vecAm Ende erhalten wir dann die echten generierten Messwerte indem wir zu unserem Messwert noch einen Fehler addieren. Ich nehme hier einen sehr kleinen Fehler, damit wir die Effekt auch gleich wiedersehen. Sonst müsste ich eine sehr hohe Fallzahl simulieren.
R Code [zeigen / verbergen]
fac1_tbl <- fac1_tbl |>
mutate(y = y_vec + rnorm(length(y_vec), 0, 0.001))Welche der drei Varianten du dann nutzt hängt davon ab, wozu du die generierten Datem brauchst. Jede Variante zeigt eben nochmal anders wie Daten entstehen.
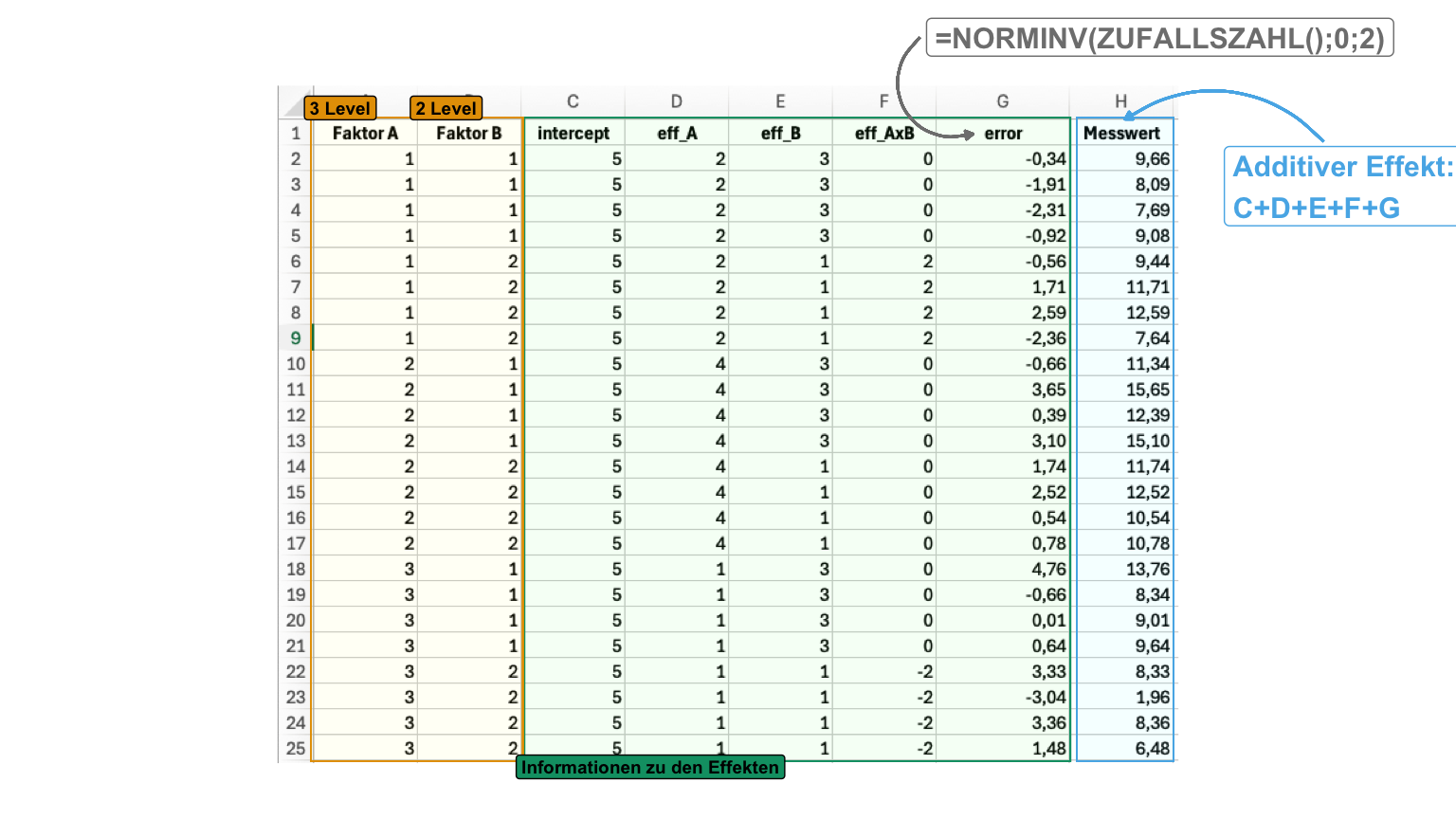
Die Erstellung von Daten in Excel ist nicht für die wissenschaftliche Bewertung von Algorithmen sinnvoll und angebracht. Wir nutzen hier Excel um recht einfach uns zufällig Daten zu erstellen. Wir nutzen dann die künstlichen Exceldaten um unsere Analyse für unsere echten experimentelle Daten zu üben. Auch ist die vereinfachte Erstellung von künstlichen Daten in Excel hilfreich um die zugrundeliegende Konzepte nochmal zu verstehen.
In der folgenden Datei findest du dann einmal die Generierung der einfaktoriellen Daten in Excel. Hier müssen wir dann das additive Zusammenbauen nutzen. Deshalb ist die Datei etwas größer. Am Ende brauchen wir die Spalte Faktor A und die Spalte Messwert.
R Code [zeigen / verbergen]
excel_fac1_tbl <- read_excel("data/data_generation_excel.xlsx",
sheet = "einfaktoriell") |>
clean_names() |>
mutate(faktor_a = as_factor(faktor_a))In der folgenden Abbildung habe ich dir einmal normalverteilte Messwerte in Excel aus einem einfaktoriellen Design generiert. Zuerst legen wir den allgemeinen Intercept fest, dann noch die Effekte der einzelnen Gruppen. Am Ende brauchen wir noch den Fehler oder die Streuung mit der Funktion NORMINV(). Hier habe ich eine Standardabweichung von \(2\) voreingestellt.
Dann schauen wir einmal, ob wir dann die Mittelwerte und die entsprechenden Differenzen auch in {emmeans} wiederfinden. Die Funktion pwpm() gibt uns auf der Diagonalen die Mittelwerte wieder und dann die Differenzen in der unteren Hälfte der Matrix. Damit haben wir dann auch alle Informationen auf einmal zusammen. Die Mittelwerte sind die voreingestellten und die Effekte sind ebenfalls richtig.
R Code [zeigen / verbergen]
lm(y ~ f_a, fac1_tbl) |>
emmeans(~ f_a) |>
pwpm() 1 2 3
1 [1.9999] <.0001 <.0001
2 -3.9997 [5.9996] <.0001
3 -1.9995 2.0001 [3.9994]
Row and column labels: f_a
Upper triangle: P values adjust = "tukey"
Diagonal: [Estimates] (emmean)
Lower triangle: Comparisons (estimate) earlier vs. laterWir können uns dann das Modell auch nochmal in dem linearen Kontext anschauen. Hier haben wir dann aber zwei Möglichkeiten die Referenz des Intercepts zu setzen. In der klassischen Kodierung setzen wir den Intercept auf den Mittelwert des ersten LEvels oder eben der ersten Gruppe. Wir nennen diese Kodierung dann Treatment coding. Dafür müssen wir nichts machen, dass ist die Standardeinstellung. Wir können aber auch das Effect coding wählen, dann liegt der Intercept auf dem globalen Mittelwert. Was ja eher der Interpretation der ANOVA entspricht. Es gibt natürlich noch weitere Kodierungen, wie du im Tutorial R Library Contrast Coding Systems for categorical variables nachlesen kannst.
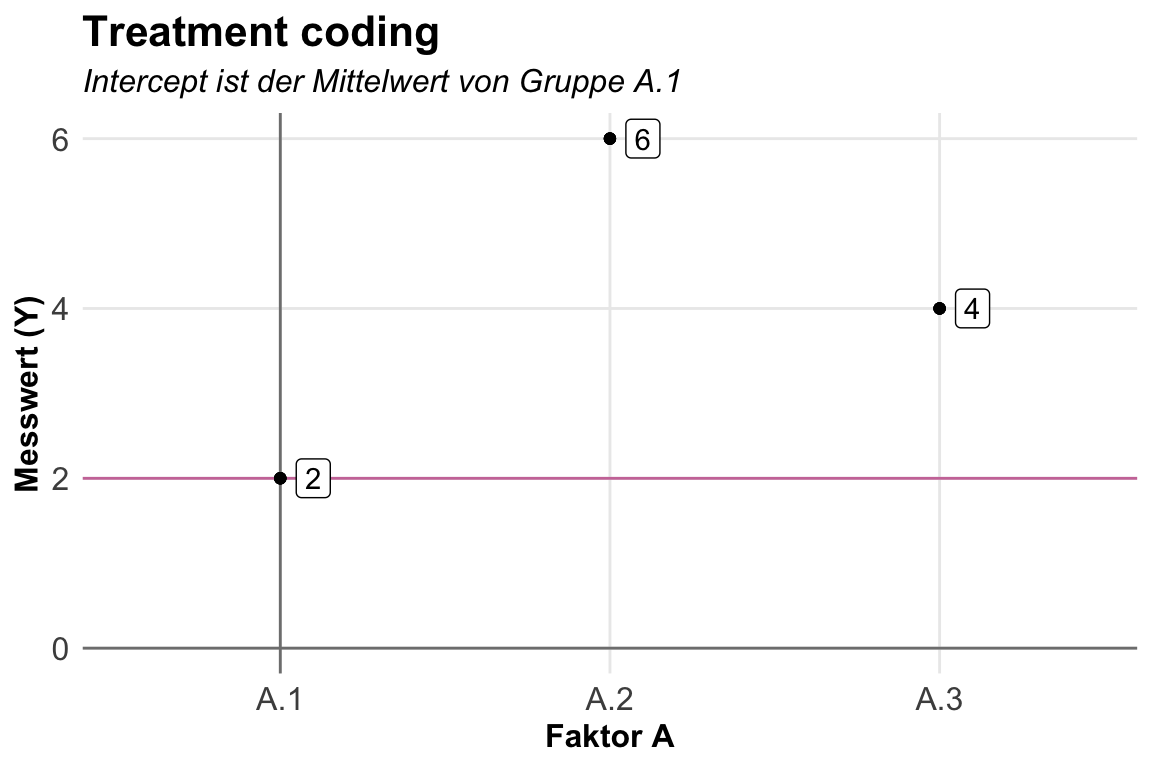
Der Standard in der linearen Modellierung von Faktoren ist das Treatment coding mit contr.treatment in der Funktion lm(). Wir müssen da auch eigentlich nichts ändern, aber ich habe es hier einmal angeben damit du siehst, wo du die Kodierung ändern musst. Jeder Faktor muss einzeln geändert werden.
R Code [zeigen / verbergen]
lm(y ~ f_a, fac1_tbl, contrasts = list(f_a = "contr.treatment")) |>
coef() |> round(2)(Intercept) f_a2 f_a3
2 4 2 Wir erhalten dann aber die zu erwartenden Koeffizienten wieder. Der Mittelwert der ersten Gruppe ist der Intercept. Die Koeffizienten sind die Änderungen der anderen Level zum Intercept. Ich habe dir den Zusammenhang nochmal in der folgenden Abbildung dargestellt. Wir sehen hier sehr schön, dass eben das erste Level \(A.1\) den Intercept ausmacht. Die Änderungen als Mittelwertsdifferenzen zum Intercept sind die Koeffizienten in dem linearen Modell.
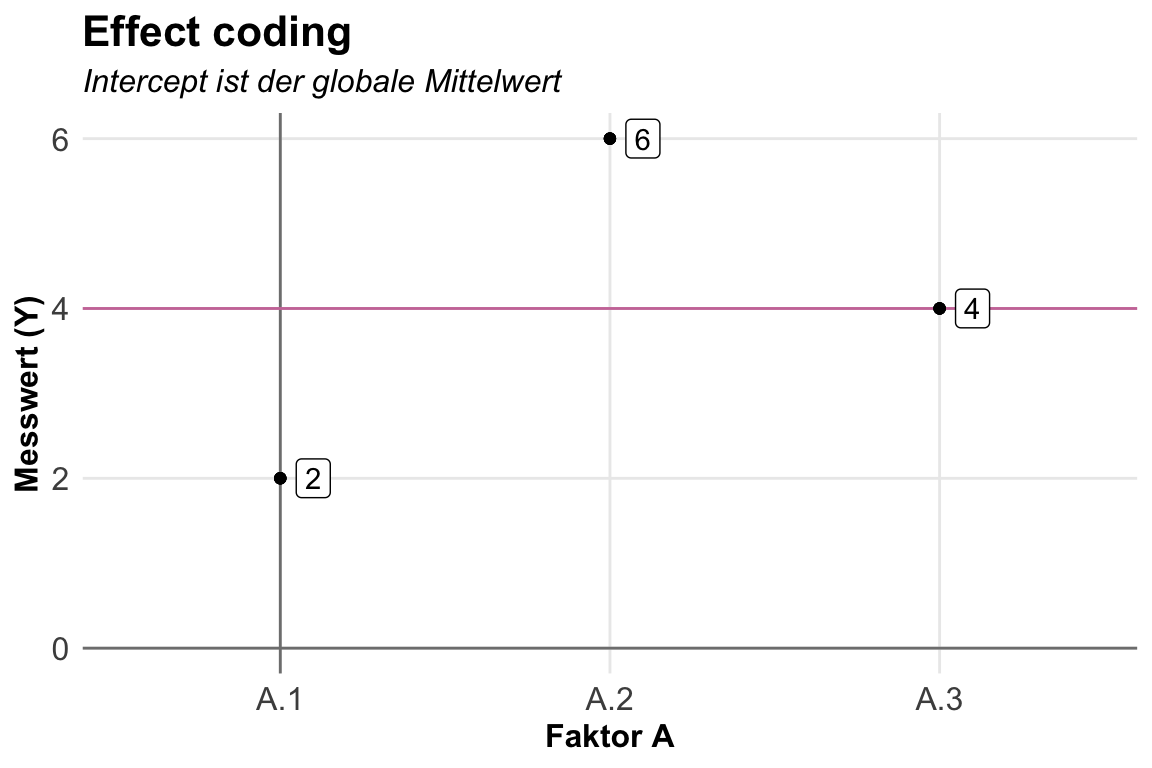
Das Effect coding können wir mit der Option contr.sum in der Funktion lm() nutzen. Wir müssen dies für jeden einzelnen Faktor einstellen. Ein Mischmasch würde ich stark vermeiden wollen. Wir haben dann als Intercept das globale Mittel des Messwerts über alle Gruppen. Die Koeffizienten sind dann die Mittelwertsdifferenzen zum globalen Mittel. Das letzte Level fehlt und lässt sich aus den vorherigen Leveln berechnen, da die Summe der Koeffizienten Null sein muss. In unserem Fall wäre die Mittelwertsdifferenz dann auch \(0\), da sich die beiden vorherigen Level schon auf Null aufaddieren.
R Code [zeigen / verbergen]
lm(y ~ f_a, fac1_tbl, contrasts = list(f_a = "contr.sum")) |>
coef() |> round(2)(Intercept) f_a1 f_a2
4 -2 2 Der Zusammenhang wird dann in der folgenden Darstellung nochmal klarer. Wir haben hier den globalen Mittelwert von \(4\) über alle Gruppen. Die Koeffizienten sind jetzt die Abweichung der lokalen Mittelwerte zu dem globalen Mittel. Eigentlich wie wir es auch aus einer einfaktoriellen ANOVA erwarten würden.
Zweifaktoriell ohne Interaktion \(f_A + f_B\)
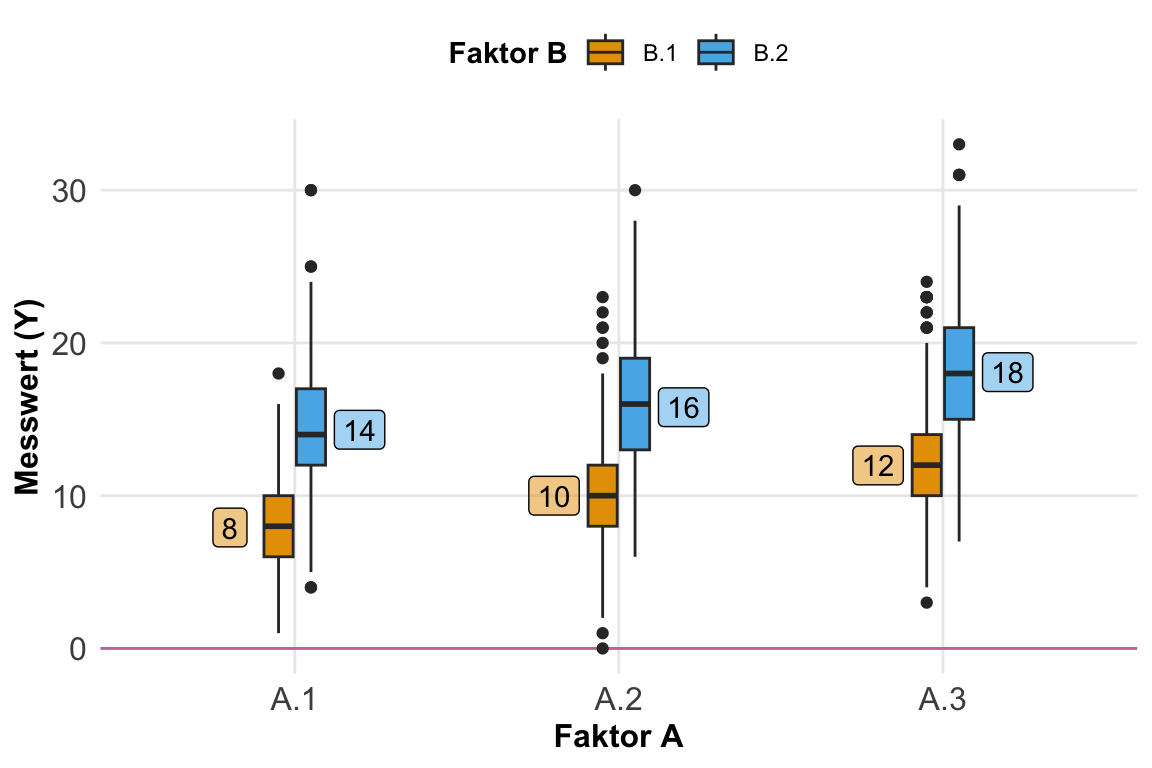
Sehr häufig haben wir aber mehrfaktorielle Designs vorliegen. Selten betrachten wir nur einen Faktor. Daher habe ich jetzt einmal ein zweifaktorielles Design in der folgenden schematischen Darstellung mitgebracht. Wir haben hier zwei Faktoren. Der Hauptfaktor \(f_A\) hat drei Level und der zweite Faktor \(f_B\) hat zwei Level. Der Messwert ist weiterhin normalverteilt. Jetzt müssen wir uns gleich etwas strecken um die Daten zusammenzubauen. Wir haben aber keine Interaktion zwischen den Faktoren vorliegen. Das schauen wir uns dann gleich nochmal getrennt an.
Da wir keine Interaktion vorliegen haben und auch Varianzhomogenität kann ich mir die Daten wie folgt bauen. Zuerst baue ich mir alle Faktorenkombinationen mit fünf Wiederholungen pro Kombination mit der Funktion expand_grid() dann kann ich mir den Messwert bauen indem ich immer den gleichen Effekt für jedes Level aufaddieren. Wir haben hier ja keine Interaktion vorliegen. Die Effekte steigen linear an. Darüber hinaus habe ich auch keine Streuung eingestellt, da mit wir bei der kleinen Fallzahl auch gleich unsere Effekte wiedersehen.
R Code [zeigen / verbergen]
fac2_nointer_tbl <- expand_grid(f_a = 1:3,
f_b = 1:2,
rep = 1:5) |>
mutate(y = 2 +
2 * f_a +
3 * f_b +
rnorm(15, 0, 0.001)) |>
mutate(f_a = as_factor(f_a),
f_b = as_factor(f_b))Dann schauen wir einmal, ob wir dann die Mittelwerte und die entsprechenden Differenzen auch in {emmeans} wiederfinden. Die Funktion pwpm() gibt uns auf der Diagonalen die Mittelwerte wieder und dann die Differenzen in der unteren Hälfte der Matrix. Damit haben wir dann auch alle Informationen auf einmal zusammen. Das wird dann doch recht groß und dann teilweise etwas unübersichtlich. Schaue gleich mal in der unteren Abbildung, da wird vermutlich alles etwas klarer.
R Code [zeigen / verbergen]
lm(y ~ f_a + f_b, fac2_nointer_tbl) |>
emmeans(~ f_a * f_b) |>
pwpm() 1 1 2 1 3 1 1 2 2 2 3 2
1 1 [ 7.0004] <.0001 <.0001 <.0001 <.0001 <.0001
2 1 -1.99988 [ 9.0003] <.0001 <.0001 <.0001 <.0001
3 1 -3.99969 -1.99981 [11.0001] <.0001 <.0001 <.0001
1 2 -3.00000 -1.00012 0.99969 [10.0004] <.0001 <.0001
2 2 -4.99988 -3.00000 -1.00019 -1.99988 [12.0003] <.0001
3 2 -6.99969 -4.99981 -3.00000 -3.99969 -1.99981 [14.0001]
Row and column labels: f_a:f_b
Upper triangle: P values adjust = "tukey"
Diagonal: [Estimates] (emmean)
Lower triangle: Comparisons (estimate) earlier vs. laterDa wir es hier auch wieder mit einem linearen Modell zu tun haben, können wir uns die Sachlage auch einmal mit den Koeffizienten des linearen Modells anschauen. Wir haben hier einen linearen Anstieg in unserem ersten Faktor und einen Unterschied von \(3\) in unserem zweiten Faktor. Der Intercept ist dabei die Faktorkombination \(A.1.B.1\) als niedrigste Levelkombination. Wir schauen uns hier nur das Treatment coding an.
R Code [zeigen / verbergen]
lm(y ~ f_a + f_b, fac2_nointer_tbl) |>
coef() |> round(2)(Intercept) f_a2 f_a3 f_b2
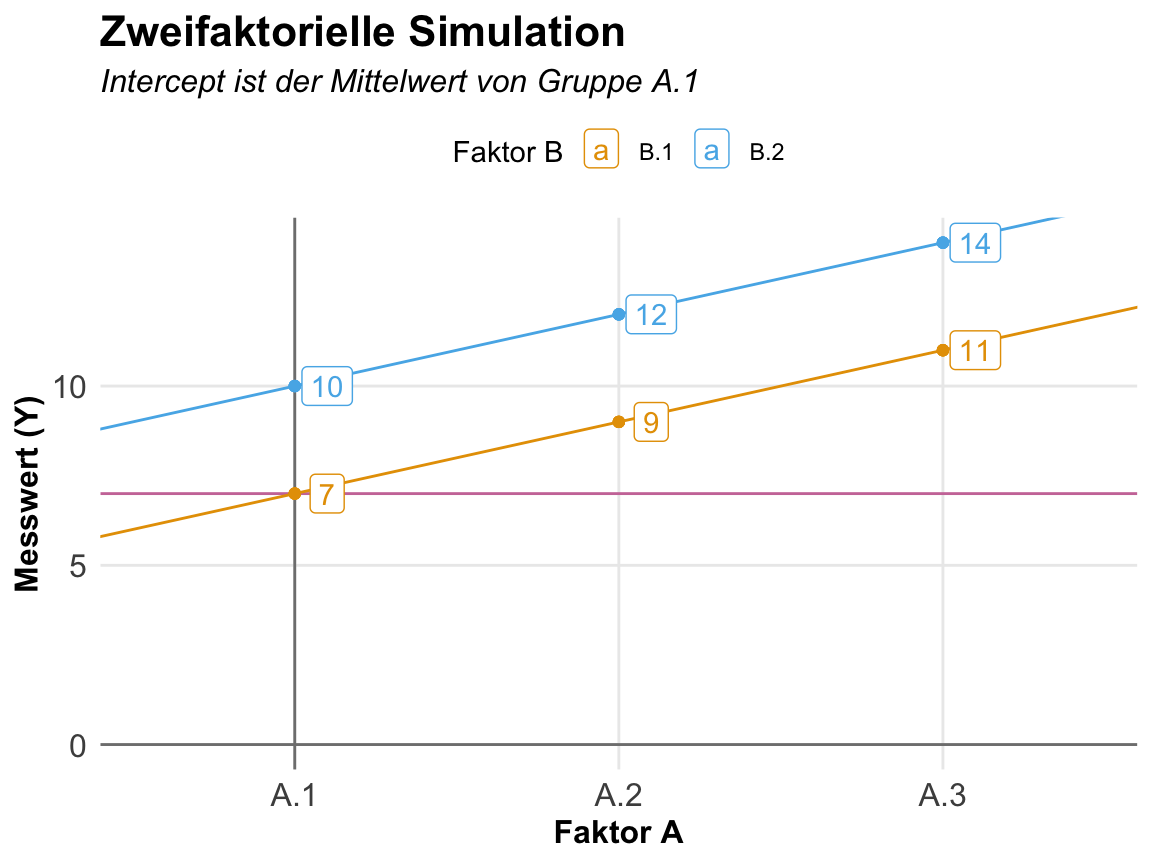
7 2 4 3 In der folgenden Abbildung siehst du dann einmal den Zusammenhang und die Koeffizienten visualisiert. Der Intercept liegt auf der Faktorkombination \(A.1.B.1\). Die Level von \(B.2\) sind parallel um den Koeffizienten von \(B.2\) verschoben. Der Anstieg ist aber immer noch \(+2\) da wir ja hier keine Interaktion vorliegen haben. Über den Faktor \(A\) verhält sich der Faktor \(B\) gleich. Hätten wir eine Interaktion vorliegen, dann würden wir keinen parallelen Verlauf sehen. In der Praxis beobachten wir solche Daten eher selten sondern sehen immer eine leichte Interaktion. Insbesondere da wir hier ja keine Streuung in den Daten vorliegen haben.
Zweifaktoriell mit Interaktion \(f_A + f_B + f_A \times f_B\)
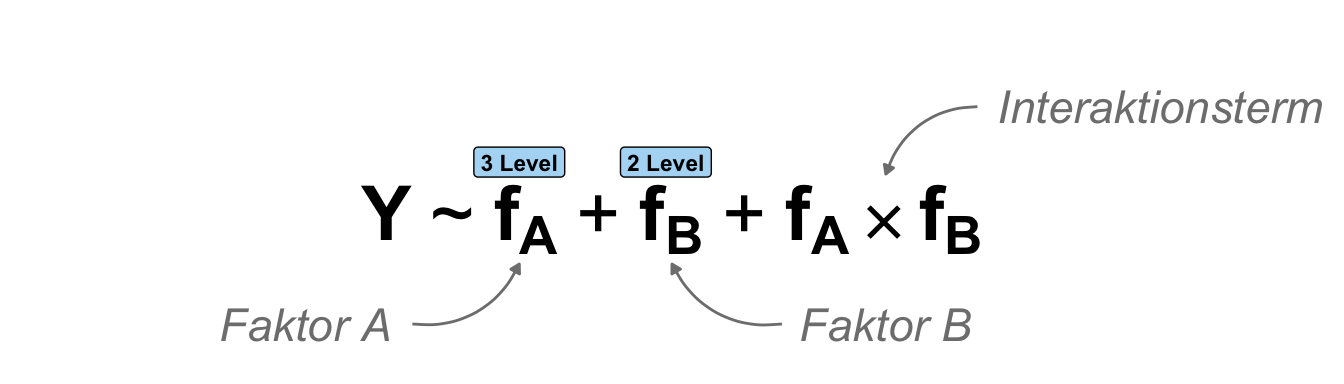
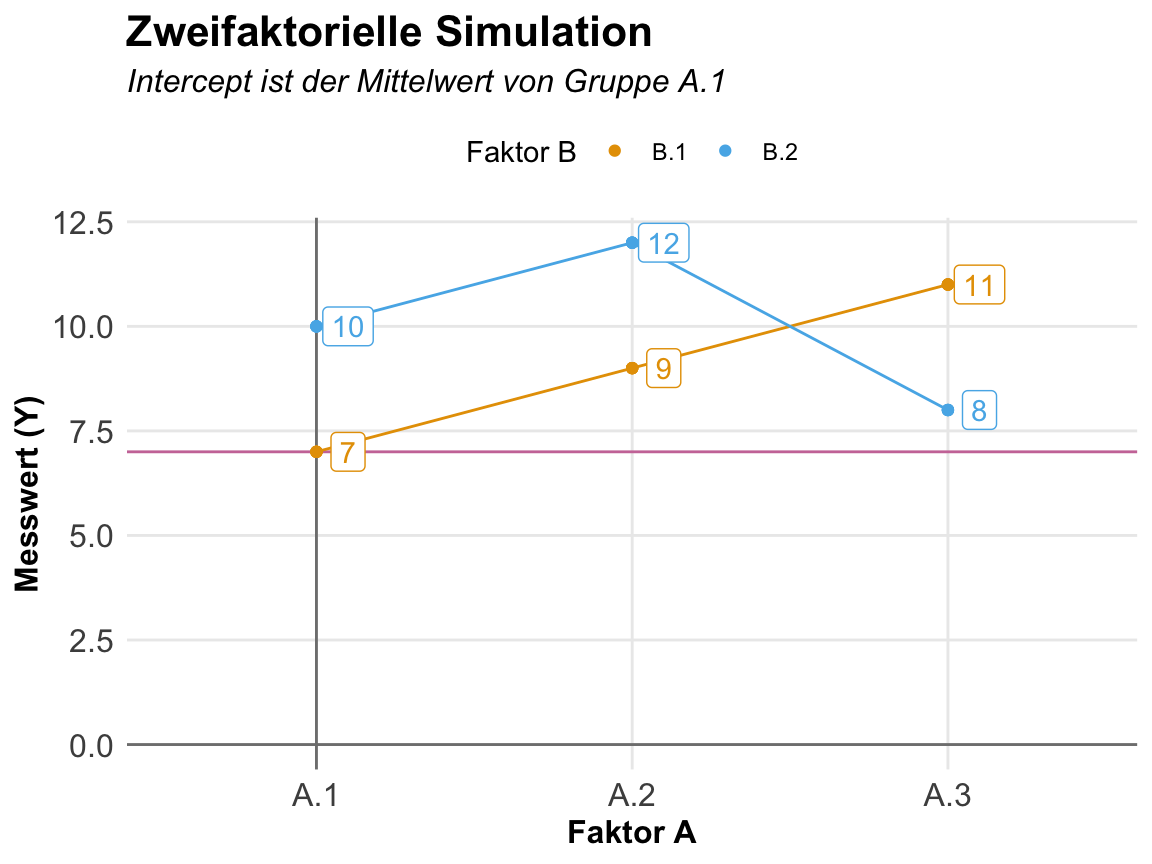
Selten betrachten wir nur einen Faktor sondern eben zwei Faktoren. Daher habe ich jetzt einmal ein zweifaktorielles Design in der folgenden schematischen Darstellung mitgebracht. Wir haben hier zwei Faktoren. Der Hauptfaktor \(f_A\) hat drei Level und der zweite Faktor \(f_B\) hat zwei Level. Der Messwert ist weiterhin normalverteilt. Jetzt müssen wir uns gleich etwas strecken um die Daten zusammenzubauen. Wir haben nun nämlich endlich eine Interaktion zwischen den beiden Faktoren vorliegen. Der Faktor \(f_B\) verhält sich nicht gleich in allen Leveln des Faktors \(f_A\). Die Graden laufen nicht parallel.
In den beiden folgenden Tabs zeige ich dir einmal wie du die Daten für ein zweifaktorielles Design mit Interaktion in einem tibble() sowie in Excel zusammenbaust. In Excel ist es etwas übersichtlicher, da du ja dort die einzelnen Spalten händisch anlegst. Zum Verstehen vielleicht etwas besser als in R. Dennoch ist die Erstellung in R stabiler und nicht so fehleranfällig. Für mal eben zum probieren reicht natürlich wie immer auch Excel super aus.
Wenn wir mit Interaktionen einen Datensatz bauen, dann müssen wir den Datensatz über die Effekte aufbauen. Wir legen also fest, wie stark sich die Mitterlwerte unterscheiden sollen anstatt die Mittelwerte selber. Daher eben der Effekt als Ausgangspunkt für unsere Datengenerierung. Als erstes bauen wir uns aber einmal den Datensatz nur für die Faktoren. Die Spalte rep gibt an wie viele Wiederholungen wir pro Level haben.
R Code [zeigen / verbergen]
fac2_tbl <- expand_grid(f_a = 1:3, f_b = 1:2, rep = 1:5) |>
mutate(f_a = as_factor(f_a),
f_b = as_factor(f_b))Dann nutzen wir die Faktoren um eine Dummykodierung zuerstellen. Mit der Dummykodierung können wir dann die Level der Faktor und der Interaktion die Effekte zuordnen. Das ist hier super wichtig, da wir sonst kaum die Interaktion modellieren können.
R Code [zeigen / verbergen]
fac2_mat <- fac2_tbl |>
model_matrix(~ f_a * f_b) |>
as.matrix()Jetzt brauchen wir die Effekte für die Faktoren und die Interaktion. Wir haben hier den Mittelwert der ersten Gruppe aus dem Faktor \(f_A\) und dem ersten Level im Faktor \(f_B\) in den Intercept. Danach kommen dann die Effekte der anderen Level plus die Interaktion. Ich schreibe hier die Effekte der Interaktion mit dem x zwischen den Namen.
R Code [zeigen / verbergen]
eff_vec <- c(intercept = 7,
f_a2 = 2, f_a3 = 4, f_b2 = 3,
f_a2xf_b2 = 0, f_a3xf_b2 = -6)Dann kombinieren wir die Modellmatrix mit den Information wo der Effekt angewendet werden soll mit dem Vektor der Effekte. Wir erhalten dann unsere Messwerte in dem Vektor y_raw. Hier sind natürlich pro Faktorkombination die Werte immer gleich. Wir haben ja noch keine Streuung hinzugefügt.
R Code [zeigen / verbergen]
y_raw <- fac2_mat %*% eff_vecAm Ende erhalten wir dann die echten generierten Messwerte indem wir zu unserem Messwert noch einen Fehler addieren. Ich nehme hier einen sehr kleinen Fehler, damit wir die Effekt auch gleich wiedersehen. Sonst müsste ich eine sehr hohe Fallzahl simulieren.
R Code [zeigen / verbergen]
fac2_inter_tbl <- fac2_tbl |>
mutate(y = y_raw + rnorm(length(y_raw), 0, 0.001))Wenn du jetzt noch Varianzheterogenität haben möchtest, dann müsstest du dir einen Vektor mit den verschiedenen Aufrufen der Funktion rnorm() für die jeweiligen Faktorkombinationen bauen. Hier wären es dann bei sechs Faktorkombinationen eben sechsmal rnorm() mit jeweils fünf Beobachtungen als Anzahl der Wiederholungen und dann eben einer eigenen Streuung.
Die Erstellung von Daten in Excel ist nicht für die wissenschaftliche Bewertung von Algorithmen sinnvoll und angebracht. Wir nutzen hier Excel um recht einfach uns zufällig Daten zu erstellen. Wir nutzen dann die künstlichen Exceldaten um unsere Analyse für unsere echten experimentelle Daten zu üben. Auch ist die vereinfachte Erstellung von künstlichen Daten in Excel hilfreich um die zugrundeliegende Konzepte nochmal zu verstehen.
In der folgenden Datei findest du dann einmal die Generierung der zweifaktoriellen Daten mit Interaktion in Excel. Hier müssen wir dann das additive Zusammenbauen nutzen, da wir keine Matrizen in Excel muliplizieren wollen. Deshalb ist die Datei etwas größer. Am Ende brauchen wir die Spalte Faktor A und die Spalte Faktor B sowie die Spalte Messwert.
R Code [zeigen / verbergen]
excel_fac2_tbl <- read_excel("data/data_generation_excel.xlsx",
sheet = "zweifaktoriell") |>
clean_names() |>
mutate(faktor_a = as_factor(faktor_a),
faktor_b = as_factor(faktor_b))In der folgenden Abbildung habe ich dir einmal normalverteilte Messwerte in Excel aus einem zweifaktoriellen Design mit Interaktion generiert. Zuerst legen wir den allgemeinen Intercept fest, dann noch die Effekte der einzelnen Gruppen sowie der Interaktion. Am Ende brauchen wir noch den Fehler oder die Streuung mit der Funktion NORMINV(). Hier habe ich eine Standardabweichung von \(2\) voreingestellt.
Die Zahlen kommen jetzt nicht perfekt hin, da ich hier mit einer Standardabweichung von 2 eine recht hohe Streuung gewählt habe. Darüber hinaus habe ich dann auch sehr wenige Beobachtungen gebaut. Deshalb passt der Intercept nicht ganz, den der Intercept müsste eigentlich 10 sein und nicht 9. Daher stimmen dann auch die anderen Effekt nicht perfekt. Aber so ist das dann manchmal mit künstlichen Daten
R Code [zeigen / verbergen]
lm(messwert ~ faktor_a * faktor_b, data = excel_fac2_tbl) |>
coef() |> round() (Intercept) faktor_a2 faktor_a3 faktor_b2
9 5 2 2
faktor_a2:faktor_b2 faktor_a3:faktor_b2
-4 -6 Diese Generierung in Excel eigentlich sich sehr gut um dir selber Daten zu bauen, wenn du schonmal testen wilst wie die Analyse laufen würde bevor du eigentlich echte Daten aus deinem Experiment erhoben hast. Hier kommt es ja nicht auf die perfekten Effekte an. Hauptsache es sind Daten, die nicht alle gleich sind und du nichts siehst. Daher kann ich dir diesen Weg der Datengenerierung zum Testen deines R Codes nur empfehlen.
Fangen wir mit der Überprüfung an und schauen dafür erst mal in eine zweifaktorielle ANOVA mit Interaktion. Wir wollen hier natürlich, dass alle Faktoren signifikant sind und ebenfalls die Interaktion. So haben wir ja die Daten oben auch gebaut.
R Code [zeigen / verbergen]
aov(y ~ f_a + f_b + f_a:f_b, fac2_inter_tbl) |> summary() Df Sum Sq Mean Sq F value Pr(>F)
f_a 2 20.00 10.001 9217080 <2e-16 ***
f_b 1 7.50 7.498 6910404 <2e-16 ***
f_a:f_b 2 60.01 30.004 27651951 <2e-16 ***
Residuals 24 0.00 0.000
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Das passt soweit, wir haben alles so wie wir es wollen. Leider sehen wir nicht die direkten Mittelwerte in einer ANOVA, daher hier nochmal emmeans() um uns die Mittelwerte wiedergeben zu lassen. Auch hier sehen wir, eine Menge Zahlen, aber soweit passen die Werte. Wir brauche in der Faktorkombination \(A.1.B.1\) einen Mittelwert von 7, da diese Gruppe ja unser Intercept ist. Das passt soweit. Die anderen Mittelwerte berechnen sich dann aus den obigen Effketen.
R Code [zeigen / verbergen]
lm(y ~ f_a + f_b + f_a:f_b, fac2_inter_tbl) |>
emmeans(~ f_a * f_b) f_a f_b emmean SE df lower.CL upper.CL
1 1 7.0001 0.000466 24 6.9992 7.0011
2 1 8.9999 0.000466 24 8.9990 9.0009
3 1 11.0003 0.000466 24 10.9993 11.0012
1 2 9.9998 0.000466 24 9.9989 10.0008
2 2 12.0003 0.000466 24 11.9993 12.0013
3 2 7.9999 0.000466 24 7.9989 8.0008
Confidence level used: 0.95 Dann schauen wir nochmal die Koeffizienten eines linearen Modells an um dann nochmal die Effekte zu sehen. Das ist natürlich alles etwas doppelt, aber es ist auch eine Übung zu verstehen woher die Zahlen kommen. Hier also einmal die Effekte und die Interaktionen für unser zweifaktorielles Design. Bitte beachte, dass sich natürlich das niedrigste Level des Faktors \(f_A\) und des Faktors \(f_B\) im Intercept vereinen. Ja, das ist manchmal echt nicht so einfach.
R Code [zeigen / verbergen]
lm(y ~ f_a + f_b + f_a:f_b, fac2_inter_tbl) |>
coef() |> round(2)(Intercept) f_a2 f_a3 f_b2 f_a2:f_b2 f_a3:f_b2
7 2 4 3 0 -6 In der folgenden Abbildung siehst du einmal die Interaktion in der Faktorkombination \(A.3.B2\). Hier haben wir einen Effekt von -6 vorliegen. Durch die Interaktion fällt der Mittelwert weit nach unten. Weiter als er eigentlich sein sollte, wenn wir einen linearen Anstieg über den Faktor \(f_A\) erwarten würden. Wir haben eben nicht eine klassische Verschiebung der Graden durch den Faktor \(f_B\) sondern gegeben durch die Interaktion schneiden sich die Graden.
Wir haben uns jetzt einmal durch das faktorielle Design durchgearbeitet. Das war jetzt auch bei einem normalverteilten Messwert relativ einfach. Wenn wir dann noch eine andere Verteilung im Messwert vorliegen haben, dann wird die Sache noch etwas komplizierter. In den folgenden Abschnitten gehe ich dann nicht für jede Verteilung auf alle Kombinationen ein, sondern nutze immer die wichtigsten faktoriellen Designs, wie eben die zweifaktoriellen mit und ohne Interaktion. Die einfaktoriellen Designs mit mehr als zwei Leveln dienen eher der Veranschaulichung und werden in der Praxis kaum genutzt.
Kovariates Design
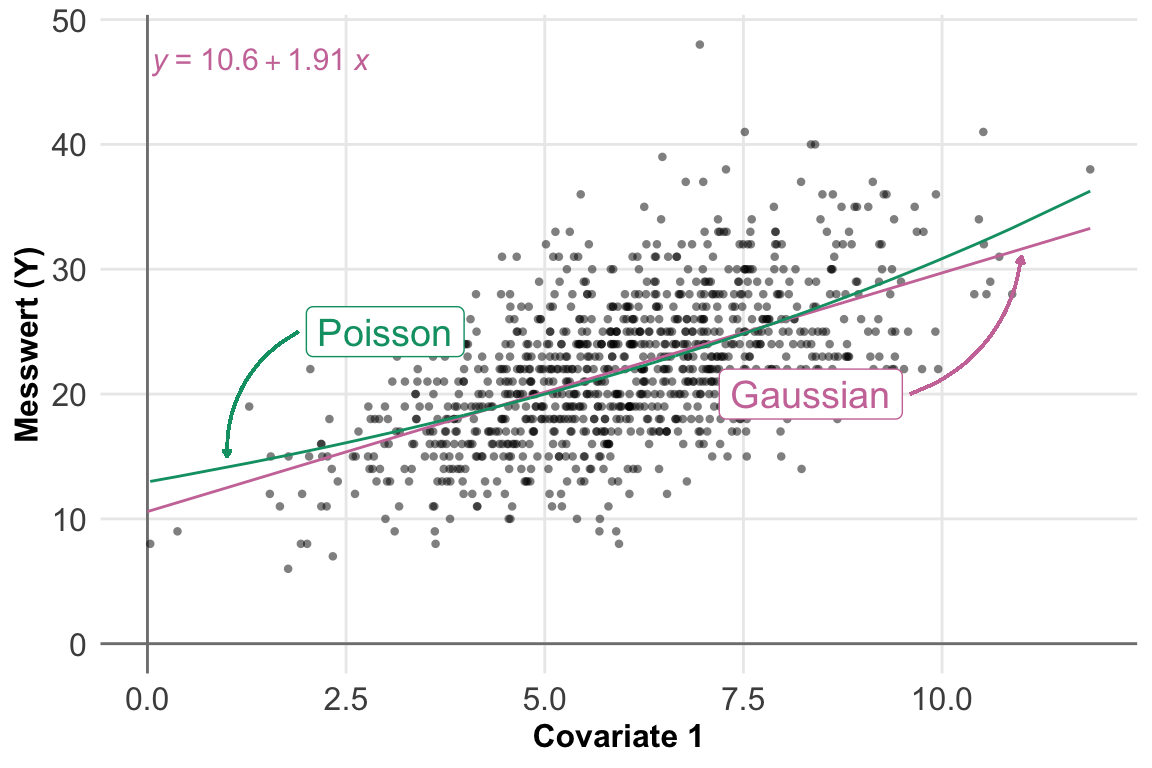
Etwas seltener kommt der Fall vor, dass wir uns nur eine Kovariate anschauen wollen. In diesem Fall haben wir ja eine numerische, kontinuierliche Einflussvariable sowie einen normalverteilten Messwert vorliegen. Wir sind jetzt an der Steigung der Graden durch die Punktewolke interessiert. Daher stellen wir uns hier als Effekt eine Steigung ein und schauen, ob wir diese Steigung wiederfinden. Da wir natürlich auch einen Intercept für die Gradengleichung brauchen, müssen wir diesen auch definieren. Der Intercept ist aber meistens nicht von so großer praktischer Bedeutung.
Einkovariat \(c_1\)
In der folgenden schematischen Darstellung ahbe ich dir einmal einkovariates Modell mitgebracht. Wir haben auf der linken Seite einen kontinuierlichen, normalverteilten Messwert und auf der rechten Seite der Tilde eine kontinuierliche Einflussvariable als Kovariate vorliegen. Wir wollen jetzt die Daten in diesem Kontext generieren.
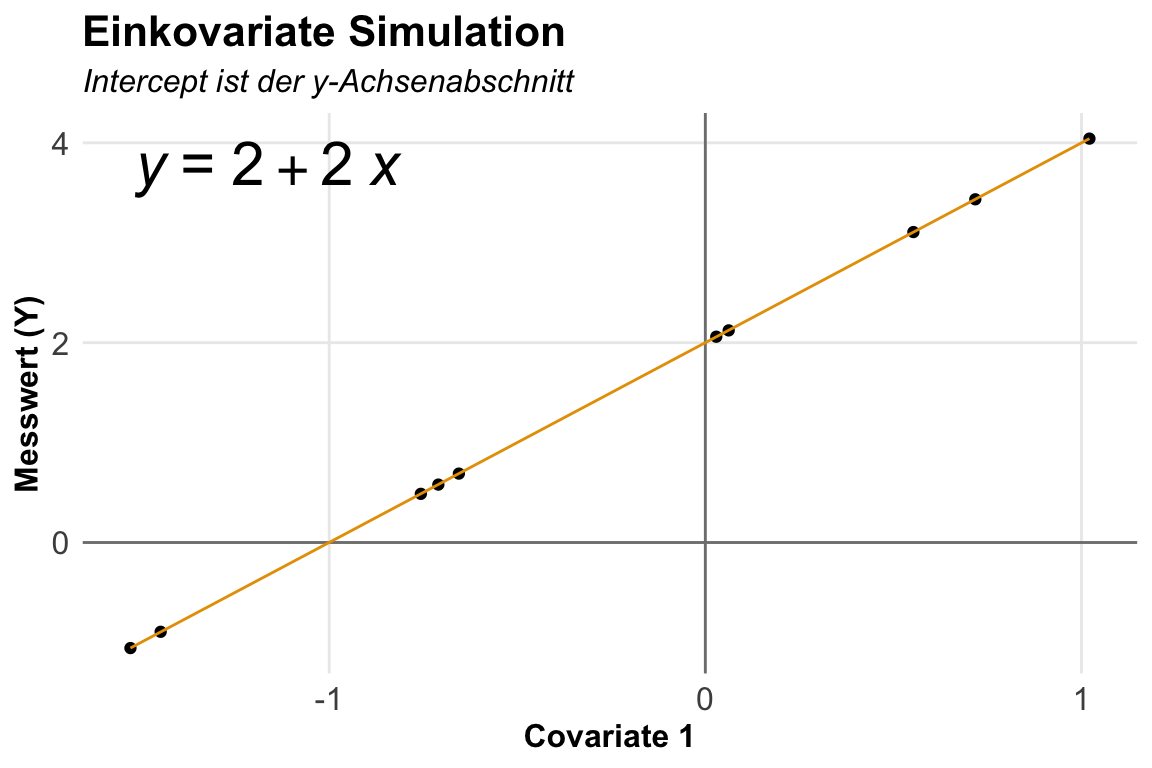
Wenn wir eine normalverteilte Kovariate haben, dann können wir erstmal die Kovariate erschaffen. Dafür nutzen wir dann die Funktion rnorm(). Dann müssen wir noch unseren Messwert mit der Gradengleichung festlegen. Ich zeige hier einmal die Variante mit dem linearen Zusammenhang und habe dann auch noch einen quadratischen mit der Helferfunktion I() ergänzt. Wie du dann möchtest, kannst du auch noch andere nicht lineare Zusammenhänge ergänzen.
R Code [zeigen / verbergen]
cov1_tbl <- tibble(c_1 = rnorm(10, 0, 1),
y_lin = 2 + 2 * c_1 + rnorm(10, 0, 0.001),
y_square = 2 + 1.1 * c_1 + I(1.5*c_1^2) + rnorm(10, 0, 0.001)) |>
gather(key = "power", value = "y", y_lin:y_square)In der folgenden Abbildung zeige ich dir einmal die Ergebnisse der Simulation. Da ich auch hier die Streuung faktisch ausgestellt habe, liegen die Punkte perfekt auf der Gradengleichung wie ich das will. Die lineare Gleichung hat noch einen Keoffizienten für den quadratischen Anteil, der ist hier ab faktisch Null. Wir können hier also sehr gut Zusammenhänge darstellen und dann schauen, ob wir die Koeffizienten dann in der linearen oder nicht-linearen Regression wiederfinden.
Zweikovariat unabhängig \(c_1 + c_2\)
Wenn wir mehrere Kovariaten simulieren wollen, dann erweitern wir das Modell einfach um eine weitere Kovariate. Im simpelsten Fall sind die beiden Kovariaten voneinander unabhängig, daher kommen aus zwei verschiedenen Normalverteilungen und haben damit nichts miteinander zu tun. Daher sind die Werte der Kovariaten untereinander nicht korreliert oder sonst wie voneinander abhängig.
Hier bauen wir uns zwei voneinander unabhängige Kovariaten. Die beiden Kovariaten entstammen aus unterschiedlichen Normalverteilungen und sind somit untereinander nicht korreliert. Dann legen wir einmal den Intercept fest und ergänzen noch die Steigungen als Effekte für die Kovariaten. Am Ende ergänze ich dann noch einen winzigen Fehler damit das Modell auch gerechnet werden kann.
R Code [zeigen / verbergen]
cov2_tbl <- tibble(c_1 = rnorm(10, 0, 1),
c_2 = rnorm(10, 2, 1),
y = 2 +
1 * c_1 +
2 * c_2 +
rnorm(10, 0, 0.001))Dann nutze ich die Funktion correlation() aus dem R Paket {correlation} um mir einmal die Korrelation zwischend den beiden Kovariaten wiedergeben zu lassen. Wir sehen hier, die beiden Kovariaten sind nicht miteinander korreliert. Der Korrelationskoeffizient liegt eher bei Null und ist nicht signifikant.
R Code [zeigen / verbergen]
cov2_tbl |>
correlation(select = c("c_1", "c_2"))# Correlation Matrix (pearson-method)
Parameter1 | Parameter2 | r | 95% CI | t(8) | p
---------------------------------------------------------------
c_1 | c_2 | -0.34 | [-0.80, 0.37] | -1.01 | 0.340
p-value adjustment method: Holm (1979)
Observations: 10Dann schauen wir nochmal auf die Koeffizientend es Modells, ob wir auch unsere Effekte wiederfinden. Das sieht soweit gut aus. Wir haben die eingestellten Steigungen in den Koeffizienten wiedergefunden.
R Code [zeigen / verbergen]
lm(y ~ c_1 + c_2, cov2_tbl) |>
coef() |> round(2)(Intercept) c_1 c_2
2 1 2 Ich erstelle hier keine Abbildung in einem 3D Scatterplot, da mir das zu viel Aufwand ist und wir sehr wenig auf den 3D Scatterplots sinnvoll interpretieren können. Wir schauen uns auch selten zweikovariate Modelle in einer Abbildung an, sondern bewerten eben die Koeffizienten und andere statistsiche Maßzahlen für die Güte der Modellierung. Hier soll es dann reichen. Mehr dazu dann in den Kapiteln zu den Grundlagen des statistsichen Modellierens.
Multikovariat abhängig \(c_1 + c_2 + c_3\)
Jetzt kommen wir zu einem etwas nischigen Thema, nämlich der Tatsache, dass wir korrelierte Kovariaten vorliegen haben. Deshalb nehmen wir auch gleich drei Korvariaten. Das ist natürlich in der Statistik ein Problem, wie wir es auch im Kapitel zu der multiplen Regression nachlesen können. Mit Statistik meine ich, dass wir dann eben ein paar Probleme bei der Modellierung haben. Da gehe ich hier aber nicht tiefer drauf ein. Solche Daten zu generieren ist dann daher eher selten von Interesse in einem praktischen Kontext. Entweder liegen die Kovariaten korre,iert vor oder eben nicht. Wenn wir natürlich Algorithmen entwicklen wollen, dann ist das schon ein wichtiges Theme, wenn die Kovariaten nicht unabhängig voneinander sind.
Wir nutzen jetzt im Folgenden einmal das R Paket {simstudy} da die Genierung von korrelierten Daten mit einem Paket einfacher ist als alles per Hand zu machen. Es geht auch alles per Hand in einem tibble mit {mvtnorm} aber das machen wir hier jetzt nicht, da es viel zu kompliziert ist. Ich kann es nur empfehlen, wenn du dich mit dem Simulieren von Daten beschäftigen willst. Dann müsstest du dich auch nochmal mit dem Tutorium How would you explain the difference between correlation and covariance? beschäftigen, da wir in {mvtnorm} mit Kovarianzen beschäftigen.
Damit wären wir auch gleich beim großen Vorteil von {simstudy}. Wir können nämlich direkt eine Korrelationsmatrix einpflegen und müssen nicht über die Kovarianz als schwierigeres Konzept gehen. Die Kovarianz festlegen ist nämlich nicht ganz so einfach. Die Korrelation vorher festzulegen aber schon. Was wir dann auch in der Vingette Correlated Data nachlesen können. Im folgenden habe ich dir einmal eine Korrlationsmatrix für die drei Kovariaten \(c_1\) bis \(c_3\) mitgebracht. Die Korrelationsmatrix muss symmetrisch sein. Je höher die absolute Korrelation desto stärker ist der Zusammenhang.
R Code [zeigen / verbergen]
cor_mat <- matrix(c(1, 0.7, 0.2,
0.7, 1, 0.8,
0.2, 0.8, 1), nrow = 3,
dimnames = list(c("c_1", "c_2", "c_3"), c("c_1", "c_2", "c_3")))
cor_mat c_1 c_2 c_3
c_1 1.0 0.7 0.2
c_2 0.7 1.0 0.8
c_3 0.2 0.8 1.0Dann können wir die Funktion genCorData() nutzen um uns korreltierte Kovariaten zu erstellen. Ich habe hier einmal eintausend Beobachtungen gewählt. Dann müssen wir noch den Mittelwert in allen Kovariaten festlegen sowie die Streuung. Wir nehmen dann die Korrelationsmatrix mit in die Funktion und benennen noch die Kovariaten entsprechend. Wir erstellen uns dann den Messwert separat mit den gewollten Effekten.
R Code [zeigen / verbergen]
cov2_dependent_dt <- genCorData(1000, mu = c(4, 12, 3), sigma = c(1, 2, 3),
corMatrix = cor_mat,
cnames = c("c_1", "c_2", "c_3")) |>
mutate(y = 2 + 1.5 * c_1 + 2 * c_2 + 2.5 * c_3 + rnorm(1000, 0, 0.001))Im Folgenden siehst du dann einmal die Datentabelle der eintausend Beobachtungen. Wir haben die ID Spalte und dann die drei Kovariaten gefolgt von dem Messwert.
R Code [zeigen / verbergen]
cov2_dependent_dtKey: <id>
id c_1 c_2 c_3 y
<int> <num> <num> <num> <num>
1: 1 6.396581 14.050618 3.067163 47.36484
2: 2 4.139633 13.254738 6.046905 49.83687
3: 3 3.428215 8.120047 -3.039935 15.78313
4: 4 3.062534 11.051392 5.231867 41.77626
5: 5 1.878001 10.890102 5.654227 40.73364
---
996: 996 4.459314 12.990747 4.349280 45.54327
997: 997 4.217033 12.806682 3.063211 41.59494
998: 998 3.619132 11.552059 3.209882 38.55683
999: 999 3.964356 12.338354 2.840538 39.72552
1000: 1000 3.955933 12.639828 2.007950 38.23297In den rohen Daten ist die Korrelation faktisch nicht zu sehen. Dafür müssen wir uns dann einmal die Korrelation berechnen lassen. Wie wir dann im Folgenden einmal sehen, passt es sehr gut. Wir erhalten die Korrelationen wieder, die wir auch eingestellt haben.
R Code [zeigen / verbergen]
correlation(cov2_dependent_dt, select = c("c_1", "c_2", "c_3")) |>
summary(redundant = TRUE)# Correlation Matrix (pearson-method)
Parameter | c_1 | c_2 | c_3
---------------------------------------
c_1 | | 0.72*** | 0.21***
c_2 | 0.72*** | | 0.79***
c_3 | 0.21*** | 0.79*** |
p-value adjustment method: Holm (1979)Am Ende nochmal schauen, ob wir auch die Effekte wiederbekommen, die wir eingestellt haben. Auch hier sieht es sehr gut aus. Hier muss ich natürlich erwähnen, dass die Effekte groß sind die Fallzahl riesig und auch die Streuung in den Daten gering. Daher klappt hier auch alles so schön.
R Code [zeigen / verbergen]
lm(y ~ c_1 + c_2 + c_3, data = cov2_dependent_dt)
Call:
lm(formula = y ~ c_1 + c_2 + c_3, data = cov2_dependent_dt)
Coefficients:
(Intercept) c_1 c_2 c_3
1.999 1.500 2.000 2.500 Dann haben wir auch unsere Effekte in dem Modell wiedergefunden. Es gibt verschiedene Korrelationsstrukturen, die wir auch in {simstudy} berücksichtigen können. Eine weitere Möglichkeit sind natürlich auch Zeitreihen in denen wir eine Abhängigkeit vorliegen haben. Aber auch hier reicht es meist, wenn der Trend einigermaßen stimmt. Dazu dann aber mehr in dem Abschnitt zu Zeitreihen.
“Brauchen wir eine Generierung von korrelierten Kovariaten? Ich würde mal sagen in der allgmeinen Anwendung und Auswertung reicht es, wenn wir die Daten einfach unkorreliert generieren und dann darauf die Algorithmen laufen lassen. Aber ich selber brauche es manchmal für Beispieldaten. Daher gibt es diesen Abschnitt wirklich.” — Jochen Kruppa-Scheetz, meiner bescheidener Meinung nach.
Mischung von Normalverteilungen
Wenn wir eine etwas komplexere Verteilung des Messwerts vorliegen haben wollen, dann lohnt sich auch einmal eine gemischte Normalverteilung zu bauen. Wir haben dann nicht eine Normalverteilung vorliegen sondern eine sehr bergige. Die bergige Normalverteilung besteht dann aber aus verschiedenen anderen Normalverteilungen, die wir dann übereinanderlegen. So können wir dann auch zweigipfelige oder wie in diesem Fall dreigipfelige Verteilungen bauen. Wir nutzen hier das R Paket {simstudy} und generieren uns erstmal drei Normalverteilungen.
R Code [zeigen / verbergen]
mix_def <- defData(varname = "y_1", formula = 1, variance = 1)
mix_def <- defData(mix_def, varname = "y_2", formula = 5, variance = 4)
mix_def <- defData(mix_def, varname = "y_3", formula = 9, variance = 1)Die drei Normalverteilungen, die wir gerade generiert haben \(y_1\) bis \(y_3\), mischen wir jetzt einmal nach festen Anteilen miteinander. Dafür nutzen wir die folgende Schreibweise in {simstudy}. Die Schreibweise ist erstmal etwas ungewohnt. Wir wollen 30% der Werte von \(y_1\) also schreiben wir y_1 | 0.3. Dann noch 40% der Werte von \(y_2\) mit y_2 | 0.4 sowie 30% der Werte von \(y_3\) mit y_3 | 0.3. Wir nennen dann die Spalte y_null. Wir haben hier dann die Nullverteilung erstellt.
R Code [zeigen / verbergen]
mix_def <- defData(mix_def, varname = "y_null",
formula = "y_1 | 0.3 + y_2 | 0.4 + y_3 | 0.3",
dist = "mixture")Ich generiere mir dann eintausend Beobachtungen, damit wir am Ende auch was sehen.
R Code [zeigen / verbergen]
mix_null_dt <- genData(1000, mix_def)Dann kannst du dir in der folgenden Abbildung einmal anschauen, wie die drei einzelnen Normalverteilungen aussehen und wie sich dann die Kombination durch die Addierung ergibt. Wir nennen diese Verteilung jetzt unsere Nullverteilung, da wir ja noch eine andere Verteilung benötigen um einen Effekt darzustellen.
Jetzt möchte ich noch eine Alternative darstellen. Wir haben ja noch eine Gruppe mit Effekt. Daher adddiere ich einfach \(+2\) auf die Nullverteilung auf und generiere mir neue Werte. Achtung, ich verschiebe hier nicht einfach die Nullverteilung sondern generiere mir neue Zahlen mit einem um \(+2\) erhöhten kombinierten Mittelwert.
R Code [zeigen / verbergen]
mixed_alt_def <- defDataAdd(varname = "y_alt", formula = "y_null + 2", variance = 1,
dist = "normal")Dann müssen wir noch die beiden Verteilungen miteinander kombinieren.
R Code [zeigen / verbergen]
mix_null_alt_dt <- addColumns(mixed_alt_def, mix_null_dt)In der folgenden Abbildung siehst du nochmal die beiden Vereilungen der beiden Gruppen. Wir haben eine Gruppe, die der Nullverteilung folgt und einmal eine Verteilung für die Alternative. Wenn du noch mehr Gruppen haben wollen würdest, dann musst du entsprechend mehr Verteilungen verschieben.
Dann wollen wir natürlich auch noch zufällige Zahlen genieren. Wir ziehen jetzt jeweils drei Beobachtungen aus den beiden Verteilungen. Wenn wir natürlich eine sehr komplexe Verteilung in der Grundgesamtheit haben, dann wird diese natürlich durch sehr kleine Stichproben kaum repräsentiert.
R Code [zeigen / verbergen]
mix_null_alt_dt |>
select(y_null:y_alt) |>
pivot_longer(y_null:y_alt) |>
group_by(name) |>
slice_sample(n = 3)# A tibble: 6 × 2
# Groups: name [2]
name value
<chr> <dbl>
1 y_alt 4.23
2 y_alt 9.89
3 y_alt 9.20
4 y_null 8.51
5 y_null 9.30
6 y_null 6.51Auch hier ist die Frage, wozu brauchen wir das? Vermutlich nicht im praktischen Alltag, aber in der Testung von Algorithmen mag eine mehrgipflige Verteilung schon von Interesse sein. Wir haben ja hier mehrere Normalverteilungen, die sich aufaddieren. Jede dieser Normalverteilungen könnte eine unentdeckte Subpopulation sein, die eben dann eine andere Verteilung des Frischegewichts hat. Ich vermute aber, dass die Anwendungsfälle eher selten sind.
21.5.2 Possionverteilt
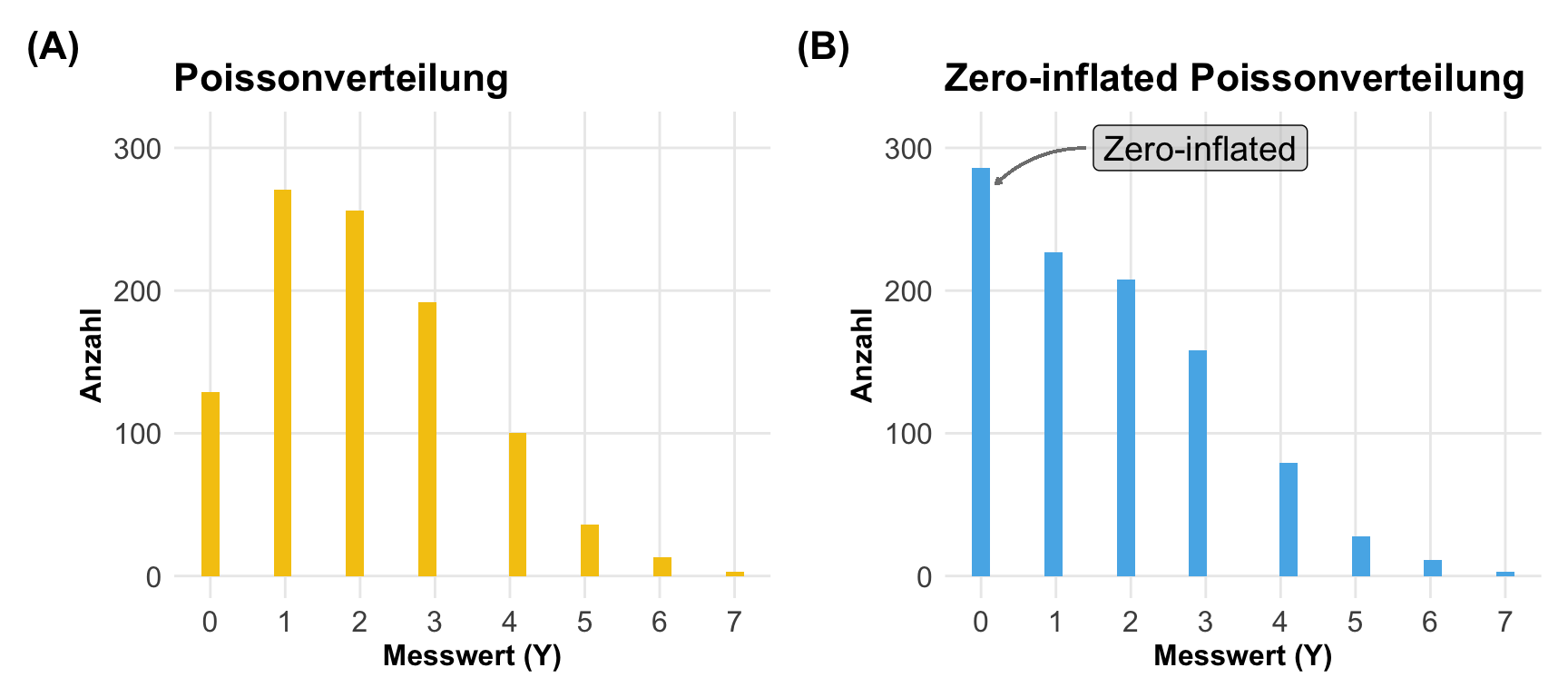
Nachdem wir vieles einmal mit einer Normalverteilung durchprobiert haben, kommen wir jetzt einmal zu einer anderen Verteilung. Wir fangen mit der Poissonverteilung an, da die Poissonverteilung der Normalverteilung gar nicht so unähnlich ist. Wir nutzen die Poissonverteilung wenn wir Zähldaten abbilden wollen. Daher ist die Poissonverteilung immer ganzzahlig oder diskret. Wenn wir was zählen, dann haben wir auch immer positive Werte einschließlich der Nullzählungen vorliegen. Mit dem Wissen können wir dann anfangen unsere Zähldaten in einer Poissonverteilung zu generieren. Wir generieren jetzt unsere Daten mit dem R Paket {simstudy}, da wir hier es dann sehr viel einfacher haben als händisch in R.
Gleich vorweg, es gibt auch eine einfachere Möglichkeit sich die Zähldaten zu genierieren. Du kannst auch eine Normalverteilung nehmen und dann einfach runden. Dann hast du zwar eine leichte Abweichung, aber für das Ausprobieren für Analysen macht es fast keinen Unterschied.
R Code [zeigen / verbergen]
rnorm(n = 5, mean = 10, sd = 1) |> round()[1] 11 11 10 11 11Die Abweichungen sind tolerable wie Kruppa et al. (2016) gezeigt hat. Wenn du dann noch darauf achtest, dass du keine negativen Werte simulierst, dann ist alles soweit in Ordnung. Ich würde alle negativen Werte aus einer Normalverteilung dann auf Null setzen. Das simuliert eine Poissonverteilung am Besten.
Gegenüber der Normalverteilung müssen wir jetzt einige Anpassungen vornehmen. Im Folgenden müssen wir die Fallzahl erhöhen, da die Poissonverteilung keine eigenen Parameter für die Varianz hat. Die Streuung ist nämlich eien Funktion des Mittelwerts. Je größer der Mittelwert, desto größer die Varianz. Daher kann ich keine sehr kleine Streuung einstellen um auch bei kleinen Fallzahlen meine Effekte oder Mittelwerte wiederzuerhalten. Ich nutze daher hier immer um die eintausend Beobachtungen, damit wir hier auch stabile Effekte wiedergegeben bekommen.
Faktorielles Design
Beginnen wir mit einem faktoriellen Design. Wir schauen uns jetzt einmal einen Faktor mit mehr als zwei Leveln an sowie zwei Faktoren mit einmal drei Leveln und einmal zwei Leveln. Dann nehmen wir noch die Interaktion mit hinzu. Wenn wir uns mit dem faktoriellen Design beschäftigen dann generieren wir zuerst das Faktorenraster und ergänzen dann den Messwert aus der Poissonverteilung. Das ist jetzt hier der etwas größere Unterschied zu den normalverteilten Messwerten. Wir brauchen hier immer unser Raster und dann ergänzen wir unseren Messwert mit dem R Paket {simstudy}. Prinzipiell geht es auch für das einfaktorielle Design einfacher über rpois(), aber wir üben ja hier auch für die komplexeren Modelle.
Das R Paket {simstudy} nutzt als Speicherung der Daten nicht ein tibble sondern das R Paket {data.table}. Um unsere tibble dann umzuformen nutze ich die Funktion as.data.table(). Der Rest bleibt aber faktisch gleich.
Wir beginnne gleich mit einem einfaktoriellen Design mit mehr als 2 Leveln. Ich schaue hier mir mal einen Faktor mit drei Gruppen an. Der simple Fall mit nur zwei Gruppen ist eher selten und deshalb überspringe ich auch den Zweigruppenfall. Im Prinzip kannst du dir dann den Zweigruppenfall in rpois() schnell selber bauen.
Einfaktoriell \(f_A\) mit >2 Leveln
Wenn wir ein einfaktorielles Design vorliegen haben, dann brauchen wir einen Faktor mit drei Leveln sowie eine Anzahl an Beobachtungen pro Level. In den folgenden Tabs zeige ich dir dann einmal wie wir die Daten in {simstudy} generieren können. Das ist hier dann eher eine Fingerübung und etwas umständlich. Das geht natürlich für so einen einfachen Fall mit einem Faktor schneller mit der Funktion rpois() wo wir dann schneller die Daten erstellen können. Am Ende gehe ich nochmal auf Excel ein.
Wir bauen uns hier erstmal das Datenraster für den Faktor auf, da wir dann das Raster mit dem R Paket {simstdy} erweitern und mit einem poissonverteilten Messwert ergänzen. Das heißt, wir nutzen die Funktion expand_grid() um uns für die drei Level jeweils eintausend Beobachtungen oder Wiederholungen zu erstellen. Da wir gleich mit {simstudy} weiterarbeiten brauche ich das Datenraster als ein data.table Objekt.
R Code [zeigen / verbergen]
fac1_dt <- expand_grid(f_a = 1:3,
rep = 1:1000) |>
as.data.table()Dann können wir uns auch schon einmal den Datensatz anschauen, wo wir eben nur das Datenraster für die Faktoren enthalten haben. Wir haben dreitausend Beobachtungen jeweils eintausend Beobachtungen pro Level des Faktors.
R Code [zeigen / verbergen]
fac1_dt f_a rep
<int> <int>
1: 1 1
2: 1 2
3: 1 3
4: 1 4
5: 1 5
---
2996: 3 996
2997: 3 997
2998: 3 998
2999: 3 999
3000: 3 1000Jetzt brauchen wir noch die Definition für unseren poissonverteilten Messwert in {simstudy}. Dafür benennen wir dann den Messwert mit y und geben noch die Formel an. Jedes Level hat einen Effekt von \(+4\). Dann noch die Verteilung angeben, damit wir hier auch aus der Possionverteilung ziehen.
R Code [zeigen / verbergen]
y_pois_def <- defDataAdd(varname = "y", formula = "0 + 4*f_a",
dist = "poisson")Jetzt bringen wir unser Datenraster mit unserer Datendefinition zusammen. Wir erhalten dann einen neuen Datensatz mit unserem poissonverteilten Messwert. Am Ende möchte ich dann noch die Spalte f_a als Faktor mutieren und die Level umbenennen. Dann sind wir auch schon fertig.
R Code [zeigen / verbergen]
fac1_pois_dt <- addColumns(y_pois_def, fac1_dt) |>
mutate(f_a = factor(f_a, labels = str_c("A.", 1:3))) In der folgenden Abbildung siehst du einmal die dreitausend generierten Beobachtungen pro Gruppe. Wir sehen hier einmal den Intercept bei Null sowie den Effekt von \(+4\) in jedem Level des Faktors. Die Varianz steigt mit dem Mittelwert an. Die Poissonverteilung ist eine schiefe Verteilung bei einem geringen Mittelwert. Je größer der Mittelwert wird, desto ähnlicher wird die Poissonverteilung einer Normalverteilung.
In dem einfaktoriellen Fall können wir uns auch die Daten einmal mit der Funktion rpois() erstellen. Wir brauchen hier einmal die Fallzahl sowie den Mittelwert als lambda. Wir können in einer Poissonverteilung keine Streuung angeben, da die Streuung eine Funktion des Mittelwerts ist. Je größer der Mittelwert desto größer die Streuung.
R Code [zeigen / verbergen]
tibble("1" = rpois(n = 3, lambda = 2),
"2" = rpois(n = 3, lambda = 8)) |>
gather(key = "f_a", value = "y")# A tibble: 6 × 2
f_a y
<chr> <int>
1 1 1
2 1 2
3 1 2
4 2 7
5 2 9
6 2 12In diesem Fall ist eben sehr einfach sich die Daten zu bauen. Meiner Meinung nach reicht so eine simple Art auch vollkommen aus, wenn wir die Daten nur zum Ausprobieren brauchen und nicht unbedingt gerundete Normalverteilungsdaten wollen.
In Excel können wir uns keine Zuafallszahlen aus einer Poissonverteilung generieren lassen. Daher nutzen wir hier auch NORMINV() und runden dann die Ergebnisse ganzahlig. Wenn wir negative Werte haben sollten, dann müssen wir diese Werte dann auf Null setzen.
Zweifaktoriell ohne Interaktion \(f_A + f_B\)
Jetzt kommen wir zu den etwas interessanteren Design mit dem zweifakotriellen Experiment ohne Interaktion. Wir haben jetzt hier zwei Faktoren \(f_A\) und \(f_B\) mit jeweils drei oder zwei Leveln vorliegen. Die beiden Faktoren sind untereinander unabhängig, daher haben wir hier keine Interaktion vorliegen. Wir wollen jetzt einmal eintausend poissonverteilte Messwerte generieren. Dafür nutze ich wie immer einmal das R Paket {simstudy} sowie den Weg über die Funktion rpois(). In Excel können wir keine Poissondaten erschaffen. Hier müssen wir dann die Daten aus einer Normalverteilung entsprechend runden.
Wir bauen uns hier erstmal das Datenraster für die beiden Faktoren auf, da wir dann das Raster mit dem R Paket {simstdy} erweitern und mit einem poissonverteilten Messwert ergänzen. Das heißt, wir nutzen die Funktion expand_grid() um uns für die drei Level in der Kombination der anderen zwei Level jeweils eintausend Beobachtungen oder Wiederholungen zu erstellen. Da wir gleich mit {simstudy} weiterarbeiten brauche ich das Datenraster als ein data.table Objekt.
R Code [zeigen / verbergen]
fac2_dt <- expand_grid(f_a = 1:3,
f_b = 1:2,
rep = 1:1000) |>
as.data.table()Dann können wir uns auch schon einmal den Datensatz anschauen, wo wir eben nur das Datenraster für die Faktoren enthalten haben. Wir haben sechstausend Beobachtungen jeweils eintausend Beobachtungen pro Faktorkombination der beiden Faktoren.
R Code [zeigen / verbergen]
fac2_dt f_a f_b rep
<int> <int> <int>
1: 1 1 1
2: 1 1 2
3: 1 1 3
4: 1 1 4
5: 1 1 5
---
5996: 3 2 996
5997: 3 2 997
5998: 3 2 998
5999: 3 2 999
6000: 3 2 1000Jetzt brauchen wir noch die Definition für unseren poissonverteilten Messwert in {simstudy}. Dafür benennen wir dann den Messwert mit y und geben noch die Formel an. Jedes Level des Faktors \(f_A\) hat einen Effekt von \(+2\) und jedes Level des Faktors \(f_B\) hat einen Effekt von \(+6\). Da wir hier nur zwei Level vorliegen haben, unterschieden sich die beiden Level \(B.1\) und \(B.2\) einfach um sechs Einheiten. Dann noch die Verteilung angeben, damit wir hier auch aus der Possionverteilung ziehen.
R Code [zeigen / verbergen]
y_pois_def <- defDataAdd(varname = "y", formula = "0 + 2*f_a + 6*f_b",
dist = "poisson")Jetzt bringen wir unser Datenraster mit unserer Datendefinition zusammen. Wir erhalten dann einen neuen Datensatz mit unserem poissonverteilten Messwert. Am Ende möchte ich dann noch die Spalte f_a und f_b als Faktoren mutieren und die Level umbenennen. Dann sind wir auch schon fertig.
R Code [zeigen / verbergen]
fac2_pois_dt <- addColumns(y_pois_def, fac2_dt) |>
mutate(f_a = factor(f_a, labels = str_c("A.", 1:3)),
f_b = factor(f_b, labels = str_c("B.", 1:2)))In der folgenden Abbildung siehst du einmal die sechstausend generierten Beobachtungen pro Gruppe. Wir sehen hier einmal den Intercept bei Null sowie den Effekt von \(+4\) in jedem Level des Faktors \(f_A\) sowie die Verschiebung in dem Faktor \(f_B\). Die Varianz steigt mit dem Mittelwert an. Die Poissonverteilung ist eine schiefe Verteilung bei einem geringen Mittelwert. Je größer der Mittelwert wird, desto ähnlicher wird die Poissonverteilung einer Normalverteilung.
In dem zweifaktoriellen Fall können wir uns auch die Daten auch einmal mit der Funktion rpois() erstellen. Wir brauchen hier einmal die Fallzahl sowie den Mittelwert als lambda. Wir können in einer Poissonverteilung keine Streuung angeben, da die Streuung eine Funktion des Mittelwerts ist. Je größer der Mittelwert desto größer die Streuung. Hier bauen wir uns aber erst den Vektor der Effekte und stecken dann diesen in die Funktion rpois(). Das ist etwas anders als bei der Normalverteilung. Wir haben ja hier keinen symmetrischen Fehlerterm, den wir aufaddieren.
R Code [zeigen / verbergen]
fac2_nointer_tbl <- expand_grid(f_a = 1:3,
f_b = 1:2,
rep = 1:1000) |>
mutate(y_eff = 0 +
2 * f_a +
6 * f_b,
y = rpois(length(y_eff), lambda = y_eff)) |>
mutate(f_a = as_factor(f_a),
f_b = as_factor(f_b))Dann schauen wir uns nochmal an, ob wir auch die Mittelwerte und Mediane wiedererhalten.
R Code [zeigen / verbergen]
fac2_nointer_tbl |>
group_by(f_a, f_b) |>
summarise(round(mean(y)), median(y))`summarise()` has grouped output by 'f_a'. You can override using the `.groups`
argument.# A tibble: 6 × 4
# Groups: f_a [3]
f_a f_b `round(mean(y))` `median(y)`
<fct> <fct> <dbl> <dbl>
1 1 1 8 8
2 1 2 14 14
3 2 1 10 10
4 2 2 16 16
5 3 1 12 12
6 3 2 18 18In diesem Fall ist schon nicht mehr so einfach sich die Daten zu bauen. Meiner Meinung nach solltest du dann spätenstens hier einmal zu {simstudy} überwechseln. Die Funktionalität von dem R Paket ist da wirklich besser, als sich das selber in einem tibble zusammenzubauen.
In Excel können wir uns keine Zuafallszahlen aus einer Poissonverteilung generieren lassen. Daher nutzen wir hier auch NORMINV() und runden dann die Ergebnisse ganzahlig. Wenn wir negative Werte haben sollten, dann müssen wir diese Werte dann auf Null setzen. Bei zwei Faktoren hört hier schon etwas der Spaß auf, da wir ja alle Fakotrkombinationen dann einzeln anlegen müssen.
Zweifaktoriell mit Interaktion \(f_A + f_B + f_A \times f_B\)
Für den zweifaktoriellen Fall mit Interaktion haben wir jetzt hier zwei Faktoren \(f_A\) und \(f_B\) mit jeweils drei oder zwei Leveln vorliegen. Ich gehe diesmal nur nochauf auf das R Paket {simstdy} ein, da es händisch und in Excel jhier nicht mehr funktioniert. Wenn du es händisch machen möchtest, dann kannst du dir natürlich für alle Faktorkombinationen die Effekte zusammenbauen und dann rpois() für die Zufallszahlen nutzen. Ich empfinde daran aber keinen Gewinn mehr.
Wir bauen uns hier erstmal das Datenraster wie schon vorab für die beiden Faktoren auf, da wir dann das Raster mit dem R Paket {simstdy} erweitern und mit einem poissonverteilten Messwert ergänzen. Das heißt, wir nutzen die Funktion expand_grid() um uns für die drei Level in der Kombination der anderen zwei Level jeweils eintausend Beobachtungen oder Wiederholungen zu erstellen. Da wir gleich mit {simstudy} weiterarbeiten brauche ich das Datenraster als ein data.table Objekt. Soweit ist alles gleich. Wir bauen uns die Interalktion jetzt anders.
R Code [zeigen / verbergen]
fac2_tbl <- expand_grid(f_a = 1:3, f_b = 1:2, rep = 1:1000) |>
mutate(f_a = as_factor(f_a),
f_b = as_factor(f_b))Jetzt gehen wir aber noch einen Schritt weiter und bauen das Datenraster als Dummykodierung. Somit steht jetzt jede Spalte dafür, ob ein Effekt dann entsprechend für die Beobachtung berücksichtigt wird. Hier können wir dann auch die Interaktion in den jeweiligen Faktorkombinationen \(A.2.B.2\) und \(A.3.B.2\) richtig modellieren. Es ist aber immer noch nicht einfach, das kann ich unterschreiben.
R Code [zeigen / verbergen]
fac2_model_dt <- fac2_tbl |>
model_matrix(~ f_a * f_b) |>
clean_names() |>
as.data.table()Dann können wir uns auch schon einmal die Modellmatrix für den Datensatz anschauen, wo wir eben die Faktoren und die Interaktion enthalten haben. Wir haben sechstausend Beobachtungen jeweils eintausend Beobachtungen pro Faktorkombination der beiden Faktoren. In den Faktorkombinationen sind dann auch die Interaktionen enthalten.
R Code [zeigen / verbergen]
fac2_model_dt intercept f_a2 f_a3 f_b2 f_a2_f_b2 f_a3_f_b2
<num> <num> <num> <num> <num> <num>
1: 1 0 0 0 0 0
2: 1 0 0 0 0 0
3: 1 0 0 0 0 0
4: 1 0 0 0 0 0
5: 1 0 0 0 0 0
---
5996: 1 0 1 1 0 1
5997: 1 0 1 1 0 1
5998: 1 0 1 1 0 1
5999: 1 0 1 1 0 1
6000: 1 0 1 1 0 1Jetzt müssen wir für jede Spalte den Effekt definieren. Daher haben wir dann sechs Effekte. Einmal einen für den Intercept und dann für die jeweiligen Faktorlevel. Hier müssen wir dann aber die Multiplikation mit selber einrechnen. Daher ist der Effekt des \(f_A\) zwar \(+2\), aber müssen wir dann schon bei \(A.3\) eben \(+2+2 = +4\) rechnen. Es ist wirklich etwas anstrengend. Dann kommen noch die beiden Terme für die Interaktion.
R Code [zeigen / verbergen]
y_pois_def <- defDataAdd(varname = "y",
formula = "5 * intercept + 2 * f_a2 + 4 * f_a3 +
3 * f_b2 + 0 * f_a2_f_b2 + -6 * f_a3_f_b2",
dist = "poisson")Jetzt bringen wir unser Datenraster mit unserer Datendefinition zusammen. Wir erhalten dann einen neuen Datensatz mit unserem poissonverteilten Messwert. Am Ende möchte ich dann noch die Spalte f_a und f_b auswählen sowie als Faktoren mutieren und die Level umbenennen. Dann sind wir auch schon fertig.
R Code [zeigen / verbergen]
fac2_pois_dt <- addColumns(y_pois_def, fac2_model_dt) |>
add_column(fac2_tbl) |>
select(f_a, f_b, y) |>
mutate(f_a = factor(f_a, labels = str_c("A.", 1:3)),
f_b = factor(f_b, labels = str_c("B.", 1:2)))Am Ende musst du dir das alles einmal in der Abbildung anschauen. Hier siehst du dann auch gut die Interaktion im letzten Level vom Faktor \(f_A\). Wir haben dort in der Faktorkombination \(A.3.A.2\) nicht einen Mittelwert von 12 sondern von 6 vorliegen. Hier dreht sich der immer positive Effekt des Faktors \(f_B\) einmal ins Negative um. Die Erstellung ist ohne einen Visualisierung wirklich schwer nachzuvollziehen und zu überprüfen. Am Ende musst du dann immer schauen, ob du dann die Effekte bei einer großen Fallzahl wiederfindest und dur dann einen kleineren Datensatz für dich bauen. Die Implementierung einer Interaktion ist eben nicht ganz so einfach, wenn es um die Generierung von faktoriellen Experimenten geht.
“Brauchen wir eine Generierung von zweifaktoriellen Experimenten aus einer Poissonverteilung mit Interaktion wirklich? Diese Frage kann man sich auch hier stellen. Ich selber baue mir gerne die Interaktion dann händisch in Excel oder aber Runde mir eine Normalverteilung. Das reicht im Kontext der Lehre und zum Testen vollkommen aus. Ich hatte aber Spaß zu verstehen, wie die Interaktion entsteht und wie ich die Interaktion nachbauen kann. Dafür hat es sich gelohnt.” — Jochen Kruppa-Scheetz, meiner bescheidener Meinung nach.
{simstudy} für Faktoren
Nachdem ich hier schon sehr weit war mit der Funktion expand_grid() habe ich Abends dann noch per Zufall den Blogeintrag Testing multiple interventions in a single experiment gelesen. Wir können also auch ein faktorielles Design auch in {simstudy} bauen. Hätte mich dann auch etwas gewundert wenn es dann doch nicht ginge. Es ist immer eine Frage des Nestings oder der Schachtelung, also wie wollen wir die beiden Faktoren jeweils ineinander verschränkt wissen. Wir können die Funktion genMultiFac() nutzen um uns mehrere Faktoren zu erschaffen. Wir können dabei auch gleich die Spaltennamen setzen und sagen, wie oft eine Faktorkombination wiederholt werden soll.
R Code [zeigen / verbergen]
fac2_dt <- genMultiFac(each = 5, nFactors = 2, levels = c(3, 2),
colNames = c("f_a", "f_b")) Dann einmal die Daten anschauen. Ich zeige die hier als tibble damit nicht so viel ausgegeben wird. In der Standardimplementierung des data.table kriegen wir immer einen sehr langen Ausdruck.
R Code [zeigen / verbergen]
fac2_dt |> as_tibble()# A tibble: 30 × 3
id f_a f_b
<int> <int> <int>
1 1 1 1
2 2 1 1
3 3 1 1
4 4 1 1
5 5 1 1
6 6 2 1
7 7 2 1
8 8 2 1
9 9 2 1
10 10 2 1
# ℹ 20 more rowsJetzt brauchen wir noch die Definition für unseren poissonverteilten Messwert in {simstudy}. Das ist das Gleiche wie schon bevor. Hier muss ich definieren, wie die Effekte der beiden Faktoren sind. Auch hier gilt, das Ganze ist dann ohne Interaktion gebaut. Theoretisch könnest du noch einen globalen Interaktionseffekt mit 3 * f_a * _f_b hinzufügen, aber dann wäre der Effekt der Interaktion multiplikativ eben \(+3\) über die Level der Faktoren.
R Code [zeigen / verbergen]
y_pois_def <- defDataAdd(varname = "y",
formula = "-2 + 2*f_a + 6*f_b",
dist = "poisson")Jetzt bringen wir unser Datenraster mit unserer Datendefinition zusammen. Wir erhalten dann einen neuen Datensatz mit unserem poissonverteilten Messwert. Am Ende möchte ich dann noch die Spalte f_a und f_b als Faktoren mutieren und die Level umbenennen. Dann sind wir auch schon fertig.
R Code [zeigen / verbergen]
addColumns(y_pois_def, fac2_dt) |>
mutate(f_a = factor(f_a, labels = str_c("A.", 1:3)),
f_b = factor(f_b, labels = str_c("B.", 1:2))) |>
as_tibble()# A tibble: 30 × 4
id f_a f_b y
<int> <fct> <fct> <int>
1 1 A.1 B.1 6
2 2 A.1 B.1 2
3 3 A.1 B.1 7
4 4 A.1 B.1 5
5 5 A.1 B.1 8
6 6 A.2 B.1 8
7 7 A.2 B.1 7
8 8 A.2 B.1 6
9 9 A.2 B.1 11
10 10 A.2 B.1 11
# ℹ 20 more rowsWir haben auch noch die Möglichkeit die Faktoren mit der Funktion genFactor() zu modifizieren, aber dann können wir wieder nicht mit den Leveln rechnen. Dann lohnt sich die Sache eigentlich nicht. Für mich hat die expand_grid() Variante mehr Charme. Die Variante ist einfach und schnell.
Zero-inflated Poisson
Wenn wir mit der Poissonverteilung arbeiten, dann haben wir manchmal den Fall vorliegen, dass wir eine zero-inflated Poissonverteilung (deu. Null-inflationiert, nicht gebräuchlich) vorliegen haben. Wir haben also noch mehr Nullen in dem Messwert als wir in einer normalen Poissonverteilung erwarten würden. Wir können eine solche Poissonverteilung dann ebenfalls mit {simstudy} bauen. Wir nutzen dazu das Tutorium Adding a “mixture” distribution to the {simstudy} package, welches und gut hilft einmal aus zwei Verteilungen eine Verteilung mit sehr vielen Nullen zu bauen.
Was heißt jetzt zwei Verteilungen? Wir bauen uns erstmal eine Poissonverteilung, die faktisch nur aus Nullen besteht. Die Poissonverteilung hat den Mittelwert von \(\lambda\) gleich \(0\). Wir nennen diese Verteilung dann y_0. Dann bauen wir uns noch eine klassische Poissonverteilung mit einem \(\lambda\) von \(2\) zusammen. Das können wir erstmal machen. Weiter unten zeige ich dir dann auch noch wie du dir einen Effekt mit einem einfaktoriellen Design als Beispiel baust.
R Code [zeigen / verbergen]
zeroinf_def <- defData(varname = "y_0", formula = 0, dist = "nonrandom") |>
defData(varname = "y", formula = 2, dist = "poisson")Jetzt kommt der Teil wo wir die beiden Verteilungen miteinander mischen. Wir nehmen \(20%\) der Werte der y_0 Verteilung und wir nehmen \(80%\) der Werte der y Verteilung. Das machen wir etwas umständlich über den Strich |. Wir sagen hier welche Anteile der beiden Verteilungen wir dann wollen. Am Ende benennen wir noch den Messwert neu.
R Code [zeigen / verbergen]
zeroinf_def <- defData(zeroinf_def, varname = "y_zeroinf", formula = "y_0 | .2 + y | .8",
dist = "mixture")Dann können wir schon eintausend Beobachtungen erstellen.
R Code [zeigen / verbergen]
zeroinf_dt <- genData(1000, zeroinf_def)In der folgenden Abbidlung siesht du dann einmal die Histogramme der normalen Poissonverteilung sowie der zero-inflated Poissonverteilung. Wir haben hier als Unterschied eben den klaren Exzess an Nullen in der inflationierten Poissonverteilung. Das sieht man nicht unbedingt so gut, aber wir haben eben doch einiges mehr an Nullen als es nach der klassischen Verteilung sein sollte.
Wenn wir das ganze jetzt mit einem Behandlungsfaktor generieren wollen, dann müssen wir uns erstmal das Datenraster bauen. Wir nutzen dann das Raster um uns die Daten zu generieren und zu wissen wie viele Level mit Wiederholungen wir vorliegen haben.
R Code [zeigen / verbergen]
fac1_dt <- expand_grid(f_a = 1:3,
rep = 1:1000) |>
as.data.table()Zu unser Poissonverteilung mit den Mittelwert von Null kommt dann noch die klassische Poissonverteiung mit dem Effekt des Faktors. Hier hat jedes Faktorlevel einen Effekt von \(+4\) und wir nehmen einen Intercept von Null an. Dann mische ich noch die Verteilungen im Verhältnis von 1:3, daher 25% kommen aus der Poissonverteilung y_0 und 75% aus der Poissonverteilung mit Effekt. Damit haben wir dann die Datendefinition zusammen.
R Code [zeigen / verbergen]
zeroinf_def <- defDataAdd(varname = "y_0", formula = 0, dist = "nonrandom")
zeroinf_def <- defDataAdd(zeroinf_def, varname = "y",
formula = "0 + 4*f_a", dist = "poisson")
zeroinf_def <- defDataAdd(zeroinf_def, varname = "y_zeroinf",
formula = "y_0 | .25 + y | .75", dist = "mixture")Jetzt bringen wir unser Datenraster mit unserer Datendefinition zusammen. Wir erhalten dann einen neuen Datensatz mit unserem poissonverteilten Messwert. Das ist etwas anders als eben, aber hier müssen wir ja auch zwei Dinge zusammenbringen. Eben haben wir nur die Daten aus einer Datendefinition generiert. Am Ende möchte ich dann noch die Spalte f_a als Faktor mutieren und die Level umbenennen. Dann sind wir auch schon fertig.
R Code [zeigen / verbergen]
fac1_zeroinf_dt <- addColumns(zeroinf_def, fac1_dt) |>
mutate(f_a = factor(f_a, labels = str_c("A.", 1:3))) Dann schauen wir uns einmal die beiden Verteilungen im Vergleich in der folgenden Abbildung einmal an. Ich habe hier einmal den Violinplot gewählt, da kannst du dann besser den Fuß in der zero-inflated Poissonverteilung erkennen. Wir sehen dann aber auch, dass der Mittelwert nach unten gezogen wird. Auch der Median, als grau dargestellt, wird nach unten gezogen und entspricht dann nicht den vorgegeben Werten. Durch die Zeroinflation haben wir dann einen nicht mehr ganz sauberen Bezug zwischen den voreingestellten und den sich ergebenden Effekten.
Kovariates Design
Im Folgenden nutze ich das einkovariate und zweikovariate Design nochmal um etwas mit dem R Paket {simstudy} zu üben und die Funktionen zu zeigen. Am Ende nutzen wir eher selten ein kovariates Design in den Agrarwissenschaften, wenn es um ein poissonverteilten Messwert geht, aber zur Vollständigkeit gehört auch dieser Abschnitt mit dazu. Wir können hier verschiedene Modelle rechnen, ich nutze hier gleich die Gaussian Regression um auf meine Effekte zu kommen. Dann habe ich auch gleich die voreingestellten Mittelwerte wiedergegeben. Ein poissonverteilten Messwert analysieren wir ja eigentlich mit einer Poissonregression, aber dort erhalten wir dann als Effekte eben nicht die Steigung so direkt wieder sondern eben dann das Risk Ratio. Mehr dazu dann wie immer im entsprechenden Kapitel zu der Poissonregression, wenn dich mehr zum Thema interessiert.
Einkovariat \(c_1\)
Das Ziel der einkovariaten Datengenierung ist einfach. Wir haben eine normalverteilte Kovariate vorliegen und wollen einen Effekt für die Kovariate im poissonverteilten Messwert generieren. Bei einer Kovariate ist dann der Effekt auch gleichbedeutend mit der Steigung einer Graden durch die Punktewolke im Scatterplot. Ich nutze hier jetzt einmal sehr viel mehr Beobachtungen um dann auch auf die voreingestellten Effekte im Modell rauszukommen. Als erstes baue ich mir eine normalverteilte Kovariate mit einem Mittelwert von \(7\) und einer Streuung von \(3\) zusammen. Dann kann ich mir meinen Messwert in der folgenden Definition zusammenbauen. Hier habe ich dann einen Intercept von \(10\) und einen Effekt von \(2\) als Steigung vorliegen.
R Code [zeigen / verbergen]
cov1_pois_def <- defData(varname = "c_1", dist = "normal", formula = "6",
variance = 3) |>
defData(varname = "y", dist = "poisson", formula = "10 + 2 * c_1")Dann kann ich mir auch schon die eintausend Beobachtungen einmal generieren. Da ich hier keinn Faktoren vorliegen habe, brauche ich auch kein Datenraster für die Erstellung der Daten. Wir modellieren hier nicht in der gleichen Weise mit Effektschätzern unsere Daten wie wir dann in einer Poissonregression zurückkriegen würden. Das ist aber ein Preis, den wir aus Gründen der Einfachheit bezahlen wollen.
R Code [zeigen / verbergen]
cov1_pois_tbl <- genData(1000, cov1_pois_def)Dann können wir uns auch schon das Ergebnis der Datengenerierung in der folgenden Abbildung einmal anschauen. Hier wird dann auch sehr schön deutlich, dass wir durch den poissonverteilten Messwert Banden in dem Scatterplot sehen. Wir haben ja hier nur ganzzahlige Werte für die Messwert vorliegen. Bei einer so hohen Fallzahl an Beobachtungen erhalten wir dann auch unseren voreingestellten Effekte wieder. Hier müssen wir dann ein wenig aufpassen, wie wir denn die Daten analysieren wollen. Eine Poissonregression mit einem Risikoverhältnis als Effekt ist nicht das Gleiche wie eine Gaussianregression.
Wir können dann auch nochmal ein GLM rechnen und sehen, dass wir bei einer so hohen Fallzahl dann auch in einem GLM die passenden Effekte ungefähr herausbekommen. Ich nutze hier die Funktion parameters() aus dem gleichnamigen R Paket damit ich die Schätzer noch wieder zurücktransformieren kann. Auch hier findest du dann mehr in dem Kapitel zu der Poissonregression. Ich fitte hier erstmal die passende Poissonregression.
R Code [zeigen / verbergen]
pois_c1_fit <- glm(y ~ c_1, data = cov1_pois_tbl, family = poisson)Danns schauen wir uns einmal die Koeffizienten der Poissonregression an. Wir sind hier hauptsächlich am Inzidenzratenverhältnis (eng Incidence Rate Ratio, abk. IRR) interessiert.
R Code [zeigen / verbergen]
pois_c1_fit |>
parameters::model_parameters(exponentiate = TRUE)Parameter | IRR | SE | 95% CI | z | p
-----------------------------------------------------------------
(Intercept) | 12.96 | 0.33 | [12.34, 13.61] | 102.06 | < .001
c 1 | 1.09 | 4.21e-03 | [ 1.08, 1.10] | 22.46 | < .001Was sehen wir hier als Effekte? Wir rechnen hier das Inzidenzratenverhältnis aus. Das Inzidenzratenverhältnis beschreibt die multiplikative Änderung der erwarteten Rate, wenn sich die Kovariate um eine Einheit ändert. Das heißt hier in unserem Fall, dass wenn wir die Kovariate \(c_1\) um eine Einheit erhöhen, dass unser Messwert um den Faktor \(1.09\) ansteigt. Leider siehst du auch, dass wir es hier nicht mit einer linearen Modellierung zu tun haben. Daher kann ich dir das Inzidenzratenverhältnis einmal zwischen zwei Punkten mit \(c_1 = 6\) und \(c_1 = 7\) berechnen. Das Problem ist hier wirklich, dass wir es mit einem Faktor zu tun haben. Mehr dazu nochmal weiter unten im Kasten zu den Effekten in der Poissonregression und die Vingette Targeted logistic model coefficients geht das Problem in {simstudy} nochmal an.
R Code [zeigen / verbergen]
pois_c1_fit |>
predict(newdata = tibble(c_1 = c(6, 7)), type = "response") 1 2
21.80644 23.78146 Jetzt können wir auch den Faktor zwischen den beiden Messwerten berechnen. Wie ändert sich also Multiplikativ der Messwert von \(c_1 = 6\) zu \(c_1 = 7\)? Dafür teilen wir dann die beiden Messwerte durcheinander und erhalten dann faktisch unser Inzidenzratenverhältnis aus dem obigen Modell.
R Code [zeigen / verbergen]
23.86757 / 21.72900[1] 1.09842Jetzt können wir uns das Leben auch etwas einfacher machen und einmal die Marginal effect models mit dem R Paket {marginaleffects} nutzen um uns den Faktor zwischen den beiden Kovariatenwerten berechnen zu lassen. Das ist manchmal etwas schneller und auch einfacher durchzuführen. Darüber hinaus können wir eben dann nicht nur zwei Werte eingeben, sondern haben auch noch andere Möglichkeiten.
R Code [zeigen / verbergen]
avg_comparisons(pois_c1_fit, comparison = "ratio",
variables = list("c_1" = c(6, 7)))
Estimate Std. Error z Pr(>|z|) S 2.5 % 97.5 %
1.09 0.00421 259 <0.001 Inf 1.08 1.1
Term: c_1
Type: response
Comparison: mean(7) / mean(6)Mehr zu den Möglichkeiten dann auch in der Vingette Change in numeric predictors auf der Hilfeseite dem R Paket {marginaleffects}. Bei denen heißt dann die numerische Einflussvaraiable dann numeric predictor. Je nachdem wo man dann weiterliest ändern sich dann leider auch immer wieder die Begrifflichkeiten.
In der Poissonregression kriegen wir als Effektschätzer das Risk Ratio wiedergegeben. Das ist also nicht ein absoluter Wert, wie die Steigung, sondern ein Faktor um den sich die Messwerte unterschieden. Wenn wir die Einheit der Kovariate von 0 auf 1 erhöhen, dann würden wir vom Messwert von 10 auf 12 springen. Das würde einen Faktor von \(12/10 = 1.2\) ausmachen. Wenn wir jetzt aber unsere Kovariate von 1 auf 2 erhöhen, dann haben wir einen Messwert von 12 auf 14. Jetzt haben wir aber einen Faktor von \(14/12 = 1.17\) vorliegen. Der Faktor sinkt mit dem Anstieg der Kovariate. Da der absolute Anstieg immer weniger Anteil am Messwert ausmacht. Ich zeige dir den Zusammenhang einmal in der folgenden Abbildung. Mit jedem Sprung wird der Faktor kleiner. Am Ende ist dann das Risk Ratio aus einer Poissonregression mehr oder minder das Mittel über alle Sprünge. Wirklich linear ist dann der Zusammenhang nur auf der Linkskala. Hier hilft dann die Lektüre von Dormann (2013) oder aber das Kapitel zu der Poissonregression weiter. Die Vingette Targeted logistic model coefficients geht das Problem in {simstudy} nochmal an.
Zweikovariat unabhängig \(c_1 + c_2\)
Wir können natürlich auch mehrere Kovariaten vorliegen haben. Ich zeige hier dann einmal zwei Kovariaten, die einmal einer Normalverteilung und einmal einer Poissonverteilung entstammen. Wir müssen ja nicht nur normalverteilte Kovariaten vorliegen haben. Dann baue ich mir noch meinen poissonverteilten Messwert zusammen.
R Code [zeigen / verbergen]
cov2_pois_def <- defData(varname = "c_1", dist = "normal", formula = "10",
variance = 2) |>
defData(varname = "c_2", dist = "poisson", formula = "5") |>
defData(varname = "y", dist = "poisson", formula = "8 + 2 * c_1 + 1 * c_2")Dann generiere ich mir auch schon meine zehntausend Beobachtungen. Ich werde das Ergebnis der Generierung nicht visualiseren, daher können wir hier auch einmal mit mehr Beobachtungen arbeiten und haben dann auch etwas genauere Schätzer der Effekte in den Modellen.
R Code [zeigen / verbergen]
cov2_pois_tbl <- genData(10000, cov2_pois_def)Hier einmal das lineare Modell, dort sehen wir dann ob wir auch unsere eingestellten Effekte direkt wiedererhalten. Das ist natürlich jetzt nur so semi hilfreich, da wir ja auch schauen müssen wie denn die Werte in einem GLM aussehen würden.
R Code [zeigen / verbergen]
lm(y ~ c_1 + c_2, data = cov2_pois_tbl) |>
coef() |> round(2)(Intercept) c_1 c_2
7.58 2.02 1.04 Dafür passen wir einmal eine Poissonregression an und nutzen gleich einmal das Modell um uns die Inzidenzratenverhältnisse (eng Incidence Rate Ratio, abk. IRR) für die beiden Kovariaten berechnen zu lassen.
R Code [zeigen / verbergen]
pois_c2_fit <- glm(y ~ c_1 + c_2, data = cov2_pois_tbl, family = poisson)Wir können hier uns entweder die Koeffizienten einmal direkt über die Funktion model_parameters() wiedergeben lassen oder aber dann gleich einmal in der Funktionalität des R Pakets {marginaleffects}. Wir sehen hier, dass wir jeweils einen positiven Effekt haben. Mit steigenden Kovariaten erhöhen sich auch die Messwerte.
R Code [zeigen / verbergen]
pois_c2_fit |>
parameters::model_parameters(exponentiate = TRUE)Parameter | IRR | SE | 95% CI | z | p
-----------------------------------------------------------------
(Intercept) | 15.24 | 0.20 | [14.85, 15.64] | 206.38 | < .001
c 1 | 1.06 | 1.31e-03 | [ 1.06, 1.07] | 49.55 | < .001
c 2 | 1.03 | 7.93e-04 | [ 1.03, 1.03] | 40.19 | < .001Dam Ende dann nochmal die multiplikative Änderung als Inzidenzratenverhältnis für die beiden Kovariaten an zwei definierten Werten für die jeweiligen Kovariaten. Wir sehen dann auch hier das es dann eben auch auf die Werte ankommt, die wir uns anssehen wollen. Wir können eben nicht so einfach die Steigung wieder als Risk Ratio sehen. Aber das ist eben dann auch der Unterschied zwischen einem absoluten Effekt wie der Steigung und dem Risk Ratio also Faktor. Damit werden wir leben müssen. Die Vingette Targeted logistic model coefficients geht das Problem in {simstudy} nochmal an.
R Code [zeigen / verbergen]
avg_comparisons(pois_c2_fit, comparison = "ratio",
variables = list("c_1" = c(10, 11),
"c_2" = c(5, 6)))
Term Contrast Estimate Std. Error z Pr(>|z|) S 2.5 % 97.5 %
c_1 mean(11) / mean(10) 1.06 0.001310 811 <0.001 Inf 1.06 1.07
c_2 mean(6) / mean(5) 1.03 0.000793 1301 <0.001 Inf 1.03 1.03
Type: responseMultikovariat abhängig \(c_1 + c_2 + c_3\)
Eine multivariate Poissonverteilung ist nicht einfach zu bauen. Jedenfalls um so viel schwerer, dass eine gerundete multivariate Normalverteilung mit Korrelationsstruktur viel einfacher zu erstellen ist. Die Funktionalität in {simstudy} erlaubt auch multivariate korrelierte Poissondaten zu erstellen. Die Vingette Correlated data: additional distributions hilft hierbei. Ich zeige hier einmal die Variante der Poissonverteilung mit drei Kovariaten und einer konstanten Korrelation zwischen den Kovariaten von 0.3 als compound symmetry (cs) Korrelationsmatrix.
R Code [zeigen / verbergen]
cov2_dependent_dt <- genCorGen(1000, nvars = 3, params1 = c(8, 10, 12), dist = "poisson",
rho = 0.3, corstr = "cs", wide = TRUE,
cnames = c("c_1", "c_2", "c_3")) Im Folgenden siehst du dann einmal die Datentabelle der eintausend Beobachtungen. Wir haben die ID Spalte und dann die drei Kovariaten gefolgt von dem Messwert. Die beobachtete Korrelation ist dann 0.3 zwischen den Kovariaten.
R Code [zeigen / verbergen]
cov2_dependent_dtKey: <id>
id c_1 c_2 c_3
<int> <num> <num> <num>
1: 1 15 8 20
2: 2 7 17 13
3: 3 9 13 16
4: 4 8 9 16
5: 5 8 12 17
---
996: 996 6 8 3
997: 997 4 8 6
998: 998 7 6 13
999: 999 6 8 11
1000: 1000 10 8 11In den rohen Daten ist die Korrelation faktisch nicht zu sehen. Jedenfalls kann ich sowas nicht erkennen. Dafür müssen wir uns dann einmal die Korrelation berechnen lassen. Wie wir dann im Folgenden einmal sehen, passt es sehr gut. Wir erhalten die Korrelationen wieder, die wir auch eingestellt haben.
R Code [zeigen / verbergen]
correlation(cov2_dependent_dt, select = c("c_1", "c_2", "c_3")) |>
summary(redundant = TRUE)# Correlation Matrix (pearson-method)
Parameter | c_1 | c_2 | c_3
---------------------------------------
c_1 | | 0.30*** | 0.29***
c_2 | 0.30*** | | 0.31***
c_3 | 0.29*** | 0.31*** |
p-value adjustment method: Holm (1979)Wir können jetzt mit den korrelierten Kovariaten uns nochmal neue Messwerte erstellen. Hierbei bitte bachten, dass multiple Modelle Probleme kriegen, wenn die Kovariaten zu stark miteinander korreliert sind. Dazu dann aber mehr in dem Kapitel zu der multiplen Regression.
21.5.3 Ordinalverteilt
Was sind ordinalverteilte Messwerte? Wir können diese Art von Messwerten einmal als Antwortmöglichkeiten bei einem Fragebogen oder aber bei der Bonitur beobachten. Bei Fragebögen haben wir dann meistens drei bis fünf Antwortkategorien, die dann eben auch geordnet sind. Alle Kategorien haben dabei den gleichen Abstand. Bei der Bonitur haben wir häufig eine fünf- bis neunstufige Likertskala vorliegen, wobei die Neun immer die stärkste Ausprägung beschreibt. Wenn du nur zwei Antwortmöglichkeiten vorliegen hast, dann bist du im binären Fall, das machen wir dann weiter unten nochmal. Das R Paket {simstudy} hat mit der Vingette Ordinal Categorical Data eine Hilfeseite, die dir einmal beschreibt, wie wir ordinale Messwerte erstellen. Neben dem R Paket gibt es auch noch andere Möglichkeiten. Wir haben in dem Blogeintrag A refined brute force method to inform simulation of ordinal response data auch nochmal eine andere Sicht der Dinge. Hier simulieren wir aus mittlren Noten udn einer Standardabweichung dann die entsprechenden Daten.
Am Ende kannst du dir vorstellen, dass wir als Messwert wiederum einen Faktor vorliegen haben und eigentlich keinen numerischen Messwert. Wir wollen hier aber einmal verstehen, wie sich so ordinale Messwerte zusammensetzen. In den Agrarwissenschaften wird gerne eine 9er Likert Skala verwendet. Wir haben also ein Merkmal wie Blattfäule oder Befall und benoten nun mit einem 9er Notensystem. Das Problem ist nun, dass wir bei neun Noten natürlich auch jeder Ausprägung eine Wahrscheinlichkeit zuordnen müssen. Da werden dann die einzlenen Wahrscheinlichketen sehr klein. Darüber hinaus ist dann vermutlich eine Verschiebung der Noten zwischen den Gruppen auch sehr klein. Alles nicht so einfach. Am Ende ist das Verständnis der Datengenierung auch immer sehr hilfreich bei der Auswertung im Kapitel zur Multinomialen / Ordinalen Regression.
Um jetzt irgendwas zu sehen, brauchen wir wirklich viele Fallzahlen. Wenn du später nur fünf Beobachtungen pro Gruppe hast, dann wird es sehr schwer die vorengestellten Gruppenwahrscheinlichkeiten wiederzufinden. Deshalb kann es sein, dass es beser ist, dir die Daten einfach per Hand selber in Excel zu bauen.
Faktorielles Design
Für die ordinalverteilten Daten nutzen wir jetzt hier hauptsächtlich das faktorielle Design. Wir nutzen hier dabei aber auch nur ein reduziertes Design zum Einstieg. Das hat den Grund, dass die Erstellung schon kompliziert genug ist und wir bei kleinen Fallzahlen später dann kaum noch was wieder erkennen. Daher wir stellen zwar Effekte ein, wollen aber nur so wenige Beobachtungen haben, dass wir die Effekte gar nicht wiedersehen. Dann können wir uns die Zahlen auch händisch in Excel zusammentippen. Gambarota & Altoè (2024) geht in seiner Arbeit Ordinal regression models made easy: A tutorial on parameter interpretation, data simulation and power analysis nochmal auf das faktorielle Design ein und zeigt auch, wie wir komplexere Modelle simulieren können.
Einfaktoriell \(f_A\) mit 2 Leveln
Wir beginnen mit dem einfaktoriellen Modell mit zwei Leveln. Wir nutzen wieder das simple Modell um einmal die Herausforderungen zu verstehen. Wir haben es nämlich bei einem ordinalen Messwert mit etwas komplizierteren Daten zu tun. Beginnen wir mit einem ordinalen Messwert mit fünf Kategorien. Wir haben hier also eine Frage vorliegen mit fünf Antwortmöglichkeiten. Die Antwortmöglichkeiten sind trifft gar nicht zu, trifft nicht zu, weder noch, trifft zu und trifft voll zu. Jetzt kommt der eigentlich Twist. Wir haben diese Antwortmöglichkeiten ja in unterschiedlichen Häufigkeiten vorliegen. Wir haben also 100 Personen befragt und diese 100 Personen haben jeweils eine der Antwortmöglichkeiten auf die Frage geantwortet. Damit erhalten wir dann folgende hypothetische Verteilung der Antworten.
R Code [zeigen / verbergen]
null_prob <- c("trifft gar nicht zu" = 0.1,
"trifft nicht zu" = 0.2,
"weder noch" = 0.4,
"trifft zu" = 0.2,
"trifft voll zu" = 0.1)Wir können hier nicht einfach einen Effekt aufaddieren um eien zweite Gruppe zu erschaffen. Wir können nicht die Wahrscheinlichkeiten einfach um \(+0.1\) erhöhen, dann hätten wir ja in der Summe \(5 \times 0.1 = +0.5\) erhöht. Wir brauchen aber eine Wahrschinlichkeit von Eins in Summe. Daher müssen wir von einigen Antwortmöglichkeiten etwas abziehen und bei anderen etwas aufaddieren. Die Änderungen müssen sich ja dann zu Null aufaddieren, wie du im folgenden Beispiel für eine alternative Gruppe siehst.
R Code [zeigen / verbergen]
alt_prob <- c("trifft gar nicht zu" = 0.1 - 0.05,
"trifft nicht zu" = 0.2 - 0.1,
"weder noch" = 0.4 - 0.1,
"trifft zu" = 0.2 + 0.2,
"trifft voll zu" = 0.1 + 0.05)Dann können wir einmal für die beiden Gruppen dann die fünf Antwortmöglichkeiten mit den jeweiligen Wahrscheinlichkeiten erschaffen. Beachte, dass wir die Namen der Wahrscheinlichkeitsvektoren nicht mitschleifen und dann in der Genierung verlieren. Daher müssen wir dann den Messwert nochmal wieder als Faktor mutieren. Für die Darstellung ist es dann aber mit einem numerischen Messwert einfacher.
R Code [zeigen / verbergen]
ord_fac1_tbl <- tibble("A.1" = sample(1:5, 100, replace = TRUE, prob = null_prob),
"A.2" = sample(1:5, 100, replace = TRUE, prob = alt_prob)) |>
gather(key = "f_a", value = "y") |>
mutate(y_fct = factor(y, labels = c("trifft gar nicht zu", "trifft nicht zu", "weder noch",
"trifft zu", "trifft voll zu")))
ord_fac1_tbl# A tibble: 200 × 3
f_a y y_fct
<chr> <int> <fct>
1 A.1 3 weder noch
2 A.1 3 weder noch
3 A.1 1 trifft gar nicht zu
4 A.1 4 trifft zu
5 A.1 3 weder noch
6 A.1 1 trifft gar nicht zu
7 A.1 3 weder noch
8 A.1 3 weder noch
9 A.1 2 trifft nicht zu
10 A.1 5 trifft voll zu
# ℹ 190 more rowsDann können wir uns einmal die Verteilungen der Antwortmöglichkeiten in den beiden Gruppen mit je einhundert Beobachtungen anschauen. Wie du siehst, sind die beiden Verteilungen ineinander verschoben. Dabei hat die zweite Gruppe mehr Antworten bei trifft zu und trifft voll zu. Daher muss die Anzahl der Antworten bei den negativen Antworten kleiner sein. Wie du dir vorstellen kannst, haben wir hier nicht so viel Raum um Veränderungen zu erreichen und dabei irgendiwe noch eine Verteilung zu sehen. Wenn sich alles auf einer Seite zusammenballt, dann haben wir auch schon fast keine Varianz mehr.
Jetzt ist es natürlich super nervig einen Vektor zu bauen, der die Verschiebungen der Antwortwahrscheinlichkeiten beschreibt. Insbesondere wenn wir viele Antwortmöglichkeiten oder Notenausprägungen haben. Dann wird es sehr schwierig und sehr kleinteilig. Daher ist eine Idee eine latente Variable zu generieren, die wir verschieben können und uns über einen Umweg dann die Wahrscheinlichkeiten für die Antwortmöglichkeiten wiedergibt. Das ist die Lösung in {simstudy}.
In der folgenden Abbildung habe ich dir einmal das Prinzip visualisiert. Unsere Antwortwahrscheinlichkeiten sind die Flächen unter der Kurve. Damit wir hier alles etwas besser sehen, habe ich mich auf vier Antwortmöglichkeiten beschränkt. Wir können jetzt die z-Werte auf der x-Achse verschieben und damit ändern sich auch die Flächen zwischen den z-Werten. Da die Flächen die Antwortwahrscheinlichkeiten sind, können wir hier einen Wert ändern und erhalten neue Wahrscheinlicheiten wieder. Dabei ist zu beachten, das die Verschiebung für alle Antworten in die gleiche Richtung und um den gleichen Effekt erfolgt.
Natürlich ist das etwas zu schön um wahr zu sein. Wir haben hier eine ziemlich strenge Annahme getroffen. Durch die Verschiebung durch ein festes \(z\) gleich \(+0.7\) haben wir die Annahme an ein proportionales Wahrscheinlichkeitsmodell getroffen. Wir verschieben eben alle Antwortmöglichkeiten gleichmäßig um den gleichen Wert nach rechts. Die Richtung und auch die Effektstärke ist für alle Antwortmöglichkeiten gleich.
- Proportionales Wahrscheinlichkeitsmodell
-
Was will und das Proportionales Wahrscheinlichkeitsmodell (eng. proportional odds model) sagen? Wenn wir die Wahrscheinlichkeiten der Antworten verschieben, dann verschieben wir in die gleiche Richtung und um den gleichen Wert. Alle Antwortwahrscheinlichkeiten verhalten sich gleich und dementsprechend proportional.
Wir können jetzt in {simstudy} beide Varianten durchführen. Wir können also einmal davon ausgehen, dass wir den gleichen Effekt in die gleiche Richtung für alle Antwortwahrscheinlichkeiten vorliegen haben. Wir rechnen dann mit der Proportional odds Annahme. Wir können aber auch den Fall vorliegen haben, dass wir die Antwortwahrscheinlichkeiten individuell verschieben. Dann haben wir die Annahme der Non-proportional odds getroffen. Ich zeige dir dann weiter unten einmal die Durchführung in {simstudy} und wie wir dann die zufälligen ordinalen Werte erhalten.
Wir wollen aber erstmal damit anfangen ein Nullmmodell zu bauen. Daran können wir dann nochmal besser die latente Variable \(z\) verstehen. Später ist es eigentlich nur eine kleine Anpassung zwischen proportional odds und non-proportional odds. Als erstes brauchen wir daher wieder einmal unser Klassenwahrscheinlichkeiten für unser Nullmodell oder eben wie wahrscheinlich tritt eine der Antwortkategorien auf. Später können wir dann noch einen Effekt zu dieses Nullmodell hinzufügen. Um die Sache etwas zu vereinfachen habe ich hier nur vier Antwortmöglichkeiten gewählt und die entsprechenden Wahrscheinlichkeiten festgelegt.
R Code [zeigen / verbergen]
null_prob <- c("trifft gar nicht zu" = 0.31,
"trifft nicht zu" = 0.29,
"trifft zu" = 0.2,
"trifft voll zu" = 0.2)Jetzt brauchen wir noch unsere latente Variable \(z\) um unseren Wahrscheinlichkeiten in feste Werte zu übersetzen. In der folgenden Abbildung siehst du dann einmal den Logit der Wahrscheinlichkeiten. Der Logit beschreibt die Wahrscheinlichkeiten als logarithmierte Chancen. Das klingt jetzt etwas wild, aber wir transformieren im Prinzip die Flächen so, dass wir dann die Wahrscheinlichkeiten der Antwortzugehörigkeit auch durch die x-Werte beschreiben können.
Was ist die Logitfunktion einmal mathematisch? In der folgenden Formel siehst du einmal den Zusammenhang. Der Logit einer Wahrscheinlichkeit \(Pr\) ist das logarithmierte Chancenverhältnis. Damit können wir Wahrscheinlichkeiten in feste Werte überstetzen, die wir dann durch Addition oder Subtraktion verschieben können. Auf der x-Achse können wir ja schieben, die Flächen ändern sich dann automatisch.
\[ \text{logit}(Pr) = log\left(\cfrac{Pr}{1-Pr}\right) \]
In dem Fall der ordinalen Messwerte ist es nochmal einen kleinen Schritt komplizierter, da wir immer die kummulierten Wahrscheinlichkeiten für die latente Variable brauchen. In den folgenden Tabs siehst du dann einmal den Weg um die drei Grenzwerte aus der obigen Abbildung zu berechnen. Wir haben vier Antwortmöglichkieten mit vier Wahrscheinlichkeiten, also brauchen wir dann drei Grenzwerte als latente Variable.
\[ log \left(\cfrac{Pr(A.1 \leq 1)}{Pr(A.1 > 1)} | A.1 \right) = log \left(\cfrac{Pr(0.31)}{Pr(1-0.31)}\right) = -0.80 \]
\[ log \left(\cfrac{Pr(A.1 \leq 2)}{Pr(A.1 > 2)} | A.1 \right) = log \left(\cfrac{Pr(0.31 + 0.29)}{Pr(1-0.31-0.29)}\right) = 0.4 \]
\[ log \left(\cfrac{Pr(A.1 \leq 3)}{Pr(A.1 > 3)} | A.1 \right) = log \left(\cfrac{Pr(0.31 + 0.29 + 0.20)}{Pr(1-0.31-0.29-0.20)} \right) = 1.38 \approx 1.4 \]
Natürlich gibt es auch hier eine Implementierung in R. Leider sind die Namen der Funktionen nicht so eingängig wie es sein könnte. Oder andersherum, die R Entwickler wollten keine Doppelungen. Manchmal machen aber Doppelungen Sinn, da wir dann die Namen im Kontext besser zuordnen können. Der Blogeintrag logit() and logistic() functions in R geht da nochnmal genauer drauf ein. Hier können wir dann einmal die Wahrscheinlichkeiten aufsummieren und dann die qlogis() Funktion als Logitfunktion nutzen.
R Code [zeigen / verbergen]
qlogis(cumsum(null_prob))[1:3] |> round(2)trifft gar nicht zu trifft nicht zu trifft zu
-0.80 0.41 1.39 Wir sehen, dass es passt. Die sich ergebenden Werte sind die gleichen Werte, die wir auch schon oben in der Abbildung auf der x-Achse haben. Es ist also etwas von hinten durch die Brust, aber am Ende haben wir verstanden, woher die Werte jeweils kommen.
Und wie kommen wir wieder zurück? Was ist also die Umkehrfunktion einer \(\text{logit}()\)-Funktion? Das ist die \(\text{logistic}()\)-Funktion oder aber eben die \(\text{invlogit}()\)-Funktion für inverted. In unserem Fall können wir hier die Funktion plogis() nutzen. Wir erhalten dann die aufsummierten Wahrscheinlichkeiten. Da wir nur die Wahrscheinlichkeiten zwischen den Grenzen erhalten, müssen wir noch die Null und die Eins ergänzen. Dann rechnen wir noch die Differenz aus.
R Code [zeigen / verbergen]
c(0, plogis(c(-0.8, 0.4, 1.4)), 1) |> diff() |> round(2)[1] 0.31 0.29 0.20 0.20Wie wir sehen, kommen dann die Antwortwahrscheinlichkeiten unseres Nullmodells wieder heraus. Das klappt ja super. Solange ich nicht die Funktion gefunden habe, die mir in R intern die Wahrscheinlichkeiten aus Logits ausrechnet, schreibe ich mir die Funktion hier einmal selber.
R Code [zeigen / verbergen]
logodds_to_prob <- \(logodds) {
c(0, plogis(logodds), 1) |> diff() |> round(2) |> unname()
}Und dann einmal testen, ob die Funktion auch funktioniert. Dafür können wir dann einfach den Vektor der Logitwerte in die Funktion stecken und uns die Wahrscheinlichkeiten wiedergeben lassen.
R Code [zeigen / verbergen]
logodds_to_prob(c(-0.8, 0.4, 1.4))[1] 0.31 0.29 0.20 0.20Wunderbar, dann können wir die Funktion hier auch weiter verwenden. Ich nutze gleich mal die Funktion um uns die Antwortwahrscheinlichkeiten nach der Addierung eines Effekts zu berechnen.
Jetzt können wir uns auch schon unsere Daten in {simstudy} erstellen. Wir rechnen dann mit der Proportional odds Annahme in dem ersten Tab. Wir können aber auch den Fall vorliegen haben, dass wir die Antwortwahrscheinlichkeiten individuell verschieben. Dann haben wir die Annahme der Non-proportional odds getroffen. Ich würde aus Gründen der Einfachheit immer die Proportional odds Annahme wählen. Hier siehst du dann gleich wie sich die latente Variable \(z\) als Logit dann über einen Effekt verschiebt.
Jetzt können wir uns die Daten bauen. Hier kommt ein wichtiges Achtung. Wir müssen immer mit einem \((-1)\cdot z\) in der Datengenierung rechnen. Da wir in unserem Beispiel die latente Variable um \(+0.7\) verschieben wollen, tragen wir in die Formel unten ein \(-0.7\) ein. Ja, das ist etwas nervig, hat aber mit dem Logit und dem Algorithmus dahinter zu tun. Wir leben damit und bauen uns entsprechend die Daten. Der Faktor ist hier einfaktoriell mit zwei Leveln. Das mache ich über 1:1. Wir spezifizieren hier das Verhältnis der beiden Gruppen.
R Code [zeigen / verbergen]
ord_fac1_def <- defData(varname = "f_a", formula = "1;1", dist = "trtAssign") |>
defData(varname = "z", formula = "-0.7*f_a", dist = "nonrandom")Dann bauen wir uns einmal ganz viele Beobachtungen. Später ziehe ich dann zufällig ein kleineres Set an Beobachtungen für unsere simulierten Daten. Bis jetzt haben wir aber nur die latente Avriable und die Gruppen definiert.
R Code [zeigen / verbergen]
ord_fac1_pre_dt <- genData(25000, ord_fac1_def) |>
mutate(f_a = factor(f_a, labels = c("A.1", "A.2")))Jetzt ergänzen wir noch die ordinalen Messwerte als Spalte antwort. Dafür müssen wir jetzt sagen, was die latente Variable mit adjVar ist. Dann noch die Antwortwahrscheinlichkeiten ergänzen und schon können wir uns die Daten erstellen. Ich erstetze dann noch die Level des ordinalen Messwert mit den Antwortkategorien.
R Code [zeigen / verbergen]
ord_fac1_dt <- genOrdCat(ord_fac1_pre_dt, adjVar = "z", null_prob, catVar = "antwort") |>
mutate(antwort = factor(antwort, labels = c("trifft gar nicht zu", "trifft nicht zu",
"trifft zu", "trifft voll zu")))Jetzt können wir uns für jede Gruppe jeweils fünf Beobachtungen ziehen. Auch hier machen so wenige Beobachtungen kaum Sinn. So ist aber die Ausgabe schön klein und du kannst dann später mehr Beobachtungen ziehen. Dafür dann einfach die 5 mit einer höhren Zahl versehen und mehr Beobachtungen generieren.
R Code [zeigen / verbergen]
ord_fac1_dt |>
group_by(f_a) |>
sample_n(5)# A tibble: 10 × 4
# Groups: f_a [2]
id f_a z antwort
<int> <fct> <dbl> <fct>
1 20108 A.1 0 trifft zu
2 20399 A.1 0 trifft gar nicht zu
3 17044 A.1 0 trifft nicht zu
4 16156 A.1 0 trifft gar nicht zu
5 22539 A.1 0 trifft gar nicht zu
6 6490 A.2 -0.7 trifft nicht zu
7 17412 A.2 -0.7 trifft gar nicht zu
8 19404 A.2 -0.7 trifft zu
9 12519 A.2 -0.7 trifft gar nicht zu
10 5976 A.2 -0.7 trifft nicht zu Dann schauen wir nochmal, ob wir dann auch die Werte für die Logits auf der x-Achse für das Nullmmodell im Level \(A.1\) wiederfinden. Der Weg ist etwas kompliziert, aber dafür funktioniert er gut. Da wir hier ein {data.table} Objekt vorliegen haben, können wir in den eckigen Klammern etwas mehr Voodoo betreiben. Wir kriegen jetzt die drei Grenzwerte für die vier Antwortmöglichkeiten.
R Code [zeigen / verbergen]
logOdds_A1 <- qlogis(cumsum(ord_fac1_dt[f_a == "A.1", prop.table(table(antwort))]))[1:3]
logOdds_A1 |> round(2)trifft gar nicht zu trifft nicht zu trifft zu
-0.79 0.40 1.40 Dann noch schauen, ob die Logits dann auch in den Wahrscheinlichkeiten stimmen. Also kriegen wir die Nullmodell wahrscheinlichkeiten wieder? Anscheinend klappt das auch gut. Die Wahrscheinlichkeiten weichen etwas ab, da wir ja auch hier mit generierten Daten arbeiten.
R Code [zeigen / verbergen]
logodds_to_prob(logOdds_A1)[1] 0.31 0.28 0.20 0.20Wir haben dann ja die einen Effekt von \(+0.7\) für die latente Variable \(z\) eingestellt und damit auch die Logits verschoben. Das kommt auch ungefähr hin. Auch hier kriegen wir dann die drei Grenzwerte für die vier Antwortmöglichkeiten wiedergegeben.
R Code [zeigen / verbergen]
logOdds_A2 <- qlogis(cumsum(ord_fac1_dt[f_a == "A.2", prop.table(table(antwort))]))[1:3]
logOdds_A2 |> round(2)trifft gar nicht zu trifft nicht zu trifft zu
-0.10 1.09 2.09 Jetzt wird es spannend, da wir uns jetzt einmal anschauen können welche Wahrscheinlichkeiten für die Antwortkategorien entstehen, wenn wir um \(z+0.7\) verschieben. Wir ändern ja nicht die Wahrscheinlichkeiten für die Antwortmöglichkeiten direkt sondern eben den Logit.
R Code [zeigen / verbergen]
logodds_to_prob(logOdds_A2)[1] 0.48 0.27 0.14 0.11Am Ende summiert sich alles auf Eins auf und wir haben eben eine Änderung in den Wahrscheinlichkeiten. Die Gruppen verschieben sich und wir haben dann eben auch andere Wahrscheinlichkeiten für die Antworten. Jetzt kannst du spielen, wie stark du den Effekt möchtest.
Jetzt können wir alles nochmal so bauen, dass wir keinen konstanten Shift von \(+0.7\) auf der Logitskala haben, sondern eben einen Shift für jede Antwortkategorie. Mehr dazu dann auch im Blogeintrag simstudy update: ordinal data generation that violates proportionality. Wir brauchen dann wieder unser einfaktorielles Design mit der latenten Variable. Wir geben hier dann keinen Shift an, da wir ja gleich individuelle Shifts in der Antwortkategorien haben wollen.
R Code [zeigen / verbergen]
ord_fac1_def <- defData(varname = "f_a", formula = "1;1", dist = "trtAssign") |>
defData(varname = "z", formula = "0", dist = "nonrandom")Dann einmal die Daten mit der latenten Variable genieren. Aktuell brauchen wir die latente Variable \(z\) nur als einen Platzhalter.
R Code [zeigen / verbergen]
ord_fac1_pre_dt <- genData(25000, ord_fac1_def) Dann legen wir die individuellen Shifts für die Antwortkategorien fest. Hier ist nochmal wichtig, dass der letzte Wert eine Null ist. Wir verschieben ja nur drei Grenzwerte für die vier Antwortkategorien. Die Funktionen brauchen aber einen entsprechend langen Vektor.
R Code [zeigen / verbergen]
shift_z <- c(-0.4, 0.0, -1.7, 0)Dann können wir uns auch schon die Daten zusammenbauen und nutzen die Option npAdj um unsere individuellen Verschiebungen in den \(z\)-Werten anzugeben.
R Code [zeigen / verbergen]
ord_fac1_nonprop_dt <- genOrdCat(ord_fac1_pre_dt, baseprobs = null_prob, adjVar = "z",
catVar = "antwort", npVar = "f_a", npAdj = shift_z) |>
mutate(antwort = factor(antwort, labels = c("trifft gar nicht zu", "trifft nicht zu",
"trifft zu", "trifft voll zu")),
f_a = factor(f_a, labels = c("A.1", "A.2")))Wir können dann wie schon in dem ersten Tab einmal unsere kleinere Stichprobe aus den Daten hier ziehen. Am Ende ist die Frage, ob wir sowas überhaupt brauchen, wenn wir uns Spieldaten für die Analyse erstellen wollen. Es ist sehr schwer auf dem Logit die Verschiebungen nachzuvollziehen. Daher würde ich es bei den Proportional odds belassen. Es ist aber eine nette Fingerübung für das Verständnis. Die Verschiebung um einen festen Wert in den Antwortkategorien ist auch eine starke Annahme.
Dann stellt sich noch die Frage, ob wir denn auch die voreingestellten Parameter wieder aus einer ordinalen Regression herausbekommen. Dafür nutzen wir die folgende Formel. Dabei ist wichtig, dass wir eben hier auch das Minus sehen, was wir oben bei der Erstellung der Daten beachten mussten. Der Effekt ist ja die Verschiebung um \(+0.7\) durch die latente Variable. Wir sehen dann aber hier den Schätzer \(\beta\) für den Effekt mit einem Minuszeichen versehen. So ist eben der Algorithmus und die Modellierung. Wir wollen hier in der Formel den Effekt für das zweite Level \(A.2\) bestimmen. Wir haben vier Antwortkategorien, also erhalten wir dann drei Grenzwerte \(i\) aus dem ordinalen Modell zurück.
\[ log \left(\cfrac{Pr(Antwort \leq i)}{Pr(Antwort > i)} | f_A \right) = \alpha_i - \beta*I(f_A=A.2) \ , \ i \in \{1, 2, 3\} \]
Wir nutzen jetzt gleich die Funktion clm() aus dem R Paket {ordinal} für die Analyse der Daten. In diesem Zusammenhang möchte ich nochmal auf die Arbeit von Gambarota & Altoè (2024) mit dem Titel Ordinal regression models made easy: A tutorial on parameter interpretation, data simulation and power analysis verweisen. Hier geht es ja erstmal nur kurz darum zu schauen, ob wir die voreingestellten Werte auch wiederfinden. Die ordinale Regression hat ja noch ihr eigenes Kapitel. Wir bauen uns also das Modell wie folgt.
R Code [zeigen / verbergen]
clmFit <- clm(antwort ~ f_a, data = ord_fac1_dt)Dann können wir uns auch schon die Zusammenfassung des Modells angeben lassen. Ich nutze hier nur die coef() Funktion, da ich nicht an p-Werten oder anderem interessiert bin. Dafür würde dann die summary() gebraucht. Da wir ja auf den vollen Daten rechnen, kommt hier auch was passendes raus. Aber auch hier haben wir dann eine leichte Abweichung, was eben dann auch bei generierten Daten vorkommen kann.
R Code [zeigen / verbergen]
clm_coef <- clmFit |> coef() |> round(2)
clm_coef trifft gar nicht zu|trifft nicht zu trifft nicht zu|trifft zu
-0.79 0.40
trifft zu|trifft voll zu f_aA.2
1.40 -0.69 Damit ist der z-Wert mit \((-1) \cdot -0.69 \approx +0.7\) gut getroffen. Die Antwortwahrscheinlichkeiten sind auf dem Logit eben um 0.7 verschoben. Dann erhalten wir noch die Grenzwerte wieder, die wir dann auch nochmal in die entsprechenden Wahrscheinlichkeiten der Antworten umrechnen können. Hier brauchen wir dann natürlich nur die ersten drei Werte der Koeffizienten auswählen.
R Code [zeigen / verbergen]
logodds_to_prob(clm_coef[1:3])[1] 0.31 0.29 0.20 0.20Passt doch alles super. Wir erhalten die voreingestellten Wahrscheinlichkeiten unseres Nullmodells wieder. Dann noch die Verschiebung durch den z-Wert auch korrekt wiedergegeben. Damit haben wir dann die Grundlagen gut verstanden. Wir können jetzt nochmal etwas komplexere Modelle angehen.
Einfaktoriell \(f_A\) mit >2 Leveln
Wenn du jetzt mehr Levle oder Gruppen in deinen Faktor simulieren willst, dann kannst du einfach in der Option formula mehr Gruppen durch das Semikolon ; getrennt erschaffen. Wenn du immer eine 1 schreibst, dann haben alle die gleiche Gruppengröße. Je mehr Faktoren du hast, desto kleiner sollte natürlich der Effekt der latenten Variable sein.
R Code [zeigen / verbergen]
ord_fac1_def <- defData(varname = "f_a", formula = "1;1;1", dist = "trtAssign") |>
defData(varname = "z", formula = "-0.7*f_a", dist = "nonrandom")Dann brauchen wir wieder die Daten einmal aus denen wir dann gleich die Stichprobe ziehen.
R Code [zeigen / verbergen]
ord_fac1_pre_dt <- genData(25000, ord_fac1_def)Im Folgenden musst du nur einmal darauf achten, dass du dann auch mehr Level hast und dann eben auch mehr Labels vergeben musst. Hier habe ich ja dann drei Level also muss ich auch drei Level vergeben. Ich übernehme hier einmal die Antwortwahrscheinlichkeiten null_prob aus der obigen Erstellung der Daten.
R Code [zeigen / verbergen]
ord_fac1_dt <- genOrdCat(ord_fac1_pre_dt, adjVar = "z", null_prob, catVar = "antwort") |>
mutate(antwort = factor(antwort, labels = c("trifft gar nicht zu", "trifft nicht zu",
"trifft zu", "trifft voll zu")),
f_a = factor(f_a, labels = c("A.1", "A.2", "A.3")))Dann können wir uns wieder die Stichprobe ziehen. Damit hier die Ausgabe nicht so groß ist, habe ich dann nur drei Beobachtungen pro Gruppe gezogen. Das macht natürlich nur begrenz Sinn. Wie du auch schon siehst, kommen dann kaum Antwortmöglichkeiten vor.
R Code [zeigen / verbergen]
ord_fac1_dt |>
group_by(f_a) |>
sample_n(3)# A tibble: 9 × 4
# Groups: f_a [3]
id f_a z antwort
<int> <fct> <dbl> <fct>
1 22520 A.1 -0.7 trifft nicht zu
2 11135 A.1 -0.7 trifft nicht zu
3 23279 A.1 -0.7 trifft nicht zu
4 16935 A.2 -1.4 trifft gar nicht zu
5 13301 A.2 -1.4 trifft gar nicht zu
6 16797 A.2 -1.4 trifft voll zu
7 18126 A.3 -2.1 trifft nicht zu
8 4947 A.3 -2.1 trifft gar nicht zu
9 15494 A.3 -2.1 trifft gar nicht zuSomit haben wir dann auch die Möglichkeit mehr Gruppen zu simulieren. Hier musst du dann immer schauen, dass der Effekt nicht zu groß wird. Sonst schiebst du den letzten Faktor zu weit nach rechts in der Verteilung und es bleibt keine Fläche mehr als Wahrscheinlichkeit übrig. Das ist immer so eien Sache bei den ordinalen Messwerten. Wir sind eben beschränkt in beide Seiten.
Eine weitere Möglichkeit sich ordinale Messdaten zu generieren ist die Binomialverteilung. Hier können wir auswählen, wie viele Ausprägungen wir in dem Messwert haben wollen. Hierbei ist aber anzumerken, dass wir die Varianz nicht steuern können. Wir legen mehr oder minder den Mittelwert fest und dann ergibt sich der Rest. Also was ist die mittlere Note und dann streuen die Werte mit einem fixen Wert um den Mittelwert. Wir bauen uns hier auch erstmal wieder unser Faktorenraster um es dann mit den binomiaen Daten zu ergänzen.
R Code [zeigen / verbergen]
fac1_dt <- expand_grid(f_a = 1:3,
rep = 1:1000) |>
as.data.table()Hier kommt dann die Besonderheit. Wir legen mit der Option variance die Anzahl an Ausprägungen in unserem Messwert fest. Wir wollen hier neuen Noten simulieren, also legen wir den Wert der Varianz auf acht, da wir mit der Null beginnen zu zählen. Wir haben dann einen Effekt von \(+0.2\) in dem Faktor vorliegen. Der Effekt muss zwischen \(0\) und \(1\) liegen oder andersherum, am Ende dürfen keien Werte größer als Eins in der formula entstehen.
R Code [zeigen / verbergen]
y_binom_def <- defDataAdd(varname = "bonitur", formula = "0 + 0.2 * f_a",
dist = "binomial", variance = 8, link = "identity")Dann ergänzen wir einmal den Faktorenraster um die Datendefinition und erhöhen einmal die Bonitur um einen Wert, damit wir dann die Noten auch von Null bis Neun vorliegen haben.
R Code [zeigen / verbergen]
binom_fac1_dt <- addColumns(y_binom_def, fac1_dt) |>
mutate(bonitur = bonitur + 1)Dann können wir uns die Daten auch einmal als Tabelle anschauen.
R Code [zeigen / verbergen]
binom_fac1_dt |>
tabyl(f_a, bonitur) f_a 1 2 3 4 5 6 7 8 9
1 178 339 271 144 59 8 0 1 0
2 15 85 204 306 213 130 39 7 1
3 0 2 36 127 236 268 217 95 19Wir sehen hier also ganz gut, wie sich die Noten um eine mittlere Note gruppieren und dann eben streuen. Wir können die Streuung nicht anpassen, so dass hier eventuell nicht das rauskommt, was wir möchten. Eine Alternative ist es aber allemal und daher hier einmal erwähnt.
Mehrfaktorielles Design
Ich lasse hier auch einmal das mehrfaktorielle Design offen. Zwar liegt mit Gambarota & Altoè (2024) und der Arbeit Ordinal regression models made easy: A tutorial on parameter interpretation, data simulation and power analysis R Code vor, den du nutzen kannst um auch komplexere ordinale Daten zu modellieren, aber ist der R Code nur über GitHub | {ordinalsim} zu erreichen. Das Laden ist aber etwas komplizierter und am Ende ist die Frage, ob wir das auch wollen. Du kannst dir gerne die Arbeit anschauen. Wir machen hier einmal für das Erste Schluss.
Kovariates Design
Machen wir es kurz, wir betrachten hier nicht ein kovariates Design mit einem ordinalen Messwert. Das ergibt für mich keinen wirklichen Sinn. Theoretisch könnten wir eine Kovariate messen und dann schauen, wie sich der ordinale Messwert ändert, aber das würde ich dann eher als Gaussian linear Regression modellieren. Wir nehmen also einfach die mittleren Noten. Das würde dann reichen. Hier jetzt noch für Kovariaten die ordinale Messwerte zu generieren ist zu wild.
21.5.4 Binomialverteilt
Kommen wir am Ende noch zu einem binären Messwert oder auch \(0/1\)-Daten. Binäre Messwerte sind Messwerte, die nur zwei Gruppen beinhalten. Daher ist die Pflanze gesund (\(y = 0\)) oder krank (\(y = 1\)). Wir haben also keine kontinuierlichen Messwerte vorliegen sondern eben nur zwei Kategorien. Wir nennen es dann auch gerne zwei Klassen und damit wäre auch schonmal erklärt, warum wir binäre Daten in der Klassifikation im Kapitel zum maschinellen Lernen nutzen. Gerne werden daher auch \(0/1\)-Daten generell als Daten für die Prädiktion benutzt. Das ist aber nicht generell der Fall. In den Humanwissenschaften haben wir es fast nur mit \(0/1\)-Messwerten für die gesunden und kranken Patienten zu tun und daher nutzen wir dort auch gerne die logistische Regression für die Analyse der Daten. In den Agrarwissenschaften ist der binäre Messwert eher selten, da wir wirklich eine Menge an Beobachtungen brauchen um sinnvoll mit einem binären Messwert rechnen zu können. Da wir bei solch einem Messwert auch gerne an Anteile denken können, fragen wir uns dann schnell, wie viel Prozent der Beobachtungen sind krank? Wenn wir da nicht in die höheren zweistelligen Fallzahlen kommen, dann ist ja eine Beobachtung gleich sehr viele Prozentpunkte “wert”. Das macht dann meistens wenig Sinn tiefer zu analysieren.
Die Erstellung von binären Messwerten ist recht mühselig. Zum einen liegt es daran, dass wir ja immer die Anteile an den Einsen vorgeben. Daher sind wir auch in dem Zahlenraum \([0;1]\) beschränkt. Die Effekt müssen dann entsprechend klein sein, wenn du viele Level im Faktor hast. Der Vorteil ist aber, dass ich keien Lust habe hundert oder mehr Beobachtungen pro Gruppe mit Null und Eins in Excel zu versehen. Das geht dann hier sehr viel schneller. Da die logistsiche Regression als Analysemethode so eine große Bedeutung in den Humanwissenschaften hat, ist {simstudy} sehr um diesen Messwert mit Beispielen gesegnet, wie ich im folgenden Kasten nochmal erwähne.
Die Folgenden beiden Tutorien helfen nochmal bei der Erstellung von binären Daten. Am Ende ist es immer etwas schwierig Daten mit Effekt in einem 0/1 Raum zu erschaffen. Hier mögest du dann noch mehr Inspiration finden.
- Generating binary data by specifying the relative risk, with simulations hier gehen wir nochmal auf das relative Risiko ein. Ein häufiger Effektschätzer in der Epidemiologie und den Humanwissenschaften.
- Targeted logistic model coefficients geht nochmal auf das Problem der transformierten Effekt auf der log-Skala ein. Wir können die Daten auf verschiedene Weise mit Effekten versehen und daher gibt es auch etwas mehr zu Lesen.
Beginnen wollen wir wieder mit einem einfaktoriellen Design mit drei Leveln. Im Prinzip können wir auch nur zwei Level nehmen oder mehr, aber wir haben hier schon eine Menge Text produziert, da lohnt isch dann etwas mehr Kürze. Danach schauen wir uns dann noch den Fall mit einer Kovariaten an, dort können wir dann noch etwas zum Logit und der logistischen Funktion lernen. Die mehrfaktoriellen Fälle lasse ich hier einmal außenvor, dass würde alles noch mehr verkomplizieren. Meistens ist es nämlich das Problem, dass wir nicht in dem Bereich von Null bis Eins in der Formula durch die Effekte bleiben. Dann haben wir auf einmal sehr viele fehlende Werte in unseren binären Messwert. Die Lösung wäre dann alles auf dem Logit zu modellieren, aber das hat dann den nachteil der einfacheren Interpretierbarkeit. Dazu dann aber geich mehr.
Einfaktoriell \(f_A\) mit >2 Leveln
Wir wollen uns hier zwei Wege anschauen uns binäre Messwerte zu erschaffen. Zum einen können wir die Binäreverteilung nutzen. Eigentlich ist die Binäreverteilung nur ein Spezialfall der Binomialverteilung, so dass wir uns dann auch noch gleich die Implementierung in der Binomialverteilung anschauen. Zuerst brauchen wir wieder unser Faktorengrid an dem wir dann die Datendefinition kleben können. Damit wir hier überhaupt etwas sehen, nutze ich wieder zehntausend Beobachtungen pro Faktorlevel.
R Code [zeigen / verbergen]
fac1_dt <- expand_grid(f_a = 1:3,
rep = 1:10000) |>
as.data.table()Dann können wir die Daten schon auf zwei Wegen zusammenbauen. Dabei würde ich hier einmal die Binäreverteilung vorziehen, denn diese Verteilung macht exakt das was wir wollen. Die Binomialeverteilung ist dann einfach nochmal hier, damit ich auch diese später mal wiederfinde.
Im Folgenden bauen wir uns einmal die binären Messwerte. Hierbei ist wichtig, dass wir uns auf dem Identitylink bewegen. Das heißt, wir definieren einen Intercept als Wahrscheinlichkeit eine Eins zu beobachten und dann den Effekt als Änderung der Wahrscheinlichkeit eine Eins zu beobachten. Daher ist alles was in der formula-Option steht der Anteil an Einsen in den entsprechenden Leveln des Faktors. Hier ist es dann noch wichtig, dass die formula-Option am Ende immer im Interval \([0;1]\) bleibt. Wenn du also drei Faktorlevel hast, die alle einen Effekt von \(+0.4\) haben sollen, dann eght es nicht. Denn dann wären wir auch mit einem intercept von Null bei dem dritten Level über Eins. Daher müssen die Effekte bei mehr Leveln auch immer kleiner werden. Wir können hier auch andere Linkfunktionen wählen, dazu dann aber mehr bei der Kovariatensimulation.
R Code [zeigen / verbergen]
y_binary_def <- defDataAdd(varname = "y", formula = "0.2 + 0.2*f_a",
dist = "binary", link = "identity")Dann bringen wir einmal das Faktorengrid mit der Datendefinition zusammen und generieren uns die entsprechenden Daten mit den binären Messwerten.
R Code [zeigen / verbergen]
binary_fac1_dt <- addColumns(y_binary_def, fac1_dt)Dann schauen wir uns einmal das Ergebnis als Tabelle an. Wir haben in dem ersten Level des Anteil an Einsen des Intercepts, nämlich \(0.2\) oder 20% plus den Effekt des ersten Levels mit \(+0.2\). Dann haben wir als Effekt ja auch \(+0.2\) also immer 20% mehr für jedes Faktorlevel. Bei der hohen Anzahl an Beobachtungen stimmt das auch so sehr gut.
R Code [zeigen / verbergen]
binary_fac1_dt |>
tabyl(f_a, y) |>
adorn_percentages("row") |>
round(2) f_a 0 1
1 0.6 0.4
2 0.4 0.6
3 0.2 0.8Dann könnten wir wieder unsere Stichprobe aus den riesigen Daten wie schon bei der ordinalverteilten Daten ziehen und damit weiterechnen. Ich würde hier aber immer mit Stichproben nahe Hundert für jede Gruppe rechnen wollen. Bei drei Leveln sollte ich also auch gut dreihundert Beobachtungen in meiner Studie haben. So große Stichproben sind dann in den Agrarwissenschaften eher selten.
Eine andere Möglichkeit einen binären Messwert zu erschaffen ist die Binomialverteilung mit einer Varianz von Eins. Damit haben wir dann den Spezialfall der Binärenverteilung vorliegen. Der Weg ist der Gleiche, daher hier nur einmal skizziert.
R Code [zeigen / verbergen]
y_binom_def <- defDataAdd(varname = "infected", formula = "0 + 0.2 * f_a",
dist = "binomial", variance = 1, link = "identity")Dann bringen wir das Faktorenraster mit der Datendefinition zusammen.
R Code [zeigen / verbergen]
binom_fac1_dt <- addColumns(y_binom_def, fac1_dt)Dann können wir uns die Daten auch einmal als Tabelle anschauen. Auch hier sehen wir dann schön, dass wir die Effekte wiederfinden, die wir eingestellt haben. Hier ist der Intercept dann mal \(0%\) und die Anzahl an Einsen in jedem Level entspricht dem kumulativen Effekt der Level von \(+0.2\) für jedes Level.
R Code [zeigen / verbergen]
binom_fac1_dt |>
tabyl(f_a, infected) |>
adorn_percentages("row") |>
round(2) f_a 0 1
1 0.79 0.21
2 0.59 0.41
3 0.40 0.60Hier sehen wir dann aber leichte Rundungsabweichungen durch die etwas geringe Anzahl an Beobachtungen. Wenn wir noch mehr Beobachtungen einstellen, dann wird auch die geringe Abweichung verschwinden.
Das war es jetzt auch schon für das faktorielle Design. Wenn du ein zweifaktorielles Design brauchst, dann wird es eben schon sehr viel schwieriger mit den kleinen Effekten damit du in dem Interval \([0;1]\) bleibst. Da mache ich dann immer eine Simulation mit der Normalverteilung und versuche mir dann einen Grenzwert so zu bauen, dass ich zwei Gruppen erhalte. Mehr dazu dann am Ende des Abschnitts nochmal in dem Kasten zu der Generierung von binären Messwerten mit der Normalverteilung.
Kovariates Design
Kommen wir nun zu dem kovariaten Design, wo wir dann nochmal etwas genauer auf die Erstellung der binären Daten eingehen können. Wenn unsere Einflussvariable numerisch ist, dann haben wir etwas mehr Möglichkeiten unsere Daten darzustellen und zu verstehen, was dort eigentlich passiert. Wir haben ja jetzt auf der x-Achse nicht nur Kategorien sondern eben eine Kovariate. Das macht dann alles etwas einfacher. Hier können wir dann auf zwei Arten nochmal nachvollziehen, wie wir die Daten bauen. Wir haben die Möglichkeit den identity-Link zu wählen. Dann erstellen wir alles mit untransformierten Wahrscheinlichkeiten. Wir sagen, dass wir durch die Kovariate als Effekt \(+0.05\) haben wollen und für jede Einheit der Kovariate stiegt die Wahrscheinlichkeit für ein Ereignis (\(y =1\)) um 5% an. Das ist super zu verstehen. Wir können aber auch die Effekte auf dem logit-Link angeben. Dann generieren wir nicht mehr so einfach zu verstehende Effekte. Mehr dazu dann im zweiten Tab.
Am einfachsten machen wir es uns, wenn wir direkt auf den Wahrscheinlichkeiten die Daten generieren. Daher nehmen wir hier einmal eine normalverteilte Kovariate an und wollen dann mit der Kovariate den binären Messwert generieren. Dabei haben wir einen Mittelwert von Null für die Kovariate angenommen. Der Effekt kann jetzt direkt als Wahrscheinlichkeit interpretiert werden. Daher haben wir als Intercept 50% Wahrscheinlichkeit eine Eins in dem Messwert zu beobachten. Je Einheit Kovariate steigt die Wahrscheinlichkeit dann um \(+0.15\) an.
R Code [zeigen / verbergen]
cov1_binary_def <- defData(varname = "c_1", dist = "normal", formula = "0",
variance = 1) |>
defData(varname = "y", formula = "0.5 + 0.15*c_1",
dist = "binary", link = "identity")Dann bauen wir uns einmal die Daten damit wir gleich einmal in der Abbildung schauen können, wie sich die Messwerte dann verteilen.
R Code [zeigen / verbergen]
cov1_binary_dt <- genData(1000, cov1_binary_def)In der folgenden Abbildung können wir uns einmal die Daten und die entsprechenden Effekte anschauen. Die Grade entspricht einer linearen Regression durch die Punktewolke. Wir sehen hier sehr schön, dass wir die voreingestellten Werte aus unserer Simulation wiederfinden. Dafür haben wir hier aber den Nachteil, dass die Grade über die Null und Eins hinausschießen kann. Jetzt können wir natürlich auch sagen, dass wir ja eigentlich eine logistische Regression rechnen würden. Das machen wir auch gleich mal.
Wir können uns auch einmal die Modellanpassung mit einer logistischen Regression anschauen. Dafür müsse wir dann erstmal die logistische Regression mit der Funktion glm() rechnen.
R Code [zeigen / verbergen]
log_fit <- glm(y ~ c_1, cov1_binary_dt, family = binomial)Jetzt können wir die Ergebnisse der Regression direkt einmal in die Punktewolke abbilden. Der große Vorteil ist, dass wir die Grade jetzt dank der logistsichen Transformation innerhalb der Modellierung der logistischen Regression jetzt zwischen Null und Eins bleibt. Dafür haben wir jetzt aber nicht direkt die logistischen Effekte modelliert. Deshalb bietet sich im anderen Tab noch die Generierung mit logistischen Effekten an.
Es ist natürlich etwas blöd, wenn wir die Daten auf der Basis einer linearen Regression abbilden und dann über die logistische Regression auswerten wollen. Da wäre es ja besser, wenn wir direkt auf der logistischen Transformation unseren Messwert generieren. Hier gibt es auch die Vingette Simulate data from a logistic regression model: How the intercept parameter affects the probability of the event, die etwas genauer auf das Problem eingeht. Am Ende wählen wir hier die Option link = "logit" um unsere Messwerte mit dem Effekt auf dem Logit zu generieren. Das macht die Interpretation um einiges schwerer bis unmöglich. Wir müssen hier dann also rumprobieren, bis alles passt.
R Code [zeigen / verbergen]
cov1_binary_def <- defData(varname = "c_1", dist = "normal", formula = "0",
variance = 3) |>
defData(varname = "y", formula = "0 + 1.5*c_1",
dist = "binary", link = "logit")Dann können wir auch einmal die Daten generieren und uns anschauen, wie sich die Messwerte ergeben. Ich generiere hier einmal richtig viele Beobachtungen, damit wir auch gleich was sehen und nicht zu viel Rauschen in den Daten haben.
R Code [zeigen / verbergen]
cov1_binary_dt <- genData(100000, cov1_binary_def)Der folgende Gedankengang ist eher etwas wild, aber er hilft vielleicht sich die Sachlage nochmal zu veranschaulichen. Rein statistisch gesehen überfahren wir hier einen statistischen Engel, aber so richtig. Mir hat es aber nochmal geholfen eine Idee dafür zu entwicklen, was diese logistische Transfomation sein soll und wie diese stattfindet. Wir können jetzt einmal die Kovariate \(c_1\) in zehn gleichmäßige Stücke oder Intervalle teilen. Für jedes dieser Intervalle berechnen wir dann den Anteil an Einsen und addieren einen kleinen Fehler drauf. Am Ende können wir dann die logistische Transformation rechnen.
R Code [zeigen / verbergen]
cov1_binary_logit_dt <- cov1_binary_dt |>
mutate(c_1_bin = cut(c_1, breaks = 10)) |>
group_by(c_1_bin) |>
summarise(mean_y = round(mean(y), 2) + 0.001) |>
mutate(mean_y = ifelse(mean_y > 1, 0.999, mean_y),
lower = as.numeric(sub("\\((.+),.*", "\\1", c_1_bin)),
upper = as.numeric( sub("[^,]*,([^]]*)\\]", "\\1", c_1_bin)),
mid = (lower + upper)/2,
logistic = plogis(mean_y))In der folgenden Abbildung siehst du einmal das Ergebnis. Wir haben hier die logistsiche Transformation von den Wahrscheinlichkeiten eine Eins zu beobachten einmal für das Beispiel einer Kovariate dargestellt. So ergibt sich dann auch die charakteristische S-Kurve der logistischen Regression.
Dann hier nochmal die Abbildung für die normale Regression mit der Annahme der Normalverteilung. In diesem Fall schießt die Gerade über das Intervall hinaus. Wir haben damit dann auch Werte kleiner als Null und größer als Eins vorhergesagt. Die Gradengleichung ist dann aber hier auf dem Identity-Link wie in dem vorherigen Tab.
Wir können uns auch einmal die Modellanpassung mit einer logistischen Regression anschauen. Dann erhalten wir auch die echte S-Kurve wiedergeben und nicht unsere obige Annäherung. Dafür müsse wir dann erstmal die logistische Regression mit der Funktion glm() rechnen.
R Code [zeigen / verbergen]
log_fit <- glm(y ~ c_1, cov1_binary_dt, family = binomial)Jetzt können wir die Ergebnisse der Regression direkt einmal in die Punktewolke abbilden. Der große Vorteil ist, dass wir die Grade jetzt dank der logistsichen Transformation innerhalb der Modellierung der logistischen Regression jetzt zwischen Null und Eins bleibt. Dafür haben wir jetzt aber nicht direkt die logistischen Effekte modelliert. Somit haben wir uns beides einmal angeschaut. Was das bessere ist, hängt immer von deiner Fragestellung ab.
Zum Abschluss dieses Abschnitts möchte ich nochmal einen kleinen Exkurs über die logistsiche Regression geben. Es ist vielleicht hilfreich sich nochmal mit der Thematik zu beschäftigen. Ich würde aber komplexere kovariate Modelle nicht mehr simulieren, dass wird sehr schnell sehr unübersichtlich. Wenn du die Modelle für die Erstellung neuer Algorithmen brauchst, dann sind wir hier auch nicht ganz richtig. Dafür musst du dann tiefer in {simstudy} einsteigen.
Wenn wir uns mir der ordinalen oder aber logistischen Regression beschäftigen dann kommen wir nicht um die logit() und logistic() Funktionen herum. Beide Funktionen haben als Idee die begrenzten Wahrscheinlichkeiten in dem Zahlenraum \([0;1]\) auf \([-Inf;+Inf]\) zu transformieren. Hier bietet sich dann eben die logit() und logistic() Funktionen dafür an. Wie immer gibt es eine Reihe an nützlichen Tutorien, die ich hier mal kurz zusammenstelle. Vieles von dem hier passt dann auch eher in das Kapitel zur logistischen Regression rein.
- Das Tutorium zur logit() and logistic() functions in R liefert nochmal den Zusammehang zwischen den beiden Transformationen und wie die Funktionen in R heißen.
- In dem Artikel Logistic Regression wird nochmal erklärt, wie die Grundlagen der logistischen Regression lauten. Sehr ausführlich gemacht.
- Manchmal möchte man dann doch die logistische Regression per Hand rechnen. Dann hilft das Tutorium zu Logistic regression (by hand)
- Noch etwas umfangreicher ist das Tutorium Logistic Regression Explained from Scratch (Visually, Mathematically and Programmatically).
Wir wollen uns jetzt nochmal die beiden Transformationen in den beiden folgenden Tabs nochmal hier anschauen. Damit hast du dann eine Idee, was die beiden Funktionen machen. Im Prinzip verwandelt die logit-Funktion Zahlen im begrenzten Wahrscheinlichkeitsraum \([0;1]\) in einen faktisch unendlichen Zahlenraum.
Der Logit einer Wahrscheinlichkeit \(Pr\) ist durch folgende Formel gegeben.
\[ \text{logit}(Pr) = log\left(\cfrac{Pr}{1-Pr}\right) \]
Wir können den Logit in R mit der Funktion plogis() bestimmen.
R Code [zeigen / verbergen]
qlogis(0.6) # logit[1] 0.4054651Manchmal ist das aber etwas wirr, deshalb hier nochmal die Funktion ausgeschrieben, wie wir die Funktion oben in der mathematischen Formel sehen.
R Code [zeigen / verbergen]
logit <- \(x) log(x / (1 - x))
logit(0.6)[1] 0.4054651In der folgenden Abbildung siehst du einmal die Transformation von Wahrscheinlichkeitswerten in den Logit.
Die logistsiche Funktion ist durch folgende Formel gegeben. Dabei handelt es sich um die Umkehrfunktion der Logit-Funktion.
\[ \text{logistic}(z) = \cfrac{1}{1 + e^{-z}} \]
Wir können den Logit in R mit der Funktion qlogis() bestimmen.
R Code [zeigen / verbergen]
plogis(0.4054651) # logistic[1] 0.6Manchmal ist das aber etwas wirr, deshalb hier nochmal die Funktion ausgeschrieben, wie wir die Funktion oben in der mathematischen Formel sehen.
R Code [zeigen / verbergen]
invlogit <- \(z) 1/(1 + exp(-z))
invlogit(0.4054651)[1] 0.6In der folgenden Abbildung siehst du einmal die Rücktransformation von Logitwerten in Wahrscheinlichkeitswerte.
Was bleibt dann noch am Ende übrig? Wir könnten uns das Leben auch einfacher machen und die binären Daten über kontinuierliche, normalverteilte Daten generieren. Das habe ich einmal in dem folgenden Kasten gemacht. Manchmal ist die Dichotomisierung eine gute Möglichkeit um binäre Daten zu erhalten.
Wir können natürlich auch die Dtane erstmal mit einer Normalverteilung erschaffen und uns dann einen wie auch immer gearteten Grenzwert ausdenken bei dem wir dann entscheiden, dass alle Werte unter dem Grenzwert dann Null sind und alle über dem Grenzwert zu Eins werden. Teilweise ist es vermutlich die bessere Idee, wenn du sehr viele Einflussvariablen vorliegen hast. Hier nutze ich dann die Funktion ifelse() um mir einen Grenzwert von 2 zu setzen.
R Code [zeigen / verbergen]
binom_norm_tbl <- tibble(c_1 = rnorm(100, 0, 3),
y = 0 + 1.2 * c_1 + rnorm(100, 0, 1),
y_bin = ifelse(y >= 2, 1, 0))Dann können wir uns auch einmal anschauen, wie sich unser binärer Messwert dann verteilt.
R Code [zeigen / verbergen]
binom_norm_tbl |>
tabyl(y_bin) y_bin n percent
0 68 0.68
1 32 0.32Besonders wenn du dann viele Einflussvariablen hast, macht es Sinn einmal mit Grenzwerten zu spielen, so dass du am Ende auch balancierte binäre Messwerte hast. Am Ende macht es ja keinen Sinn nur Messwerte zu produzieren die alle Eins oder alle Null sind.
21.6 Komplexere Messwerte
Am Ende gibt es natürlich noch komplexere statitische Modell, die wir dann mit künstlichen Daten versehen können. Das R Paket {simstudy} hat ja hier eine große Auswahl an Möglichkeiten. Wie immer gilt aber, warum willst du dir die Daten generieren? Die genierung hat ja an sich keinen Selbstzweck. Es macht Sinn sich künstliche Daten zu generieren, wenn wir die Algorithmen besser verstehen wollen. Wenn du dann aber so tief in der Thematik der Algorithmen und Methoden bist, dann übersteigt meist dein Interesse hier meine Fähigkeiten allgemeiner über das Thema zu schreiben. Deshalb hier nochmal eine lockere Linksammlung für die komplexeren Modelle.
Überlebenszeiten
Wenn es um Überlebenszeotdaten geht dann hilft die Vingette Survival Data vom R Paket {simstudy} weiter. Dort wird nochmal auf die speziellen Anforderungen einer Überlebenszeitanalyse (eng. time to event) eingegangen. Wir haben in einer Überlebenszeitanalyse eben auch einen Messwert vorliegen, der aus zwei Komponenten besteht. Wir brauchen einmal die Information, ob ein Ereignis eingetreten ist oder nicht. Damit haben wir einen binären Messwert. Darüber hinaus wollen wir noch wissen, wie lange es bis zum Ereignis gedauert hat. Das ist eher ein poissonverteilter Messwert. Es sind eben die Anzahl an Tagen oder allgemeiner die vergangene Zeit bis zum Ereignis. Das zusammen macht die Sachlage nicht einfacher. Aktuell nutze ich keine Überlebenszeitanalysen in meinen Vorlesungen und daher bleibt dieses Abschnitt auch erstmal offen.
Zeitreihen
Wenn wir noch weiter über Zeitreihen sprechen, dann können wir verschiedene Dinge meinen. Wir haben eine Kovariate als Zeit vorliegen oder aber wir messen an bestimmten Zeitpunkten. Ein Zeitpunkt ist vermutlich eher ein Faktor. Messen wir viele Zeitpunkte, dann könnte der Zeitpunkt auch wieder eine Kovariate sein. Auf jeden Fall haben die Zeitpunkte untereinander etwas miteinander zu tun. Oder anders gesprochen, die Zeitpunkte sind untereinander korreliert. Je weiter die Zeitpunkte auseinanderliegen, desto geringer ist die Korrelation. Die folgenden Vingetten des R Paketes {simstudy} beschäftigen sich mit diesen Fragen.
- Die Vingette zu den Longitudinal Data ist im Prinzip ein guter Einstieg, wenn du längere Zeitreihen vorliegen hast. Was lang ist, hängt wiederum von Betrachter ab. Aber es geht eher um mehrere Wochen bis Monate.
- Die Vingette Correlated Data beschreibt eher die korrelierten Zeitpunkte als Faktoren. Hier musst du aber mal schauen, ob nicht die Vingette zu Correlation Matrices besser geeignet ist.
- Wenn es komplizieter wird, dann können wir auch ordinale Messwerte mit der Vingette Correlated multivariate ordinal data erstellen. Hier wird es dann aber sehr komplex, wenn es dann noch um Effekte geht.
Am Ende sind alle Beispiele der Vingetten eher linear vom Verlauf über die Zeit. Wenn es wilder sein soll, also wir eher eine Kurve beobachten wollen, dann brauchen wir die folgende Vingette.
- Die Vingette Spline Data stellt im Prinzip nicht lineare Zusammenhänge dar. Wenn du also Daten brauchst, die Kurvenverläufe darstellen dann wird dir hier weitergeholfen. Es wird dann aber schnell sehr komplex, so dass du hier dann einiges an Zeit brauchen wirst um die Daten gut zu simulieren.